
|
Учебный сайт Алены Корягиной |

|
Главная |
Oбо мне |
Cеместры |
Cсылки |
Поиск сигналов. Теория
Поиск регуляторных мотивов транскрипции в бактериальных последовательностях
Сайты связывания транскрипционных факторов у прокариот достаточно длинны (~15 нуклеотидов), и часто более консервативны, чем их окружение. Также они часто имеют дополнительную внутреннюю структуру: например, являются почти строгими палиндромами. Эти свойства позволяют эффективно находить такие сайты de novo методами сравнительной геномики, например, при помощи программы MEME.
Для поиска регуляторного мотива в наборе промоторных областей генов E.coli (MEME06.txt) был использован список уже установленных экспериментально сайтов связывания белка PurR (MEMEexp.txt). Оба эти набора последовательностей были поданы на вход программе МЕМЕ.
Программа запускалась два раза с неизменяющимися параметрами: Minimum length = 16, Maximum length = 16, Maximum number of motifs to find = 1, но с
различным числом ожидаемых сайтов в последовательностях (параметр How do you think the occurrences of a single motif are distributed among the sequences?):
1. One per sequence
2. Zero or one per sequence.
Поиск 16 нуклеотидного мотива проводился по 15 промотерным последовательностям длинной в 100 нуклеотидов. Для пяти из них (folD, rpiA, carA, pdhR, fixA) нет данных об экспериментально установленных сайтах связывания транскрипционного фактора PurR. Для оставшихся десяти последовательностей предсказанные сайты совпали с сайтами, доказанными экспериментально (пересечение на 8 или более нуклеотидов). Для последовательности folD сайт был предсказан только при первом запуске программы МЕМЕ, а для последовательности rpiА сайт был предсказан при обоих запусках, но предсказанные сайты не совпали. Для оставшихся трех последовательностей сайты были предсказаны только при первом запуске, что не удивительно, так как программа запускалась с параметром, при котором она должна находить сайт в последовательности, но значение pValue для этих последовательностей больше 10-4, вследствие чего эти последовательности были проигнорированы. Также следует обратить внимание, что для последовательностей purM, codB, cvpA, purE и folD сайты были найдены на комплементарной цепи, поэтому поиск мотивов проводился на обратно-комплементарной цепи, то есть сайт был спроецирован на прямую цепь.
Результаты предсказания и экспериментальные данные о сайтах были отмечены на последовательностях и сохранены в файле sites.docx. В файле все сайты (и экспериментальные, и предсказанные) расположены на сером фоне, синим выделены экспериментально установленные сайты, курсивом выделены сайты, найденные с помощью программы MEME с параметром «One per sequence», жирным шрифтом – сайты, найденные с помощью программы MEME с параметром «Zero or one per sequence».
При анализе графического представления предсказанных и экспериментально доказанных сайтов можно определить следующую закономерность: программа МЕМЕ с параметром One per sequence предсказывает сайты на два нуклеотида ранее по сравнению с экспериментально доказанными сайтом, а с параметром Zero or one per sequence на один нуклеотид ранее или, наоборот позже. Полного совпадения предсказанного и экспериментального сайта не наблюдалось ни при дном параметре.
Описание возможностей сервера WebGeSTer
Дальнейшая работа выполнялась совместно с Швецовой Екатериной и Дюгай Ильей.
WebGeSTer – база данных и сервер для предсказаний ρ-независимых терминаторов прокариот.
Существует два альтернативных механизма терминации транскрипции прокариот: с участием ρ-фактора и без него. ρ-независимые терминаторы располагаются за 16-20 нуклеотидных пар от точки терминации и состоят из палиндромных последовательностей (последовательности, которые читаются одинаково слева направо и справа налево), которые содержат большое количество гуанина и цитозина. За этим участком может быть расположена олигоадениновая последовательность. Транскрипция в области палиндрома приводит к тому, что в получившемся РНК-транскрипте быстро образуется устойчивый элемент вторичной структуры – «шпилька», которая богата прочными G-C парами. «Шпилька» вызывает временную остановку полимеразы и прекращение транскрипции. В этот момент полимераза располагается на полиурациловом фрагменте последовательности (рис. 1). Вследствие того, что олигоуридиловый-олигоадениновой дуплекс является наименее прочным, он диссоциирует, мРНК освобождается от ДНК и РНК-полимеразы и транскрипция прекращается. Если в ДНК отсутствует полиадениновая последовательность, то во время паузы транскрипции диссоциации РНК не происходит и через некоторое время транскрипция возобновляется.
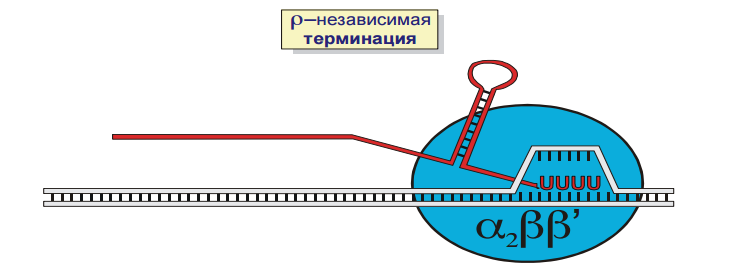
Рис. 1. Механизм ρ-независимой терминации прокариот.
Терминаторная «шпилька» имеет следующие свойства (рис. 2):
- длина стебля от 4 до 30 нуклеотидов
- длина петли от 3 до 9
- длина петли не более длины стебля + 2
- количество неспаренных нуклеотидов (мисмэтчей) не более 3
- длина полиуридинового фрагмента не менее 4
- содержание G/C более 50%
- низкое значение ΔG диссоциации РНК
- расположение вблизи открытой рамки считывания
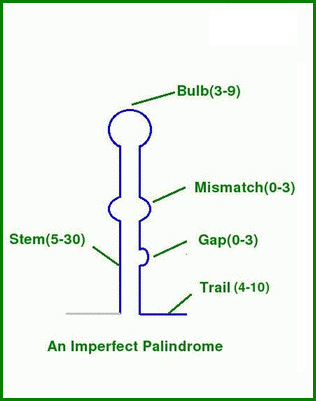
Рис. 2. Схема «шпильки». Изображение получено с помощью WebGeSTer.
Также «шпильки» могут быть различных типов (рис. 3): L, I, X, U, V. L-shaped, - палиндромные структуры с полиуридиловым хвостом, I-shaped - без полиуридилового хвоста, X-shaped - терминаторы конвергентных генов, находящихся на разных цепях, U-shaped - более одной шпильки в пределах 50 нуклеотидов downstream участка гена, V-shaped - когда следующая шпилечная структура начинается перед тем как закончится предыдущая.
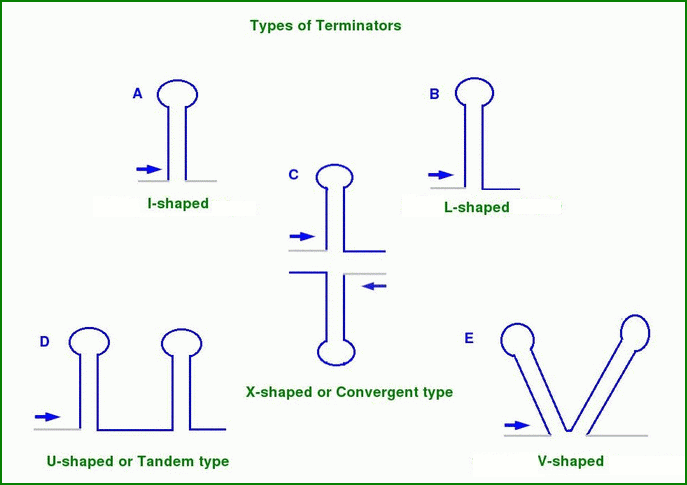
Рис. 3. Типы «шпилек». Изображение получено с помощью WebGeSTer.
Сервер WebGeSTer осуществляет поиск таких ρ-независимых терминаторов у прокариот. Алгоритм поиска состоит из четырех этапов:
- Начало поиска происходит одновременно с двух участков последовательности ДНК: первый фрагмент – G/C богатый фрагмент (≥50%), второй - обратнокомплементарный ему фрагмент в пределах следующих за первым фрагментом 70 нуклеотидов.
- Поиск мисмэтчей (некомплементарных пар) при движении поиска по направлению к другому фрагменту.
- Моделирование всех возможных структур «шпилек» с различными комбинациями гэпов и мисмэтчей. Расчет ΔG для каждой структуры по алгоритму Mfold. Выбор единственной структуры с наименьшим значением deltaG.
- Оценка характеристик полученной «шпильки»: длин стебля, петли, наличие и количество мисмэтчей и гэпов. Если значения характеристик не соответствуют установленным нормам, то поиск шпильки возобновляется.
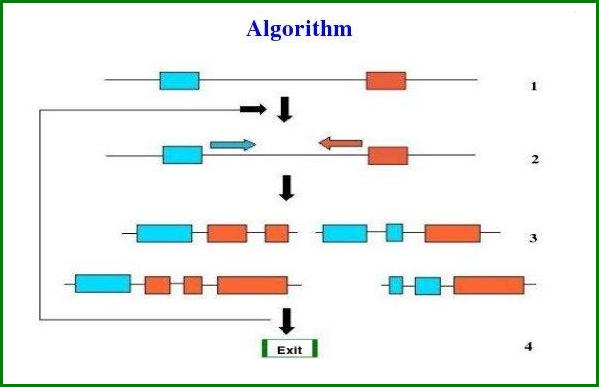
Рис. 4. Алгоритм поиска ρ-независимых терминаторов у прокариот, реализованный сервисом WebGeSTer. Изображение получено с помощью WebGeSTer.
WebGeSTer содержит как базу данных, содержащую предсказания терминаторных шпилек для большого набора прокариот, так и программу для поиска таких последовательностей в геноме любого другого прокариотического организма.
Чтобы проиллюстрировать возможности WebGeSTer мы выбрали архею Vulcanisaeta distributa DSM 14429 (длина генома 2 374 137 нуклеотидов), терминаторы которой не содержатся в базе данных WebGeSTer, и провели для неё предсказание ρ-независимых терминаторов.
Для этого нужно пройти по ссылке "Run WebGeSTer", ввести параметры поиска шпилек (например, максимальную/минимальную длину, максимальное количество мистэтчей и др.) и загрузить геном нужной бактерии в виде файла в формате *.gbk (предварительно переконвертированного в формат *.gbkf.gz, который понимает WebGeSTer) либо в виде файлов *.fna and *.ffn (предварительно сжатых). Мы запускали поиск не меняя параметров по умолчанию и использовали формат *.gbk. Получена страница с результатами работы программы. Информацию о количестве найденных шпилек разных типов можно увидеть в таблице 1.
Таблица 1. Информация о ρ-независимых терминаторах Vulcanisaeta distributa DSM 14429. Получена с помощью сервера WebGeSTer.
| Свойства | Прямая цепь | Обратная цепь | Всего |
| Гены | 1284 | 1258 | 2542 |
| Терминаторы | 204 | 178 | 382 |
| Терминаторы с лучшим deltaG | 163 | 148 | 311 |
| L-терминаторы | 58 | 47 | 105 |
| L-терминаторы с лучшим deltaG | 42 | 42 | 84 |
| I-терминаторы | 146 | 131 | 277 |
| I-терминаторы с лучшим deltaG | 121 | 106 | 227 |
| X-терминаторы | 7 | ||
| X-терминаторы с лучшим deltaG | 5 | ||
| U-терминаторы | 16 | 11 | 27 |
| V-терминаторы | 0 | 0 | 0 |
Также получен архив RawData.zip, содержащий подробную информацию о всех найденных шпильках (координаты, количество мисмэтчей и др.). Кроме того, если пройти по ссылке "Figures" на выходной странице, можно получить графические изображения каждой шпильки по отдельности. На рисунке 5 можно увидеть несколько примеров.

Рис. 5. Примеры ρ-независимых терминаторов археи Vulcanisaeta distributa DSM 14429. На изображении приведены шпильки L-shaped, X-shaped и U-shaped. Изображение получено с помощью WebGeSTer.
Также, пройдя по ссылке "Graphs" можно получить графики распределения терминаторов в downstream участке открытой рамки считывания (ORF). Пример такого графика можно увидеть на рисунке 6.
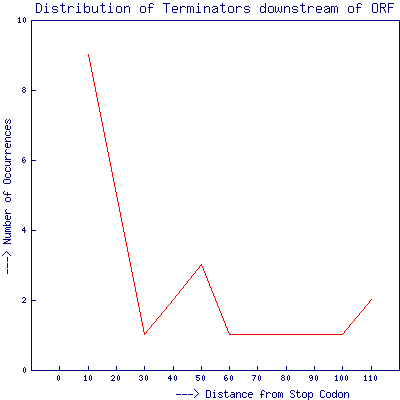
Рис. 6. График распределения L-shaped терминаторов прямой цепи (palinsreg) в downstream участке открытой рамки считывания (ORF) археи Vulcanisaeta distributa DSM 14429. "Точкой терминации" считается начало шпильки (Consider start of stem). По оси абсцисс отложено расстояние от стоп кодона ORF, по оси ординат - встречаемость терминаторов. Изображение получено с помощью WebGeSTer.
Дата последнего изменения: 17.04.15