|
 |
Учебный сайт Морозова Александра |
<< Назад к странице 4 семестра
Практикум 2Реконструкция филогенииЗадания1) С помощью таксономического сервиса NCBI определить, к каким таксонам относятся отобранные бактерии. Указать, какие нетривиальные ветви на дереве отобранных бактерий выделяют какие-нибудь из таксонов.
2) Из списка функций белков со страницы практикума выбрать одну 3) Получить из Swiss-Prot последовательности белков с данной функцией из отобранных бактерий и выровнять их. 4) Отредактировать названия последовательностей: оставить от названия каждого белка только мнемонику вида. 5) В программе MEGA (методом "Analyze") открыть файл с выравниванием и реконструировать филогению тремя различными методами. Сохранить полученные деревья в формате Newick. Результаты1. Таксономия
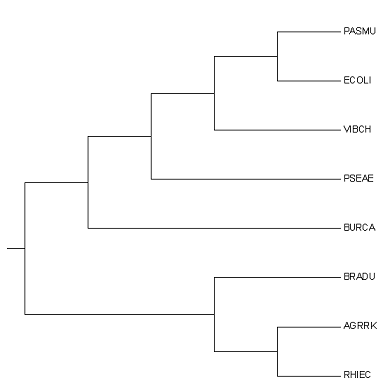
Из приведенной таблицы и рисунка филогенетического дерева видно, что : 1) нетривиальная ветвь {BRADU, AGRRK, RHIEC} выделяет таксон Rhizobiales; 2) нетривиальная ветвь {AGRRK, RHIEC} выделяет таксон Rhizobiaceae; 3) нетривиальная ветвь {PASMU, ECOLI, VIBCH, PSEAE} выделяет таксон Gammaproteobacteria. 2. Выбор функции белка.Выбранная функция: Фактор элонгации трансляции Ts (EFTS) 3-4. Выравнивание.1) Последовательности белков были получены путем использования формы загрузки из Uniprot со списком из идентификаторов белков вида "EFTS_ECOLI". FASTA-файл с последовательностями доступен по Ссылке 2) С помощью команды: muscle -in pr2_proteins.fasta -out pr2_alignment.fasta получен FASTA-файл с выравниванием данных последовательностей. Далее выравнивание было отредактировано в программе JalView. Файл с отредактированным выравниванием доступен по Ссылке 5. Построение филогенетических деревьев.1) В программе Mega файл с выравниванием открыт по схеме: File -> Open a file/session -> Analyze -> Protein Sequences -> OK. 2) Во вкладке Phylogeny перечислены доступные методы построения филогенетических деревьев. Я выбрал: Neighbor-Joining, Минимальной эволюции (minimum-evolution), Максимальной экономии (maximum parsimony). Все методы запускались с параметрами по умолчанию. 3) Полученные деревья сохранялись в формате Newick в файлах с расширением .tre. Файлы доступны для скачивания по ссылкам ниже. |
||||||||||||||||||||||||||||||||