|
 |
Учебный сайт Морозова Александра |
<< Назад к странице 4 семестра
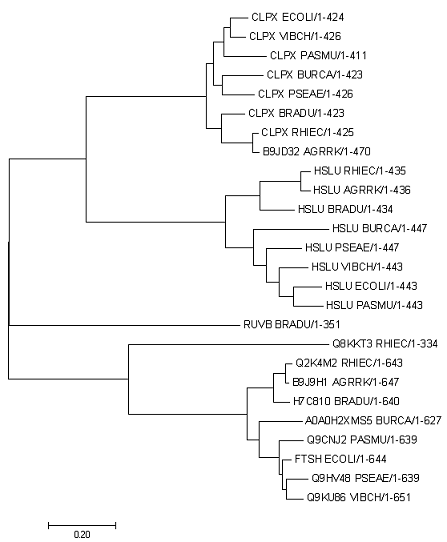
Практикум 3Укоренение и бутстрэпЗадания4. Построение и анализ дерева, содержащего паралоги. 1) Найти в отобранных бактериях достоверные гомологи белка CLPX_ECOLI; реконструировать дерево этих гомологов. 2) Указать несколько пар паралогов и две-три группы попарно ортологичных белков. 3) Привести примеры отражённых на дереве эволюционных событий двух типов: 1) дупликация гена; 2) разделение путей эволюции белков в результате видообразования. Результаты4. Построение и анализ дерева, содержащего паралоги.1) Файлы с протеомами отобранных бактерий были скопированы в собственную директорию и объединены в один файл командой: cat *.fasta >> bacteria.fasta 2*) Далее - немного о совершенной мной ошибке. Зайдя на страницу blastp, выбрав "Align two or more sequences", я загрузил файл с протеомами, затем - файл с исследуемым белком. Указав нужные параметры, запустил blast. И получил такую ошибку: Message ID#24 Error: Failed to read the Blast query: Nucleotide FASTA provided for protein sequence Пересмотрев презентацию с прошлого семестра, посвященную нуклеотидному blast, вспомнил, что сначала неободимо было создать БАЗУ ДАННЫХ из файла с протеомами, т.к. среди них будет осуществлятся поиск гомологов. В этом, собственно, и состояла моя ошибка. Поскольку БД можно создать командами из локального BLAST, установленного на kodomo, всю дальнейшую работу решил проводить на kodomo. Надеюсь, информация окажется полезной. 2) Создал базу данных BLASTP командой: makeblastdb -in bacteria.fasta -dbtype prot Запустил BLASTP командой: blastp -task blastp -query CLPX_ECOLI.fasta -db bacteria.fasta -evalue 0.001 -out blast_results.txt В итоге получил файл blast_results.txt, содержание которого аналогично тому, что я бы получил на странице с результатами, если бы использовал Интернет-версию BLASTP. Получилось 26 белков, из которых, ожидаемо, лучший E-value (0.0) и показатель Identity (100%) имеет консенсусный белок CLPX_ECOLI (т.к. ECOLI тоже входит в число отобранных мной бактерий, и данный белок представлен в протеоме). Также E-value 0.0 имеют белки CLPX из 6 других бактерий и белок B9JD32 из AGRRK, полное название которого совпадает с расшифровкой CLPX из других организмов. 3) Из файла с результатами записал в отдельный файл идентификаторы белков, после чего с помощью формы загрузки из Uniprot получил fasta-файл со всеми 26 последовательностями. Далее получил выравнивание данных последовательностей командой: muscle -in proteins.fasta -out pr3_alignment.fasta Выравнивание было откорректировано в программе JalView. Результат доступен по ссылке 4) Из полученного выравнивания в программе MEGA было построено филогенетическое дерево методом Neighbor-Joining. Файл с деревом в формате Newick (.tre) доступен по ссылке
Интересно, что в полученном дереве ортологичная группа HSLU полностью совпадает по построению дерева с деревом видов, в то время как дерево группы CLPX несколько от него отличается. а) Пары паралогов: 1) CLPX_ECOLI, HSLU_ECOLI 2) HSLU_PASMU, Q9CNJ2_PASMU 3) CLPX_BRADU, H7C81O_BRADU 4) Q8KKT3_RHIEC, Q2K4M2_RHIEC б) Группы попарно ортологичных белков: (белки CLPX + B9JD32), (белки HSLU) в) Дупликация гена: 1) Q8KKT3_RHIEC/1-334, Q2K4M2_RHIEC/1-643 2) FTSH_ECOLI/1-644, CLPX_ECOLI/1-424 г) Разделение путей эволюции белков в результате видообразования: 1)CLPX_ECOLI, CLPX_PASMU 2)Q8KKT3_RHIEC, B9J9H1_AGRRK |