Практикум 10
Программа BLAST
Автор старался, но не может гарантировать отсутствие биологических ошибок.
1. Поиск гомологов мальтоолигозилтрегалозо-синтазы
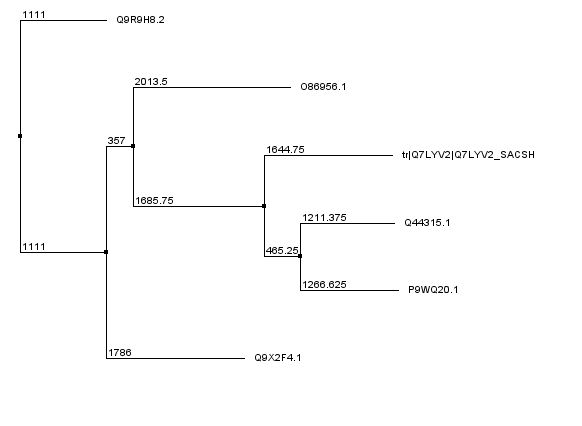
В практикуме 7 был выбран белок Maltooligosyl trehalose synthase (ID: Q7LYV2_SACSH). Для поиска гомологов мальтоолигозилтрегалозо-синтазы была запущена программа Protein BLAST, алгоритм поиска: blastp. В окошке "Enter query sequence" было выбрано подгрузить полученный из UniProtKB fasta-файл с последовательностью исследуемого белка. Банк последовательностей был выбран не Non-redundant protein sequences (nr), а UniProtKB/Swiss-Prot (swissprot). Большинство подаваемых на вход параметров были оставлены по умолчанию (порог на E-value = 0.05, матрица АК замен BLOSUM62, штраф за открытие гэпа = 11, за продление = 1, сохранен "Compositional ajustment" для борьбы с участками малой сложности), кроме длины слова - 3 (была попробована и выдача для длины слова равной 5). Текстовая выдача программы для max 100 находок. В данном случае находок ровно 100. Поэтому pBLAST был запущен заново с теми же параметрами, но уже с увеличенным числом максимально выдаваемых находок до 250. Текстовая выдача программы для max 250 находок, где находок уже 124. Было выбрано 5 находок с по возможности наибольшим процентом перекрытия последовательности-запроса. На рисунке 1 представлено их выранивание COBALT (без самой мальтоолигозилтрегалозо-синтазы). Можно уже заметить, что последние три последовательности отличаются от первых двух. Скачены fasta-форматы последовательностей выбранных белков, к файлу добавлена последовательность основного белка (данные можно найти по адресу /home/students/y22/aliserana/term2/pr10/seqdump.txt).

Проведено множественное выравнивание последовательностей через muscle:
muscle -in seqdump.txt -out q7lyv2.fasta

Множественное выравнивание было загружено в Jalview и раскрашено по проценту идентичности. По рисунку 2 видно, что Q9R9H8.2 наименее похож на остальных, удалим последовательность для него, поскольку скорее всего этот белок не гомологичен моему основному. После его удаления осталось много гэпов, поэтому последовательности заново были выравнены через встроенный в Jalview Muscle (все настройки оставлены по умолчанию). Результат виден на рисунке 4. Снова была удалена одна из предполагаемо неродственных последовательностей - для белка Q9X2F4.1, проведено перевыравнивание. На рисунке 5 представлена ситуация для 4 оставшихся белков. Был удален и белок O86956.1, после чего было получено итоговое выравнивание (рис 6, ссылка на проект Jalview). Вероятная неродственность всех удаленных белков моему основному определялась присутствием инделей на удаляемой последовательности и отсутствием гэпов на том же участке остальных, и наоборот, когда у всех последовательностей на каком-то участке индель, а у удаляемой смысловая последовательность.



| Accession | RecName | Organism | Query cover |
|---|---|---|---|
| Q44315.1 | Maltooligosyl trehalose synthase | Arthrobacter sp. Q36 | 90% |
| P9WQ20.1 | Putative maltooligosyl trehalose synthase | Mycobacterium tuberculosis CDC1551 | 89% |
| Q9R9H8.2 | Intracellular maltogenic amylase | Bacillus subtilis | 19% |
| O86956.1 | 4-alpha-glucanotransferase | Thermotoga neapolitana | 18% |
| Q9X2F4.1 | Cyclomaltodextrinase | Thermotoga maritima MSB8 | 16% |
В итоговом выравнивании видно, что у оставшихся последовательностей белков имеется большое количество участков высокой консервативности, что позволяет говорить о гомологоичности оставленных белков: Q7LYV2_SACSH, Q44315.1 и P9WQ20.1. Причем видно, что последние два белка больше похожи друг на друга (например, участки 160-172, 396-412), чем на основной, что отражено и на рисунке 3. Также можно по таблице 1 убедиться, что в итоге оказались гомологами белки с наибольшими процентами перкрытия и схожими рекомендованными названиями.
2. Поиск гомологов зрелого вирусного белка, вырезанного из полипротеина, в Swissprot
Запрос вирусного полипротеина: (protein_name:polyprotein) AND (taxonomy_id:10239) AND (reviewed:true). Решено было взять полипротеин pp62 из вируса африканской чумы свиней. Некоторые данные о нем представлены в таблице 2.
| RecName | ID | AC | Organism |
|---|---|---|---|
| Polyprotein pp62 | PP62_ASFB7 | Q65179; Q86851; | African swine fever virus (strain Badajoz 1971 Vero-adapted) |
| Название | Координаты в полипротеине |
|---|---|
| p15 | 2..158 |
| p35 | 159..463 |
| p8 | 464..530 |
В таблице 3 представлена информация о зрелых белках, на которые разрезается мой полипротеин. Был выбран белок p35. Средствами EMBOSS отдельный файл fasta-формата была вырезана последовательность зрелого белка и дано подходящее название и описание (при этом программа автоматически добавляет AC полипротеина, поэтому он был удален вручную в текстовом редакторе):
descseq -sequence 'sw:pp62_asfb7[159:463]' -name 'p35' -description 'id="PRO_0000373446"' -outseq segment.fasta
Был произведен поиск гомологов белка p35 по программе BLAST и алгоритму blastp в банке Swiss-Prot. Как и в прошлом задании почти все подаваемые на вход параметры оставлены по умолчанию, кроме длины слова = 2. Программа выдала всего лишь 4 гомолога. Текстовая выдача программы. Работаем с теми находками, которые есть. Создаем общий fasta-файл с их последовательностями: /home/students/y22/aliserana/term2/pr10/seq4.fasta. Выравниваем, используя muscle, и загружаем результат в Jalview. Итоговый проект в Jalview. В итоге видно, что участки каждой из находок гомологичны данной, что и можно было предположить из их названий - они все представляют собой разные варианты основного полипротеина.
3. Исследование зависимости E-value от объёма банка
Предыдущий поиск в BLAST был повторен с единственным изменением в фильтре по организмам на Viruses (taxid:10239). Список находок не изменился, потому что и раньше он состоял только из вирусных полипротеинов. К сожалению, E-value как и был 0.0, так и остался 0.0, поэтому оценка доли вирусных белков в Swissprot по этим результатам невозможна. Поэтому сначала была попытка провести оценку по результатам поиска для всего полипротеина pp62, но и там все E-value были равны 0.0. Тогда был взят другой полипротеин: POLG_ASGVP. AC: P36309; OS: Apple stem grooving virus (strain P-209). Результат выдачи поиска в BLAST по Swiss-Prot по всем организмам: текстовый файл. Результат при поиске по вирусам только: текстовый файл. Рассмотрим E-value для Q65652.1. В первом случае он , во втором - . Мы знаем теорему С.Карлина: . Два поиска отличались только базами данных. Параметры вычисления веса (матрицы и штрафы за гэпы) мы не меняли, поэтому константы и действительно не изменились, вес выравнивания () также остался постоянным. Последовательность в обоих случаях одинаковая, а значит ее длина не могла поменяться: . Получается, что изменение E-value связано только с - размером базы данных. E-value в первом случае больше в 20 раз, значит и база данных в первом случае в 20 раз больше. Получается, что доля вирусных белков в Swiss-Prot составляет приблизительно .