A- и В- формы ДНК. Структура РНК
Построение моделей структур A-, B- и Z-формы ДНК
С помощью инструментов пакета 3DNA построили модели структур A-, B- и Z-формы ДНК
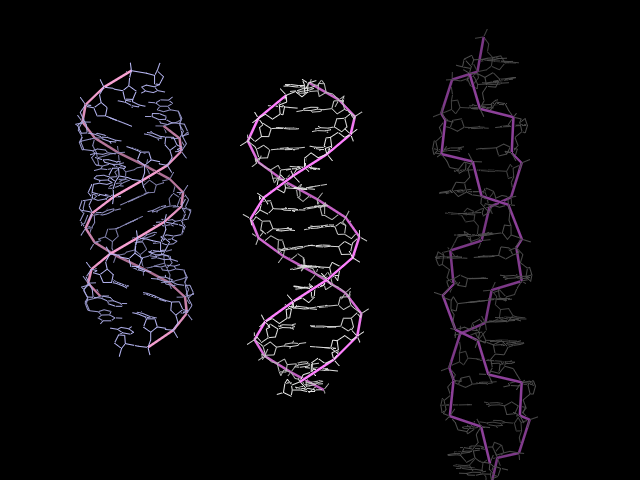
Рис.1 Формы ДНК
Слева направо А-форма ДНК, В-форма, Z-форма.
Сравнение структур 3-х форм ДНК с помощью средств JMol
Рис.2 Атомы большой и малой бороздок
Красным – атомы большой бороздки; синим – атомы малой бороздки.
В сторону большой бороздки обращены атомы [DG]9.c8, n7, c5, c6, o6.
В сторону малой бороздки обращены атомы [DG]9.c4, n3, c2, n2.
Остальные атомы основания не обращны явно в сторону большой или малой бороздки.
На самом деле, атомы с2, c5, c6 также не очень хорошо видны – их закрывают другие атомы.
Но все же они относятся к малой (с2) и большой (с5, с6) бороздкам.
В А- и Z-формах все указанные атомы обращены туда же.
Сравнение основных спиральных параметров разных форм ДНК.
В спирали В-ДНК мы можем различить 2 бороздки визуально одинаковые по глубине, но разные по ширине. Более широкая – большая бороздка, узкая – малая.
B A-ДНК одна бороздка спирали узкая и глубокая, атомы гуанина c8, n7, c5, c6, o6 обращены в сторону этой бороздки. Другая бороздка широкая и менее глубокая. Атомы c4, n3, c2, n2 гуанина обращены в сторону этой бороздки. Узкая и глубокая бороздка – это большая бороздка, широкая – малая бороздка.
Зигзагообразная левая спираль Z-ДНК также имеет два углубления. Мелкая широкая бороздка, в сторону которой обращены атомы c8, n7, c5, c6, o6, и узкая глубая бороздка, куда направлены атомы c4, n3, c2, n2 гуанина. Первая – большая бороздка, вторая – малая.
| A-форма | В-форма | Z-форма | |
|---|---|---|---|
| Тип спирали (правая/левая) | Правая | Правая | Левая |
| Шаг спирали, Å | 28,03 | 33,75 | 43,5 |
| Число основания на виток | 11 | 10 | 12 |
| Ширина малой бороздки, Å | 18,5 [DC]32:B.P #638 - [DG]5:A.P #83 |
13,2 [DC]28:B.P #556 - [DA]10:A.P #187 |
20,07 [DC]32:B.P #638 - [DC]16:A.P #310 |
| Ширина большой бороздки, Å | 9,63 [DT]3:A.P #44 - [DT]23:B.P #454 |
20,5 [DG]9:A.P #165 - [DT]19:B.P #372 |
23,5 [DC]4:A.P #64 - [DC]32:B.P #638 |
Сравнение торсионных углов в структурах А- и В-форм.
* Здесь и далее приведены торсионные углы для гуанина
| Сравнение торсионных углов в структурах А- и В-форм | |||||||
|---|---|---|---|---|---|---|---|
| угол | α | β | γ | δ | ε | ζ | χ |
| O3'(i-1)-P-O5'-C5' | P-O5'-C5'-C4' | O5'-C5'-C4'-C3' | C5'-C4'-C3'-O3' | C4'-C3'-O3'-P(i+1) | C3'-O3'-P(i+1)-O5'(i+1) | O4'-C1'-N9-C4 (для пуринов) |
|
| А-форма JMol |
-51,7° | 174,8° | 41,7° | 79.1° | -147,8° | -75,1° | -157,2° |
| В-форма JMol |
-29,9° | 136,4° | 31,1° | 143,4° | -140,8° | -160,5° | -98,0° |
Измеренные значения торсионных углов (по неизвестным причинам) не совпадают
со значениями, приведенными в презентации. Зато совпадают со значениями,
которые получили коллеги. Так что будем считать, что углы измерены правильно.
О торсионных углах в нуклетидах [1].
Определение параметров структур нуклеиновых кислот с помощью программ пакета 3DNA
Значения торсионных углов в А- и В-формах ДНК, полученные с помощью программ find-pair и analyze, полностью совпадают со значениями углов, полученными с помощью JMol.
Ниже приведены части выходных файлов программы analyze .
A-форма
В-форма
Z-форма
Определим торсионные углы для Z-ДНК
| Торсионные углы гуанина в Z-форме ДНК | |||||||
|---|---|---|---|---|---|---|---|
| угол | α | β | γ | δ | ε | ζ | χ |
| O3'(i-1)-P-O5'-C5' | P-O5'-C5'-C4' | O5'-C5'-C4'-C3' | C5'-C4'-C3'-O3' | C4'-C3'-O3'-P(i+1) | C3'-O3'-P(i+1)-O5'(i+1) | O4'-C1'-N9-C4 (для пуринов) |
|
| Z-форма | 52° | 179° | -173,8° | 94,9° | -103,6° | -64,8° | 58,7° |
Структура т-РНК
Торсионные углы
| Среднее значение торсионных углов гуанина т-РНК (pdb id 1n78) | |||||||
|---|---|---|---|---|---|---|---|
| угол | α | β | γ | δ | ε | ζ | χ |
| O3'(i-1)-P-O5'-C5' | P-O5'-C5'-C4' | O5'-C5'-C4'-C3' | C5'-C4'-C3'-O3' | C4'-C3'-O3'-P(i+1) | C3'-O3'-P(i+1)-O5'(i+1) | O4'-C1'-N9-C4 (для пуринов) O4'-C1'-N1-C2 (для пиримидинов) |
|
| т-РНК | -50,3° | 42,9° | 55,2° | 87,4° | -128,5° | -55,8° | -127,5° |
Нуклеотиды (G) с наиболее отклоняющимся значением какого-либо из торсионных углов
| Угол | alpha | beta | gamma | delta | epsilon | zeta | chi |
|---|---|---|---|---|---|---|---|
| № нуклеотида | 11 | 5, 18 | 11 | 28 | 29 | 29 | 11 |
т-РНК по структуре (по значениям торсионных углов) больше всего похожа на A-форму ДНК
Таблица с расчетамиCтруктурa водородных связей
Двутяжевые стебли т-РНК, имеющие постоянное число нуклеотидов, образованы комплементарными антипараллльными участками и представляют собой двойную спираль, по параметрам близкую к А-форме ДНК. На каждый виток этой спирали приходится 11 пар оснований. Структура т-РНК, которая характеризуется наличием четырех стеблей и трех петель, получила название "клеверный лист"[2]. Стебель с петлей формируют ветвь. На рис. 3 ниже оранжевым показана акцепторная ветвь, желтым – антикодоновая ветвь, содержащая антикодоновый триплет в составе своей петли. Слева и справа от нее расположены D- и T-ветви, соответственно названные так из-за присутствия в их петлях необычных консервативных нуклеозидов дигидроуридина (D) и тимидина (T). D-ветвь показана розовым, T-ветвь – зеленым.
В выходном файле конвейера find-pair | analyze описаны параметры структуры заданной т-РНК, в том числе структура водородных связей.
Файл t-RNA.out
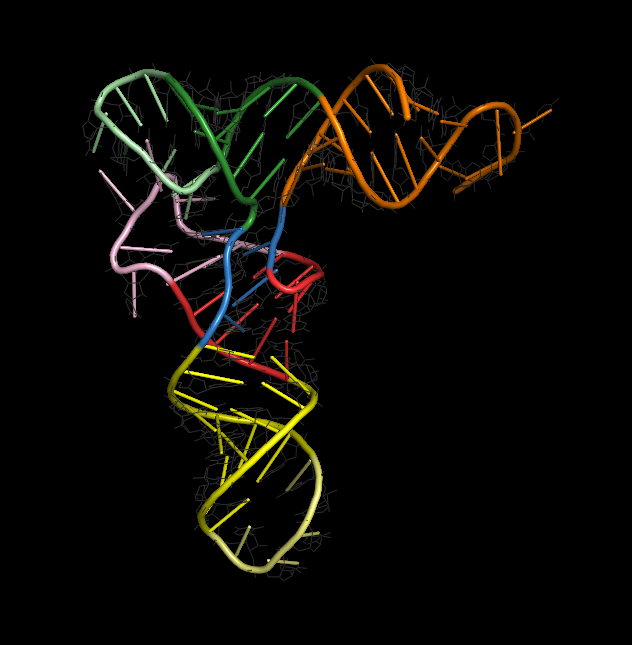 Рис.3 Третичная структура т-РНК; Разными цветами показаны стебли (ярко) и петли (бледно) |

Рис.4 Информация о нуклеотидах, образующих стебли. |
На картинке (и в приложенном pdf-документе, если нажать на картинку) мы можем видеть номера нуклетидов, образующих пары. Четыре стебля выделены разными цветами. Детальная информация о водородных связях указана ниже. Основания, образующие Уотсон-Криковские пары и входящие в стебли, соединены пятью черточками. Обозначения других – неканонических – пар включают звездочки. Вообще, неканоническими являются пары G-U, A-G, A-A, A-C, G-C с 2-мя связями между основаниями, но программа также отмечает пары A-U, не входящие в стебли.
Водородные связи, не входящие в стебли, важны для стабилизации третичной структуры т-РНК. "Клеверный лист" – это только удобная схема-развертка на плоскости. Третичная структура т-РНК формируется за счет взаимодействия элементов вторичной структуры. Она получила название L-формы из-за сходства с латинской буквой L (см. рис. 3). За счет стэкинга оснований акцепторный стебель и T-стебель клеверного листа образуют одну непрерывную двойную спираль (одну из “палочек” – доменов буквы L), а два других стебля – антикодоновый и D – другую непрерывную двойную спираль (второй домен L). При этом D- и T-петли оказываются сближенными и скрепляются между собой путем образования дополнительных, часто необычных пар оснований. Аналогичные третичные взаимодействия скрепляют и некоторые другие участки L-структуры [2]. В документе-описании пар оснований (рис. 4) пары, не входящие в стебли отмечены черным шрифтом.
Стекинг взаимодействия
Для обнаружения стэкинг-взаимодействий требуются данные о величине площади "перекрывании" 2-х последовательных пар азотистых оснований. Мы также можем получить их из файла t-RNA.out. Для пар с наибольшими значениями площади перекрывания более характерно стэкинг-взаимодействие.
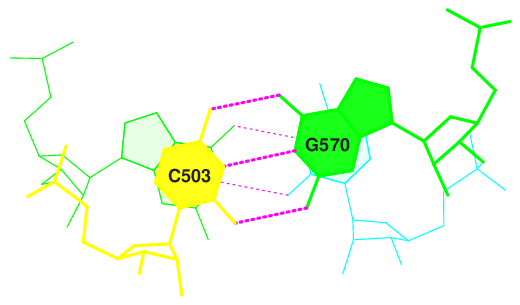
Рис. 5. Пары с наибольшей площадью перекрывания 13,8 Å^2
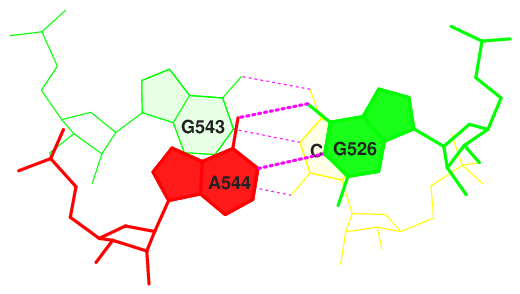
Рис. 6. Соседние пары с площадью перекрывания 6,8 Å^2
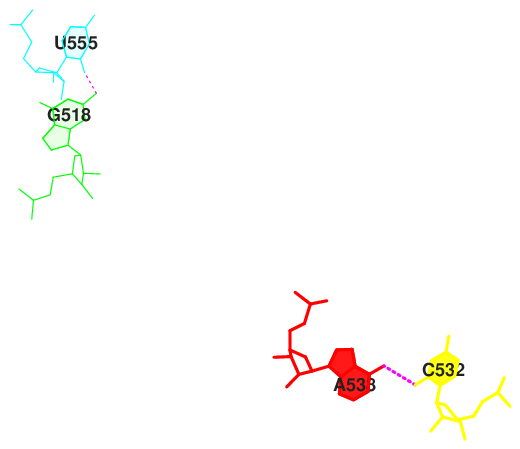
Рис. 7. Неперекрывающиеся пары оснований.
Ссылки
[1] W.Saenger. Principles of nucleic acid structure
[2] О.О.Фаворова. СТРОЕНИЕ ТРАНСПОРТНЫХ РНК И ИХ ФУНКЦИЯ НА ПЕРВОМ (ПРЕДРИБОСОМНОМ) ЭТАПЕ БИОСИНТЕЗА БЕЛКОВ. Текст статьи
