Практикум 14. Сборка генома de novo
Для выполнения данного практикума был загружен fastq-файл в .gz архиве из проекта по секвенированию бактерии Buchnera aphidicola по ссылке http://www.ebi.ac.uk/ena/data/view/SRR4240378. В папке /nfs/srv/databases/ngs/anton.vlasov/pr14 архив был распакован командой:
gunzip -d SRR4240378.fastq.gz
Задание 1. Подготовка чтений
Вначале полученный файл с ридами был проанализирован программой FastQC. Команда:
fastqc SRR4240378.fastq
Отчет работы программы: SRR4240378_fastqc.html. Всего содержится 4 420 587 ридов. Из отчёта видно, что в целом все чтения высокого качества, однако, есть проблемы с Per base sequence content, а также, с Kmer Content. Предупреждение выдано в пункте Overrepresented sequences: оно связано с поли-(А)-фрагментами, последовательностями из неопределенных нуклеотидов, а также, с адаптерами для Illumina секвенирования. Адаптеры необходимо удалить программой trimmomatic, предварительно собрав все адаптеры в один файл. Адаптера для Illumina были скопированы в рабочую директорию, а затем объединены в один файл следующей командой:
compseq *.fa -outseq adapters.fasta
Затем адаптеры были удалены из файла с ридами следующей командой:
java -jar /nfs/srv/databases/ngs/suvorova/trimmomatic/trimmomatic-0.30.jar SE -phred33 SRR4240378.fastq reads_good.fastq ILLUMINACLIP:adapters.fasta:2:7:7
Нуклеотиды с качеством чтения меньше 20 и риды с числом нуклеотидов меньше 30 были удалены командой:
java -jar /nfs/srv/databases/ngs/suvorova/trimmomatic/trimmomatic-0.30.jar SE -phred33 reads_good.fastq reads_better.fastq TRAILING:20 MINLEN:30
Файл reads_better.fastq был снова проанализирован программой FastQC, отчёт: reads_better_fastqc.html. Из повторяющихся последовательностей остались только поли-(А)-фрагменты, остальные были удалены. Выбросы, которые были видны на графике Per base sequence quality тоже пропали. Число ридов скоратилось до 4 163 033, то есть уменьшилось на 257 554 рида.
Задание 2. Подготовка k-меров
Библиотека k-меров была создана в папке kmers командой
velveth kmers 29 -fastq -short reads_better.fastq
Задание 3. Сборка контигов
Контиги были получены в папке kmers следующей командой:
velvetg kmers
Основные реузультаты работы программы: граф имеет 492 вершины, N50 = 14 910. Информация о контигах содержится в файле stats.txt. В файле contigs.fa содержится 177 контигов. Названия контигов были получены командой
grep \> *.fa > names.txt
Затем названия контигов были открыты в Excel для дальнейшего анализа.
Таблица с самыми длинными контигами:
| ID | Длина | Покрытие |
| 8 | 55699 | 27,43 |
| 6 | 32730 | 29,54 |
| 25 | 26591 | 27,35 |
Немного статистики о покрытии:
- Максимум: 140,52
- Верхний квартиль: 26,8
- Среднее: 18,46
- Медиана: 16,5
- Нижний квартиль: 4,66
- Минимум: 2,14
Обзор контигов с сильно отличающимся от типичного покрытием:
| ID | Длина | Покрытие | Комментарий |
| 55 | 926 | 140,52 | Контиг с самым большим покрытием. Результат поиска MegaBlast показывает, что данная последовательность соответствует плазмиде pTrp из Buchnera aphidicola. |
| 28 | 2108 | 137,55 | Второй по покрытию контиг, соответствует той же плазмиде. |
| 132 | 59 | 2,14 | Контиг с самым низким покрытием. Поиск в Blast для данной последовательности не дал результатов. Диина равна 2 k-мерам. |
Задание 4. Анализ контигов Megablast
Для выполнения данного задания 3 самых длинных контига были сравнены программой Megablast с хромосомой Buchnera aphidicola (GenBank/EMBL AC — CP009253).
Контиг №8 (длина: 55699 п.н.; покрытие: 27,43)
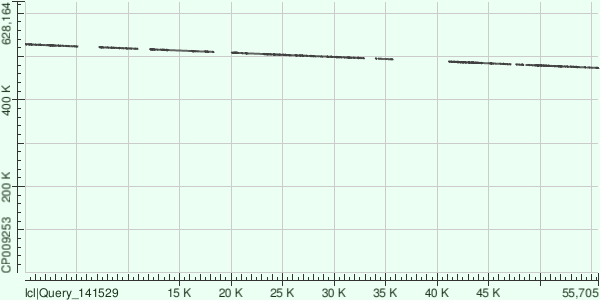
Исходя из карты локального сходства, можно заметить, что контиг инвертирован относительно последовательности генома бактерии. Было получено 11 выравниваний. Контиг на 76% идентичен с участками, с которыми он был выровнен. Он покрывает данные участки на 77%. Координаты начала выравнивания контига с хромосомой бактерии: 473 503, конца: 508 806.
Контиг №6 (длина: 32730 п.н.; покрытие: 29,54)

Исходя из карты локального сходства, можно заметить, что контиг инвертирован относительно последовательности генома бактерии. При этом контиг разбит на 2 части: половина была выровнена с началом хромосомы, другая половина - с концом. Было получено 5 выравниваний. Контиг на 78% идентичен с участками, с которыми он был выровнен. Он покрывает данные участки на 79%. Координаты нижнего фрагмента: 2004 - 17 919; верхнего: 613 658 - 627 104
Контиг №25 (длина: 26591 п.н.; покрытие: 27,35)

Данный контиг соответствует прямой цепи хромосомы. Всего было построено 2 выравнивания. Контиг идентичен с выровнеными фргаментами на 75%, покрывая их на 52%. Координаты фрагментов: 126623 - 127815 и 127825 - 140555.