 Реконструкция филогении по нуклеотидным последовательностям, паралоги.
Реконструкция филогении по нуклеотидным последовательностям, паралоги. Реконструкция филогении по нуклеотидным последовательностям, паралоги.
Реконструкция филогении по нуклеотидным последовательностям, паралоги.Для каждой из следующих бактерий:
Название |
Мнемоника |
Rhizobium etli |
RHIEC |
Burkholderia cenocepacia |
BURCA |
Ralstonia pickettii |
RALPJ |
Pseudomonas aeruginosa |
PSEAE |
Escherichia coli |
ECOLI |
Yersinia pseudotuberculosis |
YERPS |
Haemophilus influenzae |
HAEIN |
Pasteurella multocida |
PASMU |
Были взяты последовательности 16S рибосомальной РНК из базы полных геномов NCBI, они находятся в файлах с расширением '.frn'. Даллее при помощи JalView было построено их выранивание, по нему было реконструировано дерево в программе MEGA методом Neighbor-joining. Изображение полученного дерева представлено ниже (Рис.1).
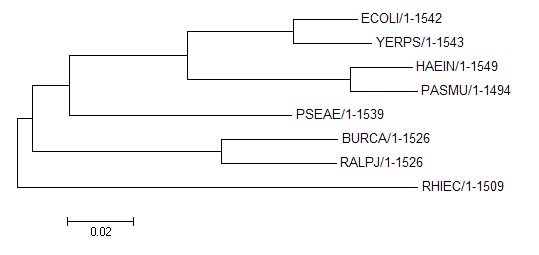
Рисунок 1. Дерево, построенное по нуклеотидным последовательностям.
Данное дерево совпадает с правильным, полученным ранее.
С помощью сервиса blastp на сайте NCBI были найдены гомологи белка CLPX_ECOLI в рассматриваемых бактериях (поиск проводился по протеомам этих бактерий). Последовательности этих гомологов были выравнены в JalView, дерево было построено в MEGA методом Neighbor-joining. Полученное дерево показано ниже(Рис.2).
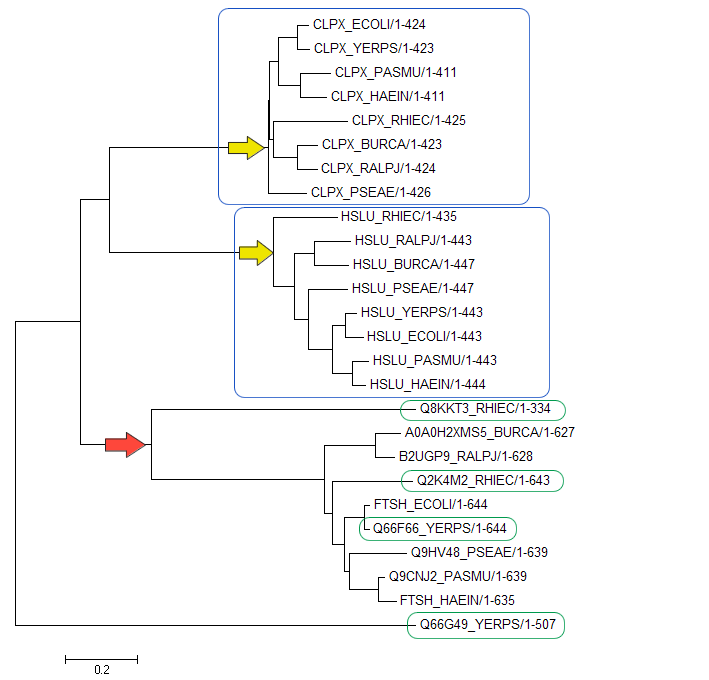
Рисунок 2. Изображение дерева.
Синими рамками на изображении дерева (Рис.2) показаны две группы попарно ортологичных белков (эти белки из разных организмов и разделение их общего предка на линии, ведущие к ним, произошло в результате видообразования), зелёным обведены паралоги (гомологичные белки из одного организма). Жёлтые стрелки указывают на разделение путей эволюции белков в результате видообразования. Красная стрелка отражает пример дупликации гена.