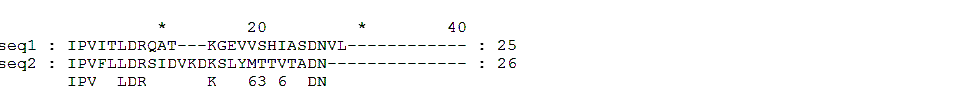BLASTP
на главную
Программа BLASTP
Таблица 1. Результаты поиска гипотетических гомологов белка RBSB_ECOLI
| |
Поиск по БД Swiss-Prot |
Поиск по БД PDB |
Поиск по БД "nr" |
| 1. Лучшая находка (в принципе должна соответствовать заданному белку) |
| Идентификатор БД |
RBSB_ECOLI |
1URP |
NP_418207.1 |
| E-value |
4e-167 |
2e-154 |
7e-166 |
| Вес (в битах) |
586 |
540 |
586 |
| % идентичности |
100% |
100% |
100% |
Найдены ли другие белки с теми же значениями E-value и веса в битах?
Если найдены, то укажите общее число и приведите один идентификатор |
не найдены |
не найдены |
не найдены |
| 2. Сколько хороших кандидатов в гомологи найдено?
(число находок в списке описаний, Descriptions,
с E-value < 1E-10)
| 25 |
24 |
99 |
| 2.
"Худшая" находка (последняя в выдаче с E-value < 1)
|
| Номер находки в списке описаний (Descriptions) |
124 |
90 |
6809 |
| Идентификатор БД |
PYRD_SCHPO |
2TOH |
YP_480637.1 |
| E-value |
0.84 |
0.74 |
0.99 |
| Вес (в битах) |
34.3 |
30.4 |
38.1 |
| % идентичности |
29% |
25% |
58% |
| % сходства |
49% |
43% |
78% |
| Длина выравнивания |
71 |
343 |
293 |
| Координаты выравнивания (номера первых и последних а.о.) |
query:с 218 по 281 а.о.
sbjct: c 364 по 434 а.о. |
query:с 92 по 171 а.о.
sbjct: с 34 по 102 |
query:с 3 по 295 а.о.
sbjct: с 1 по 293 |
| % гэпов
| 9% |
7% |
16% |
Изучаемый белок удалось найти в Swiss-Prot и "nr", а также его структуру в PDB
Описание одного и того же белка было сравнено в разных банках данных.
Результаты поиска по разным банкам данных отличаются:
Вес в битах совпадает в Swiss-Prot и "nr" (586). Вес в PDB меньше (540), т.к. туда вошла лишь
часть последовательности.
E-value выше всего в PDB (2e-154), т.к. была взята часть последовательности, поэтому вероятность случайного нахождения гомолога выше.
E-value в банке Swiss-Prot (4e-167) меньше, чем в «nr» (7e-166).
Процент идентичности в трех банках равен 100% (белок сравнивался сам с собой)
Число потенциальных гомологов в PDB наименьшее (24), наибольшее в "nr" (99).
Это объясняется разными размерами банков.
"Худшие" находки не совпадают. Объясняется объемами банков (чем больше банк,
тем больше случайных находок). Из "худших" находок самая лучшая в банке "pdb" (0.74), т.к. чем меньше
E-value, тем "лучше" белок (также это зависит от объема банка: чем больше, тем вероятность нахождения выше).
Поиск гипотетических гомологов изучаемого белка с фильтром по таксонам
Моя задача — для изучаемого белка E. coli найти лучшего гомолога в организмах таксона, филогенетически как можно более далекого от E. coli.
Для исследования предлагаются следующие таксоны: Homo sapiens, Archaea, Actinobacteria, Alteromonadales, Vibrionaceae (приведены в порядке приближения к E. coli).
Критерий: E-value<0,001.
Найден лучший гомолог в организме таксона Actinobacteria (taxid:201174)
| Номер находки в списке описаний |
1 |
| Идентификатор БД
| CELR_THEFU
|
| E-value |
8e-13 |
| Вес (в битах) |
69.3 |
| % идентичности |
28% |
| % сходства |
45% |
| Длина выравнивания |
340 |
|
Координаты выравнивания (номера первых и последних
а. о.) |
query:с 27 по 248
sbjct:с 65 по 292 |
| % гэпов |
8% |
Поиск белка по его фрагменту
Дан фрагмент:
>seq2
IPVFLLDRSIDVKDKSLYMTTVTADN
С помощью SRS я нашла полную последовательность белка:
Swiss-Prot ID: YTFQ_ECOLI
Swiss-Prot AC: P39325; Q2M678
>seq2
MWKRLLIVSA VSAAMSSMAL AAPLTVGFSQ VGSESGWRAA ETNVAKSEAE KRGITLKIAD
GQQKQENQIK AVRSFVAQGV DAIFIAPVVA TGWEPVLKEA KDAEIPVFLL DRSIDVKDKS
LYMTTVTADN ILEGKLIGDW LVKEVNGKPC NVVELQGTVG ASVAIDRKKG FAEAIKNAPN
IKIIRSQSGD FTRSKGKEVM ESFIKAENNG KNICMVYAHN DDMVIGAIQA IKEAGLKPGK
DILTGSIDGV PDIYKAMMDG EANASVELTP NMAGPAFDAL EKYKKDGTMP EKLTLTKSTL
YLPDTAKEEL EKKKNMGY
Таблица 2. Результаты поиска белка в Swiss-Prot по фрагменту последовательности
| |
Поиск по фрагменту |
Поиск по полной
последовательности |
| АС лучшей находки |
P39325 |
P39325 |
| E-value |
1e-17 |
0.0 |
| Вес (в битах) |
86.7 |
645 |
Найдены ли другие белки с теми же значениями E-value и веса в битах?
|
не найдены |
не найдены |
Т.к. вес полной последовательности белка больше, чем вес фрагмента,
то E-value полной последовательности будет меньше (0.0 < 1e-17).
Среди результатов последнего поиска есть выравнивание YTFQ_ECOLI (query) с моим изучаемым белком RBSB_ECOLI (sbjct):
Query 1 MWKRLLIVSAV--SAAMSSMALAAPLTVGFSQVGSESGWRAAETNVAKSEAEKRGITLKI 58
M K +VSAV SA +S+ A+A T+ + + + + A+ EA+K G L +
Sbjct 3 MKKLATLVSAVALSATVSANAMAKD-TIALVVSTLNNPFFVSLKDGAQKEADKLGYNLVV 61
Query 59 ADGQQKQENQIKAVRSFVAQGVDAIFIAPVVATGWEPVLKEAKDAEIPVFLLDRSIDVKD 118
D Q ++ V+ +G + I P + +K A A IPV LDR
Sbjct 62 LDSQNNPAKELANVQDLTVRGTKILLINPTDSDAVGNAVKMANQANIPVITLDRQ---AT 118
Query 119 KSLYMTTVTADNILEGKLIGDWLVKEVNGKPCNVVELQGTVGASVAIDRKKGFAEAIKNA 178
K ++ + +DN+L GK+ GD++ K+ G+ V+ELQG G S A +R +GF +A+ A
Sbjct 119 KGEVVSHIASDNVLGGKIAGDYIAKKA-GEGAKVIELQGIAGTSAARERGEGFQQAVA-A 176
Query 179 PNIKIIRSQSGDFTRSKGKEVMESFIKAENNGKNICMVYAHNDDMVIGAIQAIKEAGLKP 238
++ SQ DF R KG VM++ + A ++ V+A ND+M +GA++A++ AG
Sbjct 177 HKFNVLASQPADFDRIKGLNVMQNLLTAH---PDVQAVFAQNDEMALGALRALQTAG--- 230
Query 239 GKDILTGSIDGVPDIYKAMMDGEANASVELTPNMAG 274
D++ DG PD KA+ DG+ A++ P+ G
Sbjct 231 KSDVMVVGFDGTPDGEKAVNDGKLAATIAQLPDQIG 266
Выравнивание последовательностей seq1(RBSB_ECOLI) и seq2(YTFQ_ECOLI) с помощью GenDoc (из пробного выравнивания):
>seq1 IPVITLDRQATKGEVVSHIASDNVL
>seq2 IPVFLLDRSIDVKDKSLYMTTVTADN
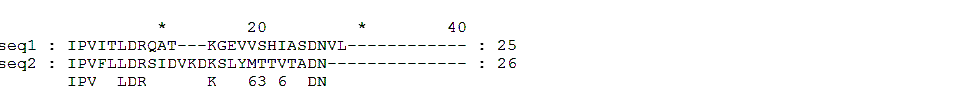
Пробное выравнивание не совпало с выравниванием в BLASTP (RBSB_ECOLI- seq1 и sbjct; YTFQ_ECOLI- seq2 и query):
они отличаются положением гэпов
(в пробном выравнивании они начинаются после треонина (T), а
в выравнивании с помощью BLAST после глутамина (Q).
пробное выравнивание:
seq2 : IPVFLLDRSIDVKDKSLYMTTVTADN--
seq1 : IPVITLDRQAT---KGEVVSHIASDNVL
с помощью BLASTP (фрагмент):
IPVFLLDRSIDVKDKSLYMTTVTADN
IPV LDR K ++ + +DN
IPVITLDRQ---ATKGEVVSHIASDNVL
Сравнение выравниваний, выданных программой BLASTP, с оптимальными глобальным и локальным выравниваниями.
Параметры выравнивания в BLAST:
Штраф за удлинение гэпа 1
Штраф за создание гэпа 11
a) Оптимальное локальное выравнивание с помощью программы water
b) Оптимальное глобальное выравнивание с помощью программы needle
Сравним полученные три выравнивания
в BLAST:
YTFQ_ECOLI 1 MWKRLLIVSAV--SAAMSSMALAAPLTVGFSQVGSESGWRAAETNVAKSEAEKRGITLKI 58
M K +VSAV SA +S+ A+A T+ + + + + A+ EA+K G L +
RBSB_ECOLI 3 MKKLATLVSAVALSATVSANAMAKD-TIALVVSTLNNPFFVSLKDGAQKEADKLGYNLVV 61
YTFQ_ECOLI 59 ADGQQKQENQIKAVRSFVAQGVDAIFIAPVVATGWEPVLKEAKDAEIPVFLLDRSIDVKD 118
D Q ++ V+ +G + I P + +K A A IPV LDR
RBSB_ECOLI 62 LDSQNNPAKELANVQDLTVRGTKILLINPTDSDAVGNAVKMANQANIPVITLDRQ---AT 118
YTFQ_ECOLI 119 KSLYMTTVTADNILEGKLIGDWLVKEVNGKPCNVVELQGTVGASVAIDRKKGFAEAIKNA 178
K ++ + +DN+L GK+ GD++ K+ G+ V+ELQG G S A +R +GF +A+ A
RBSB_ECOLI 119 KGEVVSHIASDNVLGGKIAGDYIAKKA-GEGAKVIELQGIAGTSAARERGEGFQQAVA-A 176
YTFQ_ECOLI 179 PNIKIIRSQSGDFTRSKGKEVMESFIKAENNGKNICMVYAHNDDMVIGAIQAIKEAGLKP 238
++ SQ DF R KG VM++ + A ++ V+A ND+M +GA++A++ AG
RBSB_ECOLI 177 HKFNVLASQPADFDRIKGLNVMQNLLTAH---PDVQAVFAQNDEMALGALRALQTAG--- 230
YTFQ_ECOLI 239 GKDILTGSIDGVPDIYKAMMDGEANASVELTPNMAG 274
D++ DG PD KA+ DG+ A++ P+ G
RBSB_ECOLI 231 KSDVMVVGFDGTPDGEKAVNDGKLAATIAQLPDQIG 266
Глобальное выравнивание:
RBSB_ECOLI 1 MNMKKLATLVSAVALSATVSANAMAKD-TIALVVSTLNNPFFVSLKDGAQ 49
|.|...:|||| ||.:|:.|:|.. |:........:.:..:..:.|:
YTFQ_ECOLI 1 --MWKRLLIVSAV--SAAMSSMALAAPLTVGFSQVGSESGWRAAETNVAK 46
RBSB_ECOLI 50 KEADKLGYNLVVLDSQNNPAKELANVQDLTVRGTKILLINPTDSDAVGNA 99
.||:|.|..|.:.|.|.....::..|:....:|...:.|.|..:......
YTFQ_ECOLI 47 SEAEKRGITLKIADGQQKQENQIKAVRSFVAQGVDAIFIAPVVATGWEPV 96
RBSB_ECOLI 100 VKMANQANIPVITLDRQ---ATKGEVVSHIASDNVLGGKIAGDYIAKKA- 145
:|.|..|.|||..|||. ..|...::.:.:||:|.||:.||::.|:.
YTFQ_ECOLI 97 LKEAKDAEIPVFLLDRSIDVKDKSLYMTTVTADNILEGKLIGDWLVKEVN 146
RBSB_ECOLI 146 GEGAKVIELQGIAGTSAARERGEGFQQAVA-AHKFNVLASQPADFDRIKG 194
|:...|:||||..|.|.|.:|.:||.:|:. |....::.||..||.|.||
YTFQ_ECOLI 147 GKPCNVVELQGTVGASVAIDRKKGFAEAIKNAPNIKIIRSQSGDFTRSKG 196
RBSB_ECOLI 195 LNVMQNLLTAH---PDVQAVFAQNDEMALGALRALQTAG---KSDVMVVG 238
..||::.:.|. .::..|:|.||:|.:||::|::.|| ..|::...
YTFQ_ECOLI 197 KEVMESFIKAENNGKNICMVYAHNDDMVIGAIQAIKEAGLKPGKDILTGS 246
RBSB_ECOLI 239 FDGTPDGEKAVNDGKLAATIAQLPDQIGAKGVETADKVLKGEKVQAKYPV 288
.||.||..||:.||:..|::...|:..| .|...|:..|.....|.
YTFQ_ECOLI 247 IDGVPDIYKAMMDGEANASVELTPNMAG-----PAFDALEKYKKDGTMPE 291
RBSB_ECOLI 289 DLKLVVKQ------------------- 296
.|.|....
YTFQ_ECOLI 292 KLTLTKSTLYLPDTAKEELEKKKNMGY 318
Локальное выравнивание:
RBSB_ECOLI 3 MKKLATLVSAVALSATVSANAMAKD-TIALVVSTLNNPFFVSLKDGAQKE 51
|.|...:|||| ||.:|:.|:|.. |:........:.:..:..:.|:.|
YTFQ_ECOLI 1 MWKRLLIVSAV--SAAMSSMALAAPLTVGFSQVGSESGWRAAETNVAKSE 48
RBSB_ECOLI 52 ADKLGYNLVVLDSQNNPAKELANVQDLTVRGTKILLINPTDSDAVGNAVK 101
|:|.|..|.:.|.|.....::..|:....:|...:.|.|..:......:|
YTFQ_ECOLI 49 AEKRGITLKIADGQQKQENQIKAVRSFVAQGVDAIFIAPVVATGWEPVLK 98
RBSB_ECOLI 102 MANQANIPVITLDRQ---ATKGEVVSHIASDNVLGGKIAGDYIAKKA-GE 147
.|..|.|||..|||. ..|...::.:.:||:|.||:.||::.|:. |:
YTFQ_ECOLI 99 EAKDAEIPVFLLDRSIDVKDKSLYMTTVTADNILEGKLIGDWLVKEVNGK 148
RBSB_ECOLI 148 GAKVIELQGIAGTSAARERGEGFQQAVA-AHKFNVLASQPADFDRIKGLN 196
...|:||||..|.|.|.:|.:||.:|:. |....::.||..||.|.||..
YTFQ_ECOLI 149 PCNVVELQGTVGASVAIDRKKGFAEAIKNAPNIKIIRSQSGDFTRSKGKE 198
RBSB_ECOLI 197 VMQNLLTAH---PDVQAVFAQNDEMALGALRALQTAG---KSDVMVVGFD 240
||::.:.|. .::..|:|.||:|.:||::|::.|| ..|::....|
YTFQ_ECOLI 199 VMESFIKAENNGKNICMVYAHNDDMVIGAIQAIKEAGLKPGKDILTGSID 248
RBSB_ECOLI 241 GTPDGEKAVNDGKLAATIAQLPDQIG 266
|.||..||:.||:..|::...|:..|
YTFQ_ECOLI 249 GVPDIYKAMMDGEANASVELTPNMAG 274
Глобальное и BLASTP выравнивания отличаются следующими сопоставлениями:
в BLASTP: с 247 по 274 а.о.(YTFQ_ECOLI); с 239 по 266 (RBSB_ECOLI)
YTFQ_ECOLI 247 IDGVPDIYKAMMDGEANASVELTPNMAG 274
DG PD KA+ DG+ A++ P+ G
RBSB_ECOLI 239 FDGTPDGEKAVNDGKLAATIAQLPDQIG 266
в глобальном: с 247 по 318 а.о.(YTFQ_ECOLI); с 239 по 296 (RBSB_ECOLI)
RBSB_ECOLI 239 FDGTPDGEKAVNDGKLAATIAQLPDQIGAKGVETADKVLKGEKVQAKYPV 288
.||.||..||:.||:..|::...|:..| .|...|:..|.....|.
YTFQ_ECOLI 247 IDGVPDIYKAMMDGEANASVELTPNMAG-----PAFDALEKYKKDGTMPE 291
RBSB_ECOLI 289 DLKLVVKQ------------------- 296
.|.|....
YTFQ_ECOLI 292 KLTLTKSTLYLPDTAKEELEKKKNMGY 318
Т.к. выравнивание в BLASTP локальное, то выравнивание закончилось на позициях
274 и 266 в белках YTFQ_ECOLI и RBSB_ECOLI соответственно в отличие от глобального,
а началось в белке RBSB_ECOLI не с первой аминокислоты, а со второй.
Выравнивании в BLASTP и в локальном совпадают.
| |
BLASTP |
needle |
water |
| Длина |
276 |
327 |
276 |
| Идентичность |
31% |
28.7% |
31.9% |
| Сходство |
51% |
45.9% |
51.8% |
| Гэпы |
5% |
12.2% |
5.1% |
| Score(вес) |
311 |
326 |
332 |
Все выравнивания отличаются друг от друга.
Локальное выравнивание имеет такую же длину, что и в BLASTP.
Из таблицы видно, что числа в локальном выравнивании и в BLASTP числа отличаются лишь тем,
что в локальном они даются с десятыми (одна цифра после запятой).
Разный вес возможно объясняется различиями в системе оценки выравниваний (
в матрице весов) в BLASTP и в EMBOSS.
Наибольшая длина в глобальном выравнивании, т.к. сравнивается полная последовательность, а не куски.
Глобальное выравнивание отличается от выравнивания в BLASTP: длина в глобальном
выравнивании больше, т.к. сравнивается полная последовательность, а не частичная, как в BLASTP.
Идентичность, сходство, гэпы и вес отличаются.
©Старовойтова Анна,2008