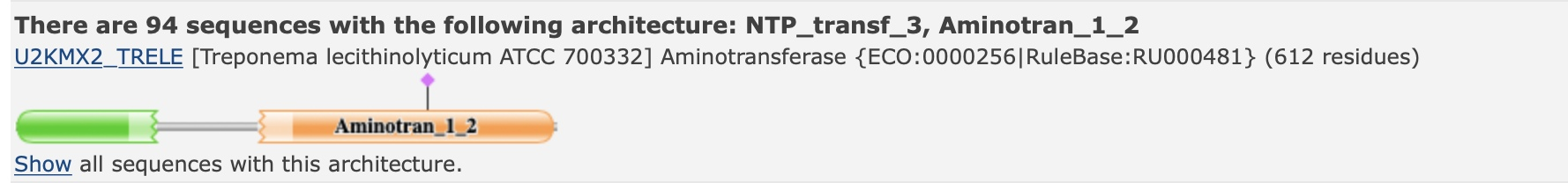
В базе данных Pfam я нашла двудоменную архитектуру, которая представлена у бактерий и встречается в 94 белках. Она включает ModA-подобный NTP-трансферазный домен (MobA-like NTP transferase domain) и домен аминотрансфераз класса 1 и 2. Данный представлены в Таблице 1.
| ID | AC | Название | Число находок в Uniprot |
| PF12804 | NTP_transf_3 | MobA-like NTP transferase domain | 78607 |
| PF00155 | Aminotran_1_2 | Aminotransferase class 1 and 2 | 387845 |
Поиск бактериальных белков, включающих оба этих домена, я проводила в бд Uniprot. Всего было найдено 466 белков, удовлетворяющих данному запросу.
Таблицу с белками можно загрузить по ссылке.

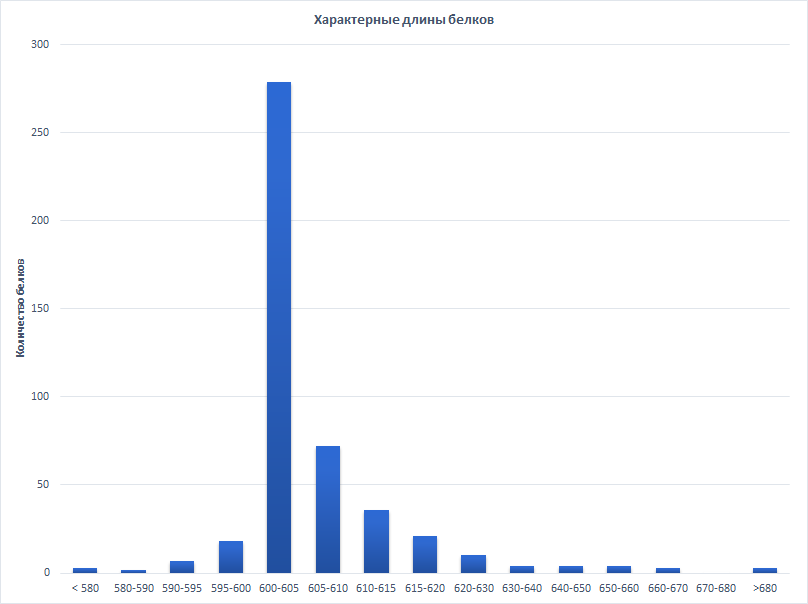
Для длин белков из таблицы была построена гистограмма. Более половины белков (279) находятся в узком диапазоне длины от 600 до 605 АК - этот интервал и будем считать характерной длиной белка.
Затем из общей таблицы я извлекла 45 белков характерной длины с помощью фильтрации и сортировки данных в Excel. Я выбирала несколько представителей из каждого семейства. Всего в таблице встретилось 32 семейства, в некоторые входило по одному-два представителя. У части бактерий не была указана принадлежность к таксону. Информацию об отборе можно увидеть в Таблице 2.
Выборку можно увидеть в таблице из Задания 2.
| # | Отдел | Семейство | Размер выборки |
| 1 | Firmicutes | Clostridiaceae | 4 |
| 2 | Firmicutes | Peptoniphilaceae | 2 |
| 3 | Firmicutes | Lachnospiraceae | 5 |
| 4 | Firmicutes | Peptoniphilaceae | 2 |
| 5 | Firmicutes | Oscillospiraceae | 2 |
| 6 | Firmicutes | Enterococcaceae | 2 |
| 7 | Eubacteriaceae | Eubacterium | 3 |
| 8 | Bacteroidetes | Bacteroidaceae | 5 |
| 9 | Bacteroidetes | Muribaculaceae | 4 |
| 10 | Bacteroidetes | Odoribacteraceae | 2 |
| 11 | Bacteroidetes | Prevotellaceae | 3 |
| 12 | Bacteroidetes | Tannerellaceae | 5 |
| 13 | Bacteroidetes | Porphyromonadaceae | 2 |
| 14 | Spirochaetes | Spirochaetaceae | 2 |
| 15 | Actinobacteria | Atopobiaceae | 2 |
Выравнивание выборки белков с двухдоменной архитектурой
Затем я получила выравнивание 45 белков в Jalview. Для этого я выбрала Fetch Sequences, затем Uniprot. В открывшемся окне я вставила AC белков, скопированные из колонки Excel. Выравнивание я выполнила с помощью Muscle с параметрами по умолчанию.
Выравнивание в формате fasta доступно по ссылке.
Ревизия белковых последовательностей
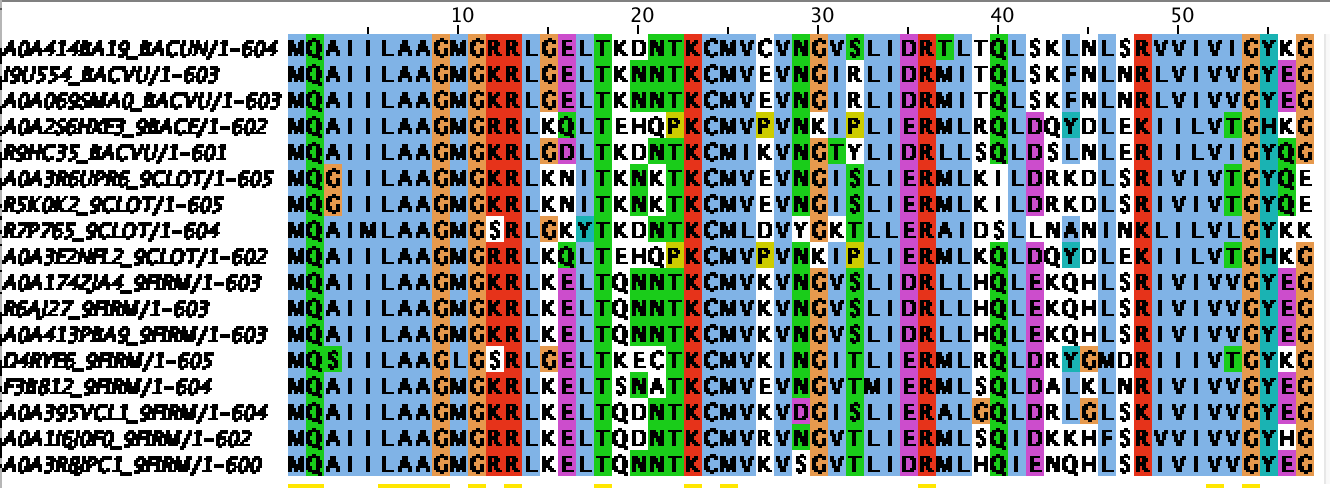
N-концевой консервативный блок начинался с первого позиции для всех последовательностей, и удалять ничего не пришлось. Фрагмент выравнивания N-концевого консервативного блока можно увидеть на Рисунке 3.
Я удалила 6 позиций после последнего C-концевого домена. Фрагмент выравнивания C-концевого консервативного блока можно увидеть на Рисунке 4

Проект в Jalview можно загрузить по ссылке.
Построение профиля
Я построения профиля я использовала пакет HMMER. Для начала я построила профиль по полученному выравниванию с помощью команды:
hmm2build -g hmm_build bac_proteins.fasta
Затем я откалибровала профиль с помощью следующей команды:
hmm2calibrate hmm_build
Получить файл с профилем выравнивания после калибровки можно по ссылке.
Проверка профиля
Чтобы проверить полученный профиль, я загрузила белковые последовательности бактерий, содержащий один домен из исследуемых. Из двух доменов я выбрала ModA-подобный NTP-трансферазный домен (PF12804). Таких белков нашлось 78607. Среди белков, содержащих один из доменов, программа ищет белки с двухдоменной архитектурой по профилю выравнивания. Я использовала следующую команду, установив порог по E-value 0.1 и порог по Score 0 (мне показалось, что отрицательный Score выглядит печально):
hmm2search -E 0.1 -T 0 hmm_build1 pr_seq.fasta > hmm_result.fasta
Скачать файл hmm_result.fasta по ссылке.
Сравнение списка находок
Я захотела сравнить, насколько похож список белков, составленный программой hmm2, со списком белков, полученным из Uniprot по двум доменам. Результат работы программы из файла hmm_result.fasta я загрузила в Excel, в таблице так же находятся расчеты и графики. Ссылка на скачивание таблицы.
Сначала я отсортировала находки по убыванию их веса. Затем я построила график распределения весов находок (Рисунок 5).
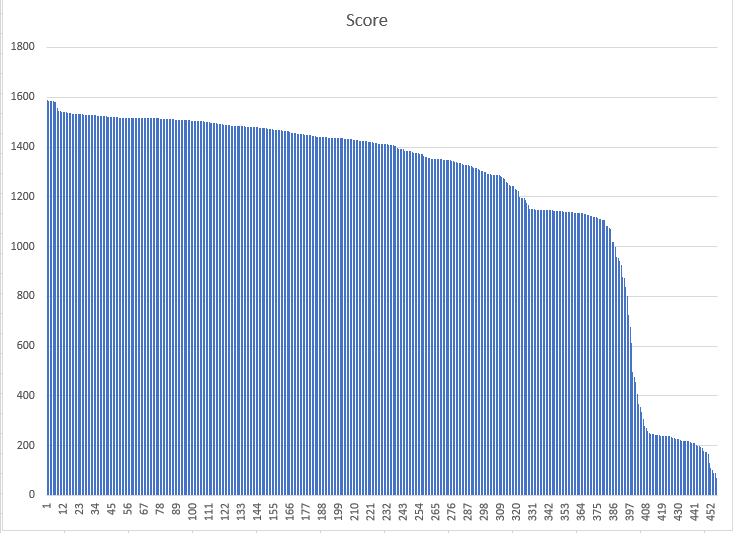
Поиск порога для предсказания доменной архитектуры
Далее моей целью было подобрать оптимальный порог веса для более менее точного предсказания находок с нужной доменной архитектурой. Для этого нужно найти баланс между чувствительностью теста и его мощностью (1 - специфичность). Справиться с этой задачей помогает построение ROC-кривой. Чтобы её построить, я перебирала всевозможные пороги (строчки в таблице) и для каждого из них показала чувствительность и специфичность предсказания. В отдельной колонке в таблице я посчитала F1-Score и приняла за пороговый вес тот, где этот параметр был наибольшим.
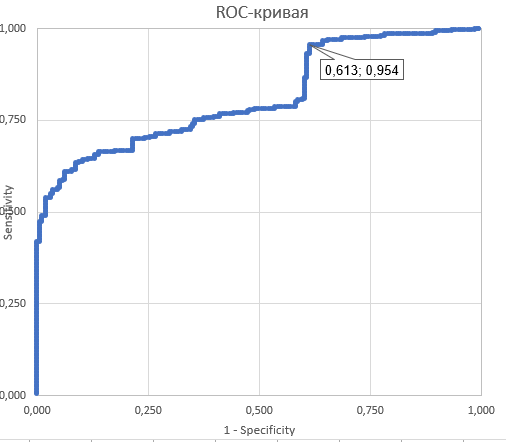
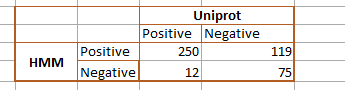
Выбранная специфичность и чувствительность достигается при установлении порога 1127,3 на Score. Такой вес располагается на 372 строке в таблице, отсортированной по убыванию веса. То есть можно увидеть, что данный порог вполне согласуется с графиком весов на Рисунке 5, где примерно на этих позициях происходит резкое снижение Score.
Если бы порог я выбирала, не обращаясь к F1-Score, то я бы стремилась к более высокой специфичности моего предсказания.
Еще я построила таблицу (Рисунок 7), по которой можно судить о качестве моего предсказания. Строила я её относительно таблицы, которую я получила со Score > 0 и E-value < 0.05.