Практикум 10. Программа BLAST
Гомологи белка Ketol-acid reductoisomerase (NADP(+)) белка в Swiss-Prot
Поскольку ни одного белка моей бактерии нет в базе данных Swiss-Prot, я выбрала белок Ketol-acid reductoisomerase (NADP(+)), он относится к Leptospira borgpetersenii serovar Hardjo-bovis (strain L550), то есть к родственной бактерии (UniProt AC Q056H8).
При запуске BLAST были заданы следующие параметры:
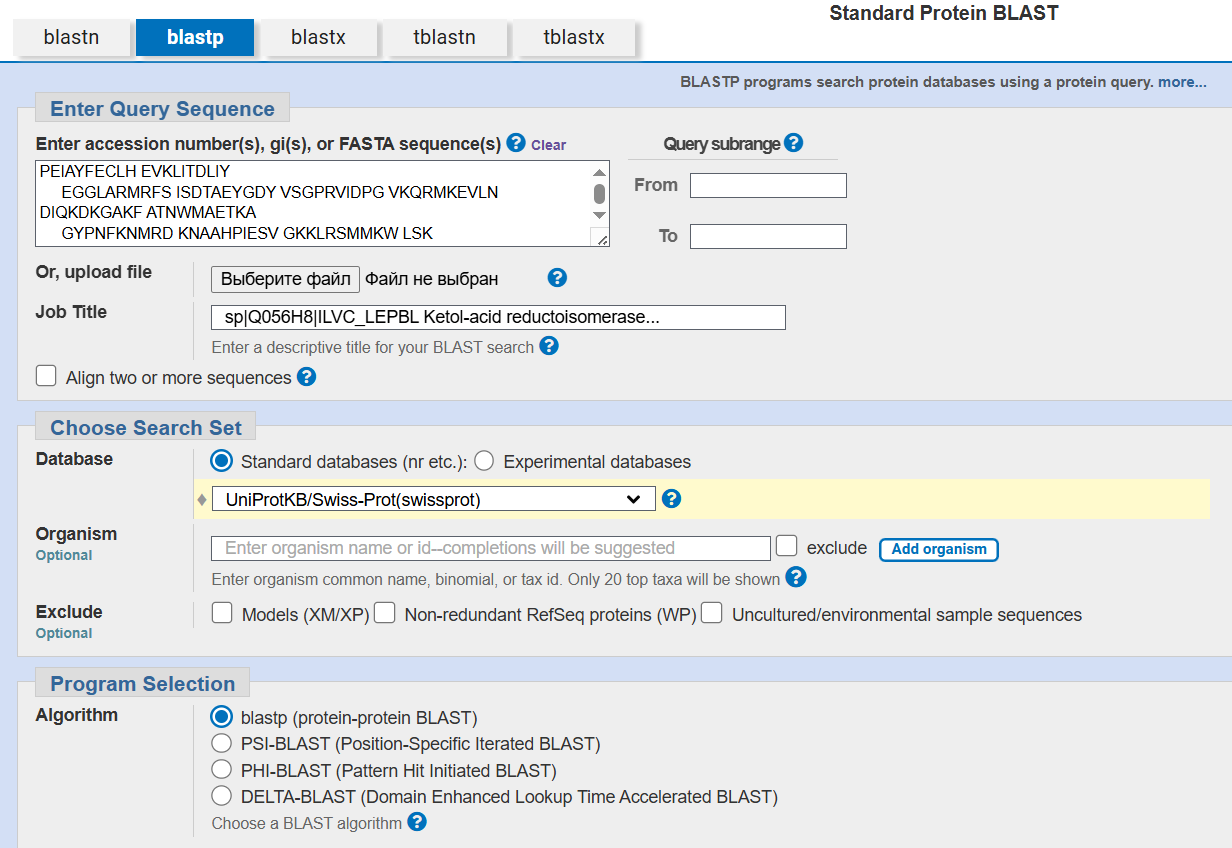
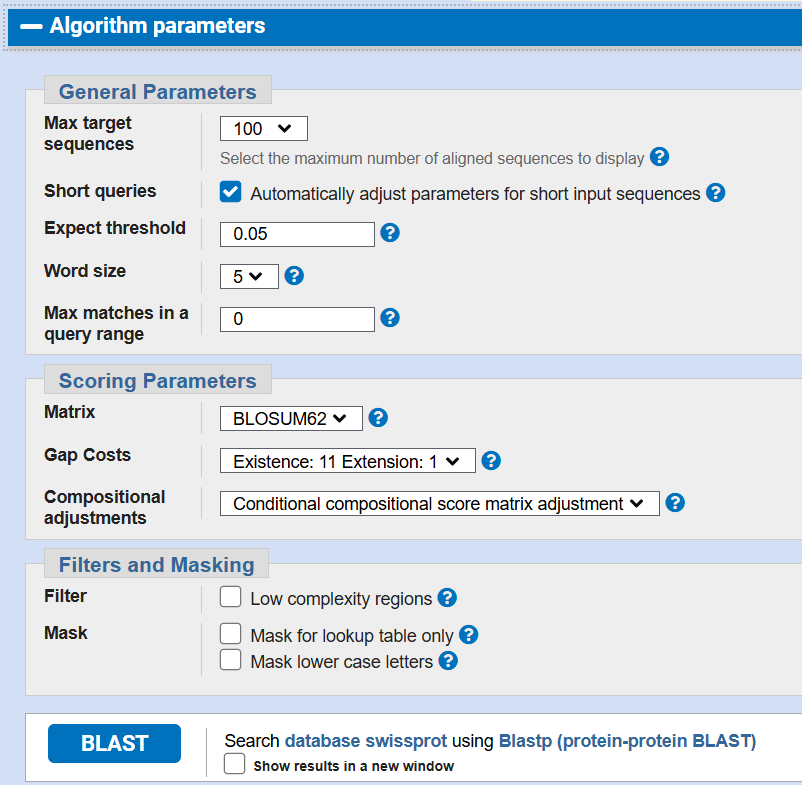
Текстовая выдача такого запроса в BLAST: Текстовая выдача программы
Также было сделанно множественное выравнивание изучаемого белка и 7 первых в выдаче BLAST
В выравнивании я оставила все 8 последовательностей, поскольку есть участки, которые имеют полностью совпадающие аминокислоты (26-34, 129-141, 178-198, 226-237)
Гомологи зрелого вирусного белка, вырезанного из полипротеина
Для задания я выбрала полипротеин вируса Feline immunodeficiency virus (isolate Petaluma) (FIV)
Белок Integrase (UniProtKB:P04585)
Координаты белка в полипротеине: 844..1124
Файл с вырезанным вирусным белком
Текстовая выдача такого запроса в BLAST (все параметры запроса аналогичны пункту выше): Текстовая выдача программы
Также было сделанно множественное выравнивание изучаемого белка и 7 первых в выдаче BLAST. В данном выравнивании я скрыла одну последовательность - TraF protein (IniProt AC A0A858XWF6), так как она сильно отличается от выбранного белка.
Исследование зависимости E-value от объёма банка
Запрос с фильтром по организмам, ограничив поиск вирусами (Viruses), незначительно отличается от аналогичного без фильтра (примерно 2-4 последовательности)
Для находки - интеграза Simian immunodeficiency virus (AGM155 ISOLATE) (P27973.2) изменилось значение E-value. До применения фильтра по организмам, только вирусам, значение составляло - 3e-60, а после - 1e-61. Данное значение определяется формулой: E-value=Kmn·e -λS, где n — размер базы данных (суммарная длина всех последовательностей)
Если первое значение (3e-60) - это ожидаемое количество случайных находок как вирусных, так и не вирусных последовательностей, а второе число (1e-61) - это ожидаемое количество случайных находок только вирусных последовательностей, то для того чтобы узнать долю вирусных белков в Swiss-Prot нужно: 3e-60 * 100% / 1e-61≈3.3%
То есть доля вирусных белков в Swiss-Prot составляет примерно 3.3%. Также стоит учесть, что в зависимости от выбраной последовательности это значение может отличаться, поэтому более правильно сказать, что доля вирусных белков в Swiss-Prot составляет примерно 3-5%
Текстовая выдача такого запроса в BLAST: Текстовая выдача программы