Построение профиля
Для построения профиля выберем хорошее подсемейство из выравнивания прошлого практикума. Так же полезно посмотреть на
дерево, наглядое изображение выравнивания. Возьмем в качестве подсемейства следующие последовательности:
- 2E_A0A067RGX7_ZOONE
- 2C_A0A026W6Y7_CERBI
- 2C_A0A067QWK2_ZOONE
- 2C_A0A087RBN7_APTFO
- 2E_A0A067REM1_ZOONE
- 2E_A0A084W1I6_ANOSI
- >2E_A0A084W1I7_ANOSI
Это представители второй доменной архитектуры, образующие наиболее внятную выборку. Других хороших выборок данного подсемейства нет. Последовательности представителей подсемейства занесены в отдельный файл.
- 2E_A0A067RGX7_ZOONE
- 2C_A0A026W6Y7_CERBI
- 2C_A0A067QWK2_ZOONE
- 2C_A0A087RBN7_APTFO
- 2E_A0A067REM1_ZOONE
- 2E_A0A084W1I6_ANOSI
- >2E_A0A084W1I7_ANOSI
Это представители второй доменной архитектуры, образующие наиболее внятную выборку. Других хороших выборок данного подсемейства нет. Последовательности представителей подсемейства занесены в отдельный файл.
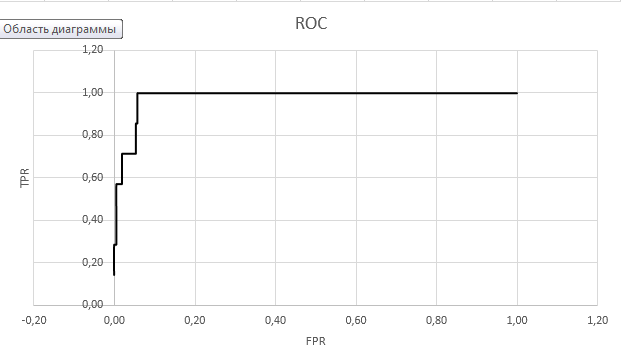
Для построения и калибровки профиля использовался пакет HMMER. Построили и откалибровали профиль. Сделали поиск по белкам Uniprot из практикума 11. Результаты поиска были записаны в отдельный файл, который обрабатывался с помощью Excel. Итоговая таблица. По приведенным в таблице данным построили ROC кривую (рис ниже).
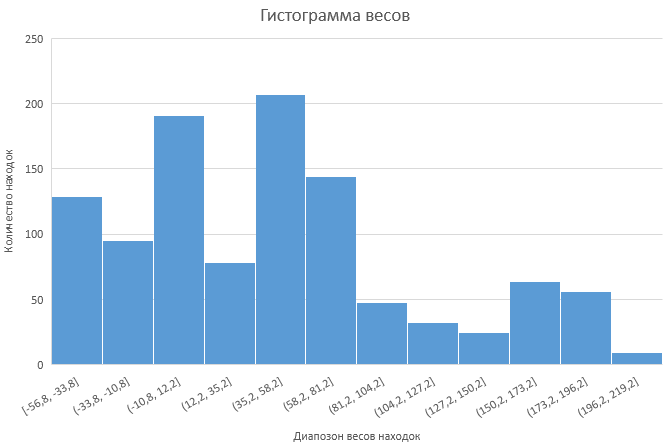
Так же была построена гистограмма весов (рис. ниже).
В качестве порога возьмем E-value = 2.3E-49. Это значение, при котором TPR становится равен 1. Возьмем эту точку по критерию max(TPR - FPR). FPR данной точки примерно равен 0.058. Построим таблицу 2 на 2 некоторых ключевых параметров профиля при данном пороге.
| На самом деле | принадлежит семейству | не пренадлежит семейстиву | всего |
|---|---|---|---|
| Выше порога по профилю | 7 | 62 | 69 |
| Ниже порога | 0 | 1009 | 1009 |
| Сумма | 7 | 1071 | 1078 |
Судя по полученным данным, профиль хорошо справляется с поиском подсемейства. Какое-то количество последовательностей могло иметь высокий score из-за того, что для построения дерева, а впоследствии и подсемейства мы брали не все последовательности, а только часть. Среди не выбранных последовательностей вполне могли быть те, которые хорошо вписываются в наше подсемейство.
© Maximov Vladislav, 2019.
