Построение дерева по нуклеотидным последовательностям
Получение последовательностей: расшифровки всех мнемоник были даны в первом практикуме,
поэтому находим папку с полным геномом нужного вида и берем последовательность 16S rRNA из файла с расширением .frn.
Все последовательности записаны в файл. Выравнивание проводилось
алгоритмом muscle программы MEGA 7. Строили дерево методом NJ.Полученное дерево
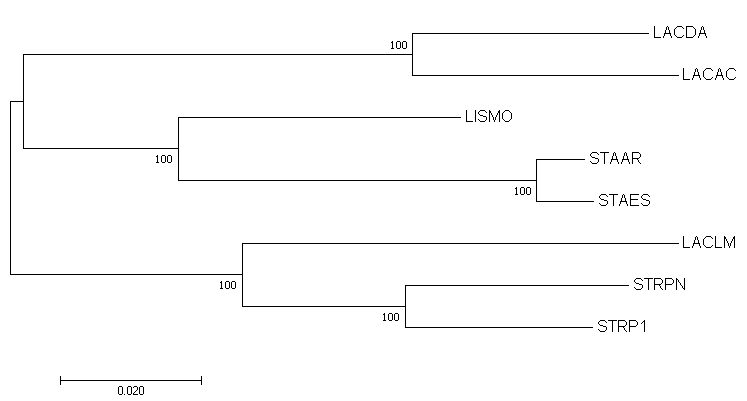

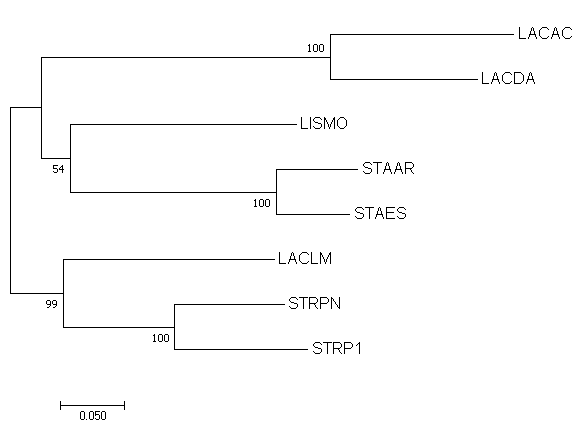
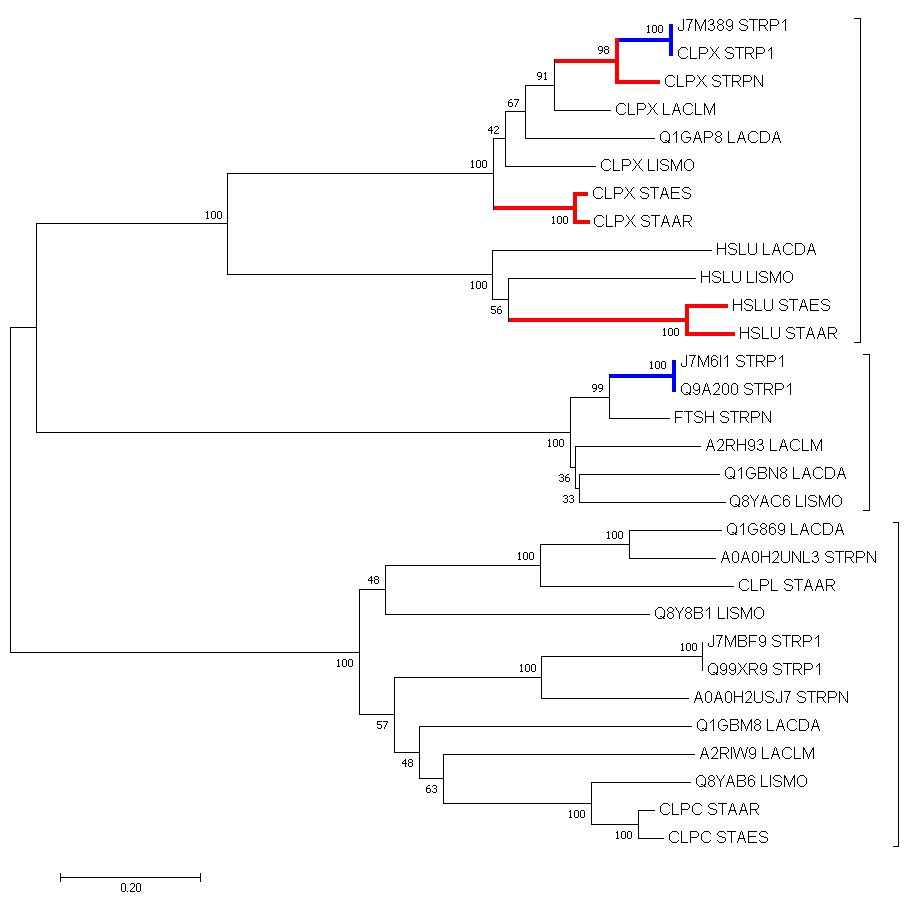
Следующий этап - построение и анализ дерева, содержащего паралоги. Для нахождения ортологов заданного белка
были скачаны протеомы наших бактерий из соответствующей директории, собраны в 1 файл, этот файл преобразован в
базу данных для локального бласта. Ну и последний шаг - запущен бласт с необходимыми параметрами. Его результаты в
файле. Всего было получено 33 гомолога. Отобранные последовательности записаны
в файл. Три последовательности плохо выравнивались с остальными и были
удалены из выравнивания. Полученное дерево изображено ниже. Некоторые примеры видообразования отражены красными ветвями,
паралогов - синими, группы, предположительно возникшие в результате дупликации, выделены скобками.
© Maximov Vladislav, 2019.
