 Всего ферментов с таким кодом — 440.
Всего ферментов с таким кодом — 440. Значение кода:
Все материалы взяты с сайта IUPAC.
Мне дан следуюший код фермента: EC 5.3.1.1.
EC 5. Isomerases (Изомеразы)
Эти ферменты катализируют изменения внутри одной молекулы.
EC 5.3 Intramolecular Oxidoreductases (Внутримолекулярные окислители-восстановители)
Эти ферменты вызывают окисление одной части молекулы с соответствующим восстановлением другой части.
EC 5.3.1 Interconverting Aldoses and Ketoses (Взаимное превращение альдоз и кетоз)
Сахарные изомеразы.
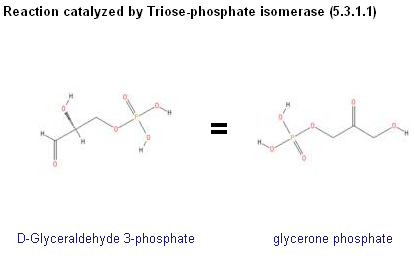
EC 5.3.1.1 triose-phosphate isomerase (Триозофосфат изомераза)
D-glyceraldehyde 3-phosphate = glycerone phosphate
Дополнительная информация о IUPAC
IUPAC был создан в 1919 году.
Международная комиссия по номенклатуре ферментов была создана в 1956 году.
Последние изменения/уточнения в номенклатуре ферментов были утверждены в 2006 году.
Характеристика фермента с помощью специализированного ресурса Brenda:


Активаторов не найдено.
Оптимум pH лежит от 6.5 для Oryctolagus cuniculus (Дикий кролик) до 7.2 для Secale cereale (Рожь).
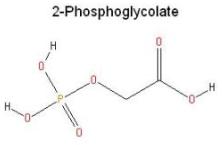
Ингибиторов у таких ферментов порядка сотни (93). Для Escherichia coli это 2-Phosphoglycolate.
Есть также и другие ингибиторы кишечной палочки, но для них показано отсутствие влияния на реакцию:
cyclo(Gly-Pro-Phe-Val-Phe-PSI[CS-NH]Phe)
cyclo[Trp-Phe-D-Pro-Phe-Phe-Lys(benzyloxycarbonyl)-]
S-Phenyl-p-toluenethiosulfonate
Дополнительная информация:
Строка запроса в SWISS-Prot SRS:
Query "([swissprot-ECNumber:5.3.1.1*] & (([swissprot-EntryName:*_Ecoli*] | [swissprot-EntryName:*_Human*]) | [swissprot-EntryName:*_Metja*])) "Для сравнения доменной структуры найденных белков использован вариант просмотра результата SW_InterProMatches, который позволяет быстро посмотреть на все мотивы в последовательностях.
Cравнивались только домены Pfam.
| Белок | Число доменов Pfam | Accession | Идентификатор Pfam | Положение в последовательности |
| TPIS_ECOLI | 1 | P0A858 | PF00121 | 4-246 |
| TPIS_HUMAN | 1 | P60174 | PF00121 | 6-244 |
| TPIS_METJA | 1 | Q58923 | PF00121 | 1-212 |
С помощью программы seqret были получены аминокислотные последовательности доменов: seqret sw:P0A858 -sask seqret sw:P60174 -sask seqret sw:Q58923 -sask
Попарные выравнивания были сделаны программой needle с помощью следующего скрипта: needle tpis_ecoli.fasta tpis_human.fasta ecoli_human.needle -gapopen 10.0 -gapextend 0.5 needle tpis_human.fasta tpis_metja.fasta human_metja.needle -gapopen 10.0 -gapextend 0.5 needle tpis_metja.fasta tpis_ecoli.fasta metja_ecoli.needle -gapopen 10.0 -gapextend 0.5
| Пара организмов | Identity | Выравнивание |
| Ecoli-Human | 45.3% | Посмотреть выравнивание |
| Human-Metja | 20.3% | Посмотреть выравнивание |
| Metja-Ecoli | 22.5% | Посмотреть выравнивание |
Комментарии: результаты выравнивания дают cтранный результат. Наиболее высокая идентичность у выравнивания белков человека
и кишечной палочки. Идентичность в 45,3% (считается, что функция сохраняется одинаковой у ферментов с Identity~40%).
В то же время идентичность белков кишечной палочки и археи, человека и археи соответственно равна 22,5 и 20,3%.
Нельзя также забывать, что на полученные значения немалый отпечаток накладывает степень "эволюционной дальности" организмов.
Что можно найти в ENZYME?
Набрав в запрос некоторый класс фермента получается выдача: