Предсказание генов эукариот
Задание 1. Предсказание генов X5 с помощью AUGUSTUS.
Для выполнения данного задания было необходимо выбрать один из контигов из сборки Х5 длиной от 20 кб до 100 kb. Для упрощения вездесущей проблемы выбора я запустила на сервере kodomo команду infoseq (с опциями -only -name -snucleotide1 -length) пакета EMBOSS, которая позволила мне вывести названия контигов и их длины (ссылка на файл с длинами контигов).
Среди контигов нужной длины я выбрала запись unplaced-676 длины 65387 п.н. и получила его нуклеотидную последовательность с помощью команды seqret пакета EMBOSS (ссылка на fasta-файл).
Предсказание генов с помощью AUGUSTUS.
Этап 1. Выбор организма, параметры генома которого будут взяты для предсказания генов.
Для того, чтобы предсказать гены в выбранном контиге с помощью ресурса AUGUSTUS, необходимо выбрать организм, параметры генома которого программа будет использовать для этого предсказания.
Вообще, ресурс позволяет решить эту проблему несколькими способами: можно либо провести "обучение" (training) программы на хороших примерах (используя гомологи хорошо изученных генов или анализируя транскриптом, если таковой имеется), либо использовать геном ближайшего организма, для которого уже было ранее выполнено предсказание генов. Эту и другую информацию, касающуюся предсказания генов с помощью ресурса AUGUSTUS, можно найти в prediction tutorial.
Мы выбрали второй путь, поэтому было необходимо найти наиболее близкий организм из предложенных ресурсом AUGUSTUS (ссылка на список организмов, которые предлагает AUGUSTUS).
Я справлялась с этой задачей при помощи BLAST (алгоритм blastx - транслирует входную нуклеотидную последовательность в 6 рамках и ищет сходства в белковом банке). Я выбрала именно этот алгоритм, так как он ищет непосредственный продукт гена, а по нуклеотидным последовательностям нет смысла искать из-за наличия интронов в генах эукариот.
Я запускала blastx, ограничив количество выводимых находок 50 (иначе он очень долго работает, а больше находок нам и не нужно) и задав область поиска только эукариотическими организмами, также я исключила (Exclude) поиск по моделям и пробам среды (Models (XM/XP) и Uncultured/environmental sample sequences).
На Рис. 1 можно увидеть выдачу blastx, где среди находок присутствуют несколько из имеющихся в базе AUGUSTUS: много находок рода Candida (красное подчеркивание) (в частности, Candida albicans) и одна находка рода Rhizopus (синее подчеркивание).
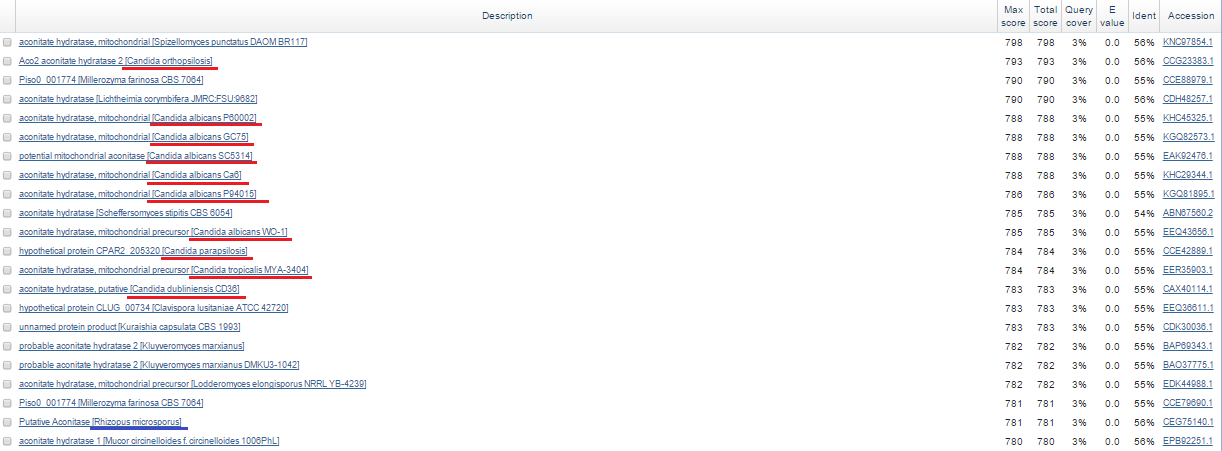
На основе этих данных я выбрала организм Candida albicans для предсказания генов в последовательности моего контига.
Этап 2. Обзор результатов работы ресурса AUGUSTUS.
Ссылка на страницу с описанием статуса задания: страница.
Результатом работы ресурса является архив predictions.tar.gz, который содержит следующие файлы:
- augustus.aa с аминокислотными последовательностями белков (в формате fasta) - продуктов каждого из предсказанных генов
- augustus.cdsexons с нуклеотидными последовательностями предсказанных экзонов (по отдельности для каждого экзона - в формате fasta)
- augustus.codingseq с кодирующими нуклеотидными последовательностями предсказанных генов целиком (все экзоны одного гена вместе - в формате fasta)
- augustus.gbrowse с информацией (координаты в геноме, прямая или обратная цепь) о структурных элементах пре-мРНК (кодирующая последовательность, интрон, старт- и стоп-кодоны, инициаторная и терминальная последовательности), предшествующей каждому из предсказанных генов (предсказание геномного браузера GBrowse)
- augustus.gtf с предсказанием генов в формате .gtf
- augustus.gff - роскошный файл со всей вышеперечисленной информацией вместе (формат .gff)
Этап 3. Проверка предсказания с помощью BLAST.
Программа предсказала 18 белок-кодирующих генов (18 последовательностей предсказанных белков в файле augustus.aa.
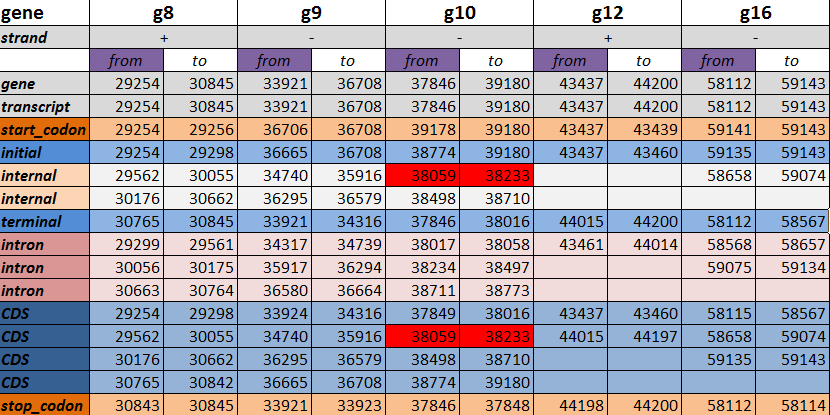
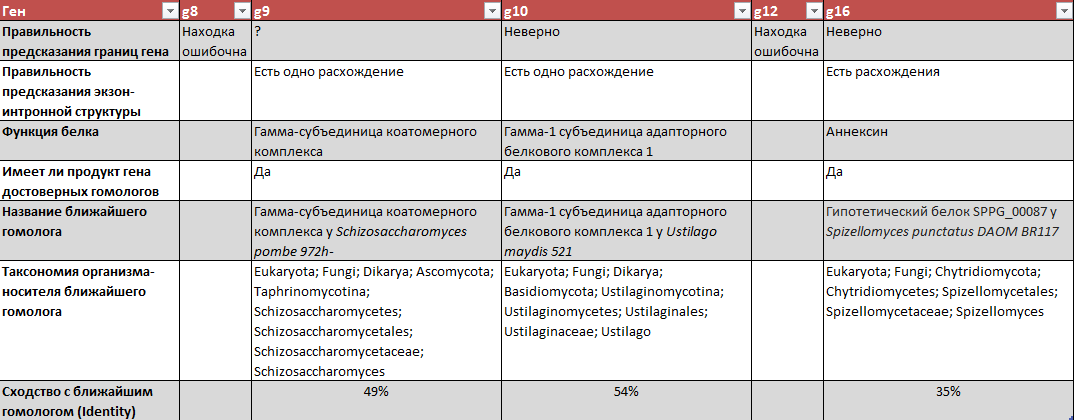
Для проверки экзон-интронной структуры я выбрала 5 генов: g8, g9, g10, g12, g16.
Данные о предсказанной экзон-интронной структуре я брала из файла augustus.gff, так как там они представлены в наиболее приемлемом для использования виде. Но для более удобного анализа результатов предсказания я продублировала эти данные для пяти выбранных белков в Таблице 1 (строки, касающиеся разных структурных элементов гена, выделены разными цветами; красным цветом выделены ячейки, которые по-особому выделены в файле .gff, и мне пока не удалось выяснить причину этого).
Таблица 1. Предсказание экзон-интронной структуры генов g8, g9, g10, g12, g16 с помощью ресурса AUGUSTUS.
Я проверяла экзон-интронную структуру с помощью алгоритма blastp (ищет входную последовательность белка в белковом банке), для которого брала последовательности белковых продуктов выбранных генов из файла augustus.aa.
Для каждой входной последовательности я запускала blastp отдельно по 2 банкам: всех последовательностей (Non-redundant protein sequences) и Swiss-Prot (белки, проверенные хотя бы по гомологии), а также я ограничивала поиск таксоном Fungi либо Eukaryota (в случаях с малым количеством или плохим качеством находок). E-value я ограничивала 1, кроме нескольких случаев, где совсем не было подходящих находок.
А теперь обо всем по порядку.
- g9
Первым делом я решила проверить белок с самой длинной последовательностью - продукт предсказанного гена g9.
На Рис. 2а представлена выдача blastp по банку nr, область поиска ограничена таксоном Fungi. Видно, что найдены сходства не с целой последовательностью, а с отдельными участками. Это возможно как по причине неверного предсказания, так и просто ввиду вариативности краевых участков в сравнении с консервативными доменами, поэтому сделать какой-либо вывод о правильности предсказания границ гена считаю невозможным.
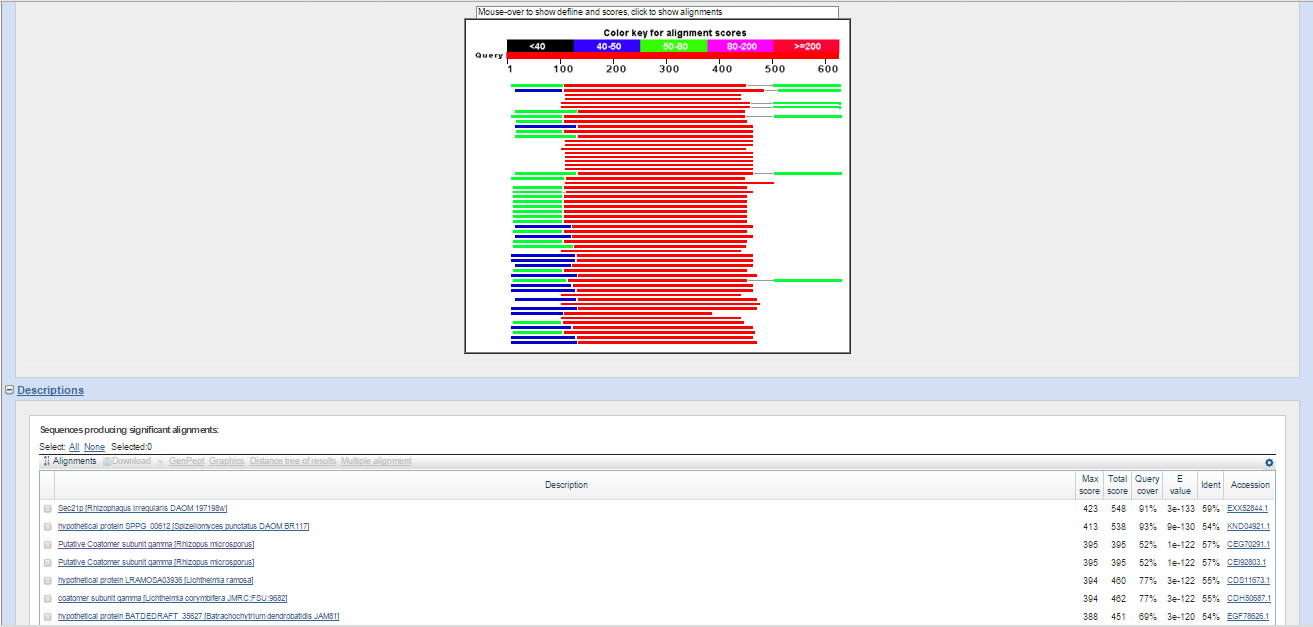
Рис. 2a. Выдача blastp (поиск по банку nr, таксону Fungi) для последовательности белка (продукта предсказанного гена g9).
Присутствует один крупный, сравнительно консервативный домен (красные полосы): могло быть и лучше, от организма к организму варьирует даже длина этого участка. Это заставляет предположить, что сам домен был затронут эволюционными изменениями, что осложняет принятие решения по поводу правильности предсказания границ гена и экзон-интронной структуры. Находки, сходные с участками по краям от "консервативного" домена, имеют очень низкий Score и присутствуют не у всех организмов, у которых был найден "консервативный" домен. Скорее всего, это, во-первых, неконсервативные участки белка, а, во-вторых, там вообще, вероятно, происходили индели, и, в-третьих, функции этих участков не определены.
Чтобы заручиться поддержкой более подтвержденных данных, я запустила поиск по базе Swiss-Prot и домену Eukaryota, и среди находок целых две принадлежат Fungi (отмечены черными звездочками на Рис. 2b).
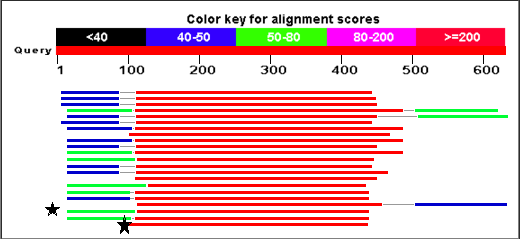
Рис. 2b. Выдача blastp (поиск по банку Swiss-Prot и домену Eukaryota) для последовательности белка (продукта предсказанного гена g9). Черными звездочками отмечены находки из организмов, принадлежащих Fungi.
Одна из "грибных" находок принадлежит организму Schizosaccharomyces pombe 972h-. Эта находка ценна для нас тем, что у этого организма данный продукт подтвержден экспериментально на уровне белка, и определена функция белка (см. красные рамки на Рис. 2с - фрагмент страницы этого белка в Swiss-Prot).
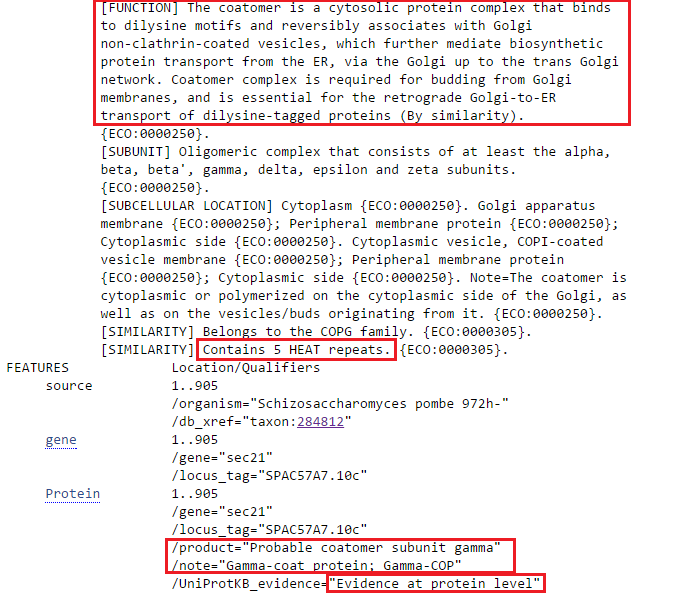
Рис. 2с. Фрагмент страницы в Swiss-Prot белка-продукта предсказанного гена g9. Красными рамками выделены функция и название белка, подтвержденность на белковом уровне и структурные особенности.
Этот белок является гамма-субъединицей цитозольного коатомерного комплекса (связывается с мотивами, содержащими два лизина, и обратимо ассоциируется с везикулами аппарата Гольджи, не покрытыми клатрином), необходимого для транспорта от аппарата Гольджи в ЭПР белков, содержащих дилизиновый мотив. Строение и функции коатомерного комплекса обозначены на Рис. 2d.
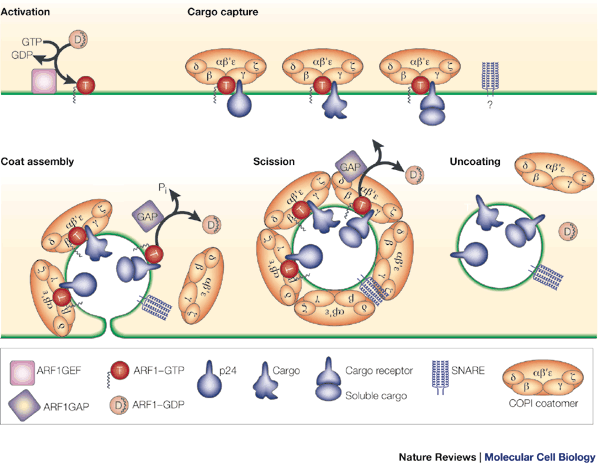
Рис. 2d. Строение и функции коатомерного комплекса.
В выравнивании находки с входной последовательностью предсказанного белка (Рис. 2е), можно увидеть два участка: большой (соответствует "консервативному" домену) и поменьше (не буду его рассматривать, так как он расположен в вариантивной области).
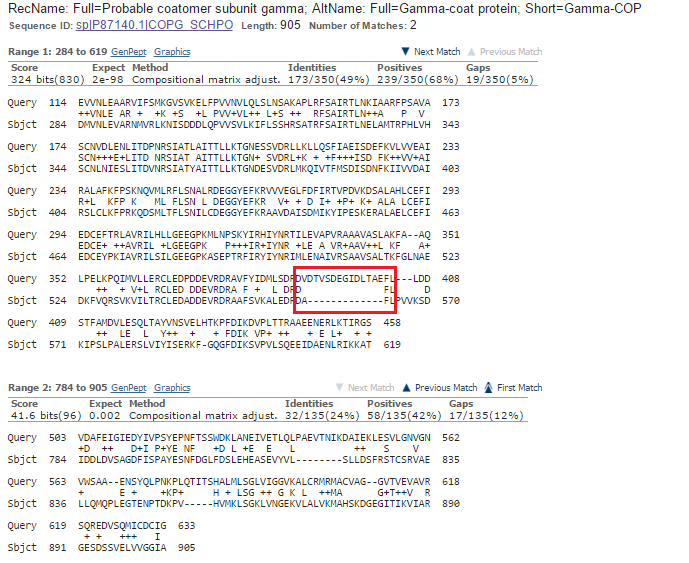
Рис. 2е. Выравнивание продукта гена g9 с гамма-субъединицей коатомерного комплекса Schizosaccharomyces pombe 972h-.
(Катарсис уже близок)
В последовательности большого участка находки можно видеть гэп приблизительно с 548 по 562 позиции (выделен красной рамкой на Рис. 2е). Примерно в том же месте был гэп почти у всех находок по банку nr. С высокой вероятностью этот гэп можно расценить как неправильное определение границы экзона ресурсом AUGUSTUS (часть интрона была рассмотрена в качестве экзона и транслирована, поэтому с ней стабильно нет сходства во всех последовательностях).
Вывод: есть ошибки в определении экзон-интронной структуры.
P.S. Интересная особенность: в последовательности белка присутствуют 5 повторов по 37-40 аминокислот, и входная последовательность выравнивается именно с участком, содержащим эти повторы. Честно говоря, не знаю, как истолковать это наблюдение, но очень интересно.
- g8
Затем я решила проверить еще один довольно крупный потенциальный белок - продукт гена g8.
Результат оказался неожиданным (Рис. 3): при поиске по банку nr была обнаружена всего одна находка с E-value=36.
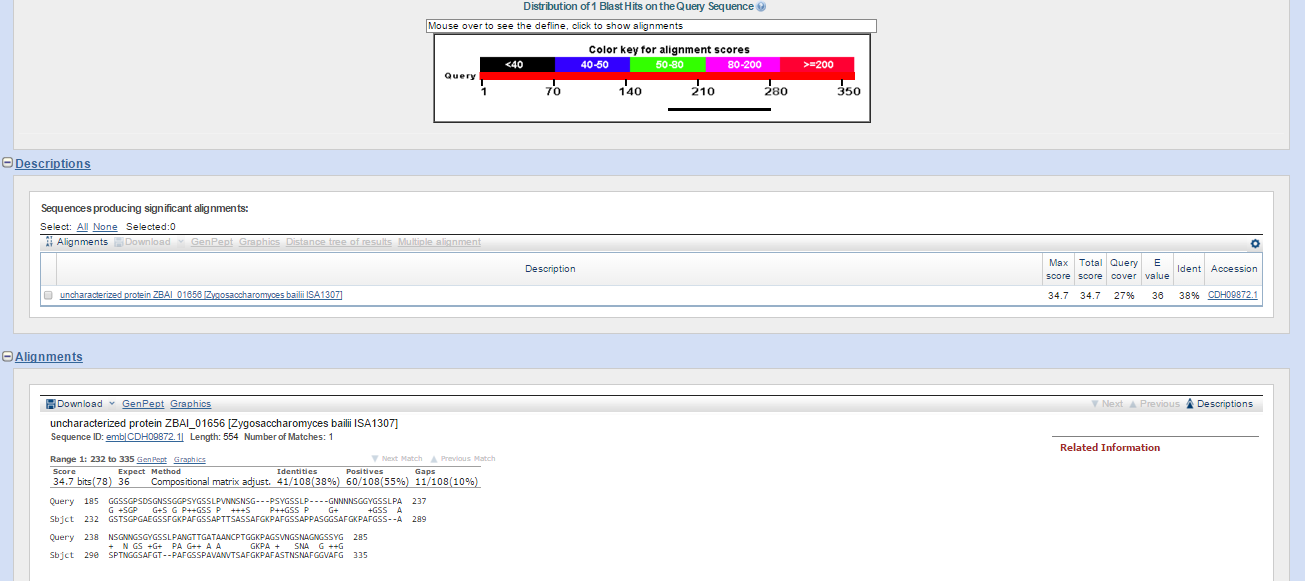
Рис. 3. Выдача blastp (поиск по банку nr, таксону Eukaryota) для последовательности продукта предсказанного гена g8.
Вывод: предсказание ошибочно.
- g10
Ситуация с продуктом этого гена очень похожа на g9, но намного лучше: на Рис. 4а (выдача blastp по банку nr, Fungi) снова виден более крупный, на этот раз по-настоящему консервативный, домен (длина и локализация в белке-находке совпадают для всех находок) и концевой участок, который присутствует не у всех находок (но среди присутствующих у многих совпадают длина и локализация в белке).
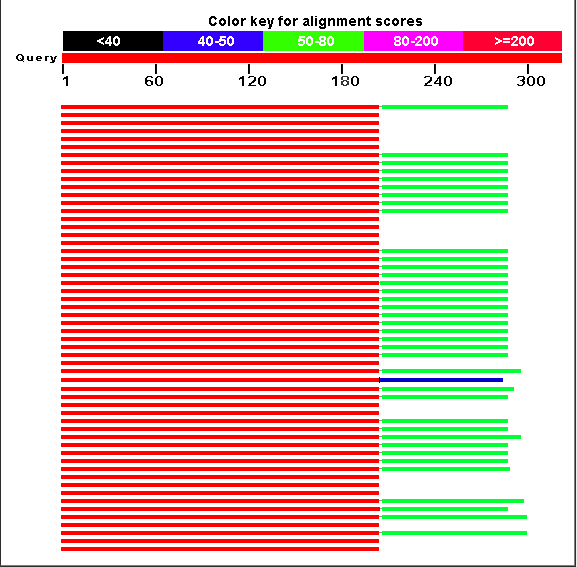
Рис. 4а. Выдача blastp (поиск по банку nr, таксону Fungi) для последовательности продукта предсказанного гена g10.
Затем я снова провела поиск по банку Swiss-Prot, где среди находок получила одну, принадлежащую Fungi: Ustilago maydis 521. Несмотря на то, что этот белок был подтвержден только на уровне гомологии, он позволяет узнать функцию предсказанного гена (Рис. 4b).
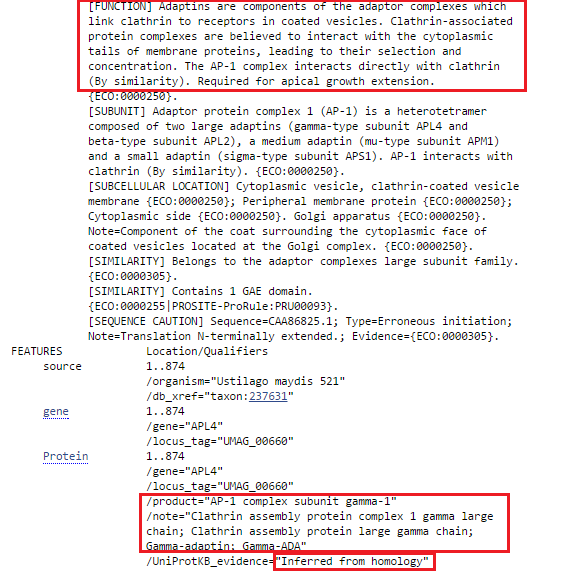
Рис. 4b. Фрагмент страницы в Swiss-Prot белка-продукта предсказанного гена g10. Красными рамками выделены функция и название белка и подтвержденность на уровне гомологии.
Итак, это гамма-1 субъединица (большая цепь) адапторного белкового комплекса 1, который напрямую связывает клатрин с рецепторами покрытых везикул. Белки, ассоциированные с клатрином, взаимодействуют с цитоплазматическими хвостами мембранных белков, приводя к их отбору и концентрации. Играет роль в продолжении апикального роста организма.
На Рис. 4с представлено выравнивание входной последовательности с вышеупомянутой находкой. Во-первых, 1 остаток входной последовательности соответствует не 1 остатку находки, следовательно, белок представлен не целиком, и границы гена предсказаны неверно. Во-вторых, во всех находках присутствует одинаковый по длине и положению гэп (красная рамка), который снова свидетельствует об ошибке в определении экзон-интронной структуры.
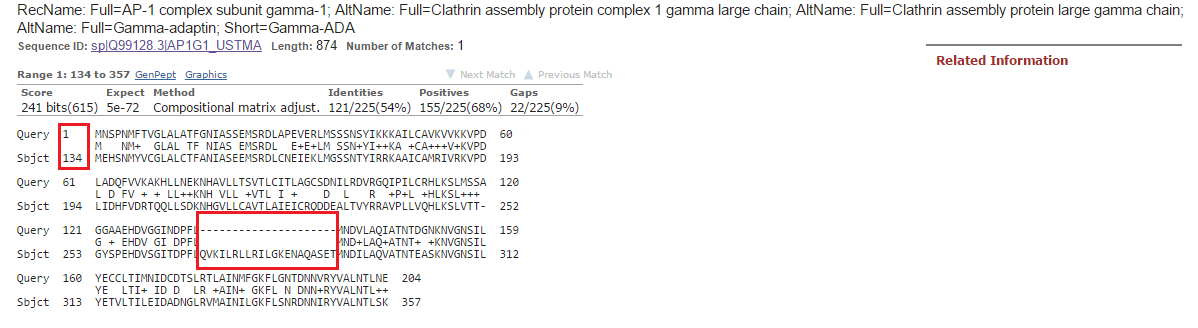
Рис. 4с. Выравнивание продукта гена g10 с гамма-1 субъединицей адапторного белкового комплекса 1 Ustilago maydis 521.
Вывод: есть ошибки в предсказании границ гена и экзон-интронной структуры.
- g12
Продукт гена g12 самый короткий среди предсказанных, что уже не предвещало ничего хорошего. И действительно (Рис. 5), в выдаче blastp всего 6 находок с E-value>=25, что уже не позволяет им доверять.
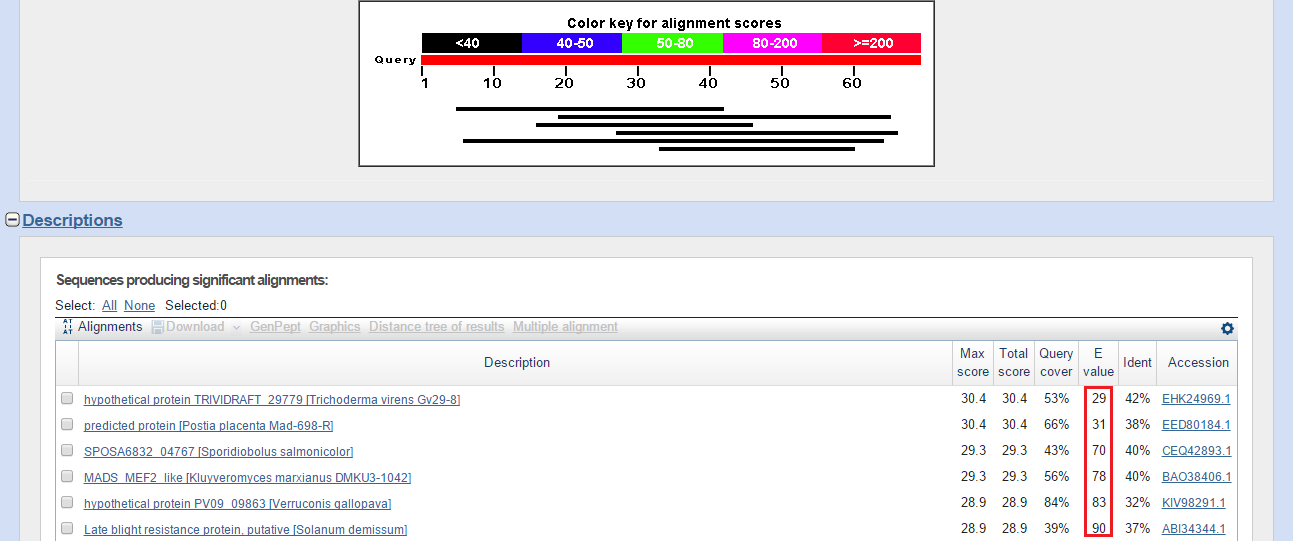
Рис. 5. Выдача blastp (поиск по банку nr, таксону Eukaryota) для последовательности продукта предсказанного гена g12.
Вывод: предсказание ошибочно.
- g16
На сладкое самая неоднозначная проверка: на Рис. 6а видно, что у находок очень хороший query cover, но недостаточно хороший Score. Возможно, это из-за того, что во всех последовательностях снова присутствует 1 гэп (красная рамка на Рис. 6b), который на этот раз довольно длинный (здесь сразу можно сказать об ошибочном определении экзон-интронной структуры). Также начало входной последовательности снова не совпадает с началом находки, то есть границы гена тоже определены неправильно.

Рис. 6а. Выдача blastp (поиск по банку nr, таксону Fungi) для последовательности продукта предсказанного гена g16.
На Рис. 6b представлено выравнивание с лучшей находкой запуска по банку nr.
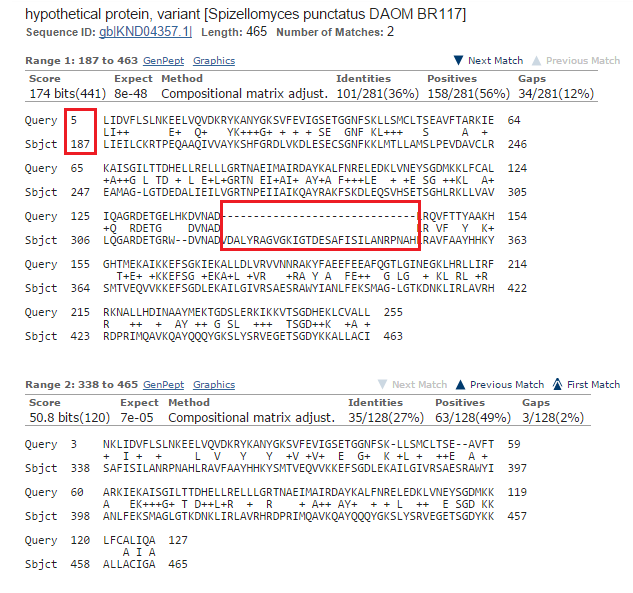
Рис. 6b. Выравнивание продукта гена g16 с лучшей находкой запуска по банку nr.
Большое количество пенициллов среди находок (красное подчеркивание на Рис. 6с) пытаются убедить меня, что продукт гена похож на аннексин - белок связывающий кальций и фосфолипиды.
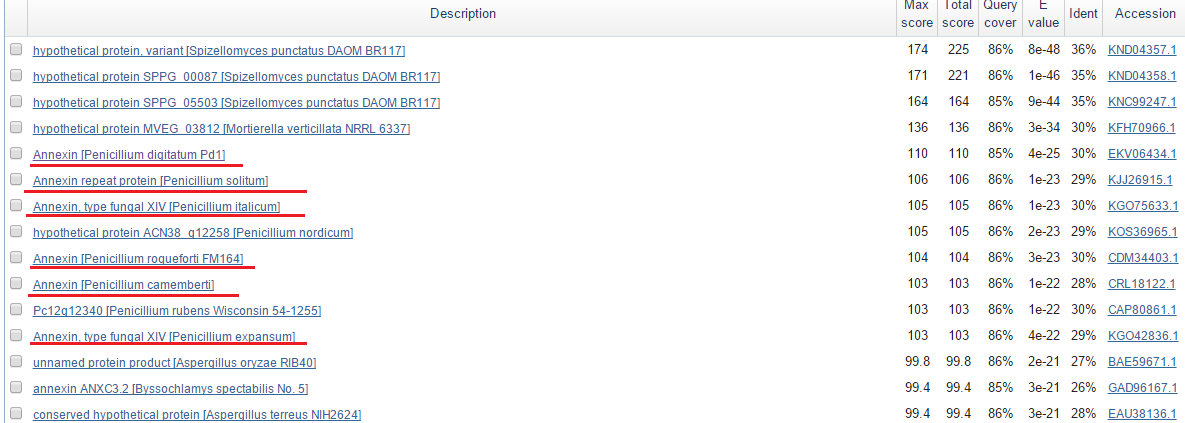
Рис. 6с. Выдача blastp (поиск по банку nr, таксону Fungi) для последовательности продукта предсказанного гена g16.
Поиск по банку Swiss-Prot не выдал ни одной "грибной" находки, а лучшей является находка из организма человека (Рис. 6d), которую язык не поворачивается назвать ближайшим достоверным гомологом. Но, по крайней мере, эта находка подтверждает, что продукт гена - аннексин.
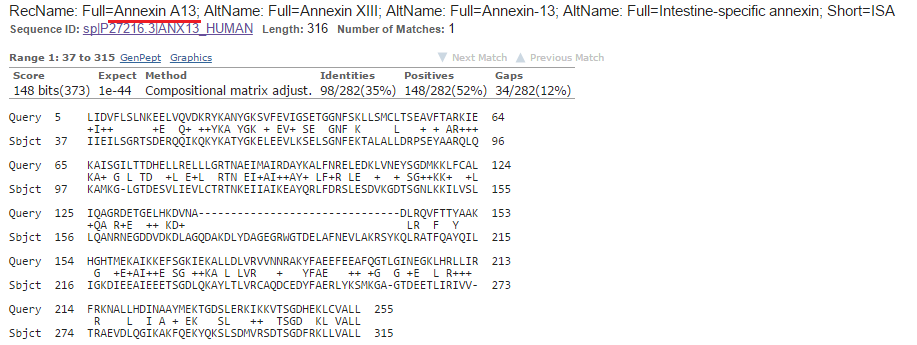
Рис. 6d. Выравнивание продукта гена g16 с лучшей находкой запуска по банку Swiss-Prot.
В создавшейся ситуации будем считать ближайшим гомологом лучшую находку при поиске по банку nr: Spizellomyces punctatus DAOM BR117 (у которой, кстати, функция белка
Вывод: находка правильная, но границы гена и экзон-интронная структура предсказаны неверно.
В Таблице 2 представлены результаты проверки предсказания для 5 выбранных генов.
Таблица 2. Результаты проверки предсказания экзон-интронной структуры генов g8, g9, g10, g12, g16 с помощью ресурса AUGUSTUS.
Материалы Таблиц 1 и 2 можно также получить в файле Excel.
Задание 2. Сравнить аннотацию RefSeq и AUGUSTUS одного гена человека.
Для выполнения данного задания я выбрала ген HDAC1 (Gene ID: 3065 в NCBI), кодирующий белок гистодеацетилазу 1, который входит в состав комплекса, деацетилирующего гистоны, что играет ключевую роль в регуляции экспрессии генов. Также взаимодействует с белком-супрессором ретинобластомы, осуществляя контроль за пролиферацией и дифференциацией клеток. Вместе с ассоциированным с метастазами белком-2 он деацетилирует p53, модулируя его влияние на рост клеток и апоптоз.
Координаты гена: хромосома 1, от 32292107 до 32333628 п.н. (по версии NCBI; от 32292086 до 32333635 в UCSC Genome Browser), прямая цепь.
Длина: 41522 п.н. (NCBI) (41550 п.н. в UCSC Genome Browser).
Данное задание выполнялось с использованием ресурса UCSC Genome Browser. Там я выбрала последнюю доступную сборку генома человека (hg38) и нашла интересующий меня ген с помощью поиска в Genome Browser.
Далее я оставила только три трека: base position, RefSeq и AUGUSTUS. На Рис. 7 приведен скриншот окна браузера с двумя аннотациями гена HDAC1.
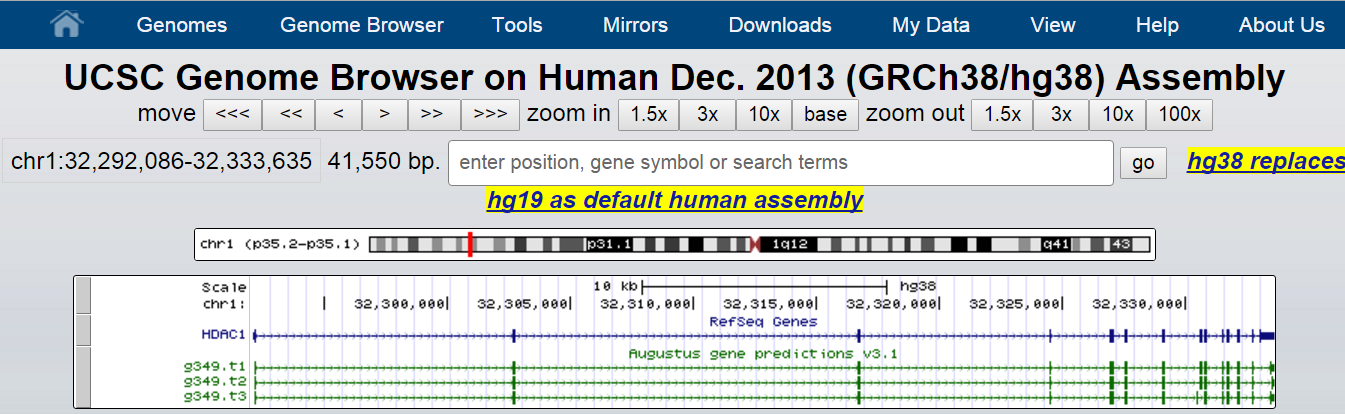
Затем я по отдельности получила предсказания экзон-интронной структуры, выполненные RefSeq и AUGUSTUS, в виде таблиц (было достаточно получить колонки с координатами экзонов и кодирующих последовательностей).
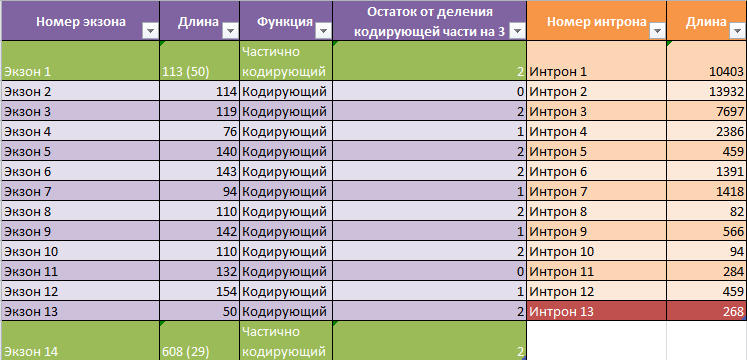
В Таблице 3 представлена информация об экзон-интронной структуре гена, в соответствии с аннотацией RefSeq: всего предсказано 14 экзонов и 13 интронов, координаты кодирующей части (CDS) - от 32292169 до 32333044 п.н. (лист RefSeq в файле Excel).
Нужно обратить внимание на то, что первый и последний экзон являются частично кодирующими, так как в них присутствуют 5' и 3'-нетранслируемые области, соответственно. Поэтому для вычисления остатка от деления длины кодирующего участка на 3 нужно было использовать полученные координаты CDS (для остальных экзонов я воспользовалась функцией вычисления остатка в Excel). Для этих экзонов в скобках указана длина кодирующей части.
В Таблице 3 цветом хаки выделены строки, относящиеся к первому и последнему экзонам. Последний интрон выделен бордовым цветом (его длина отличается у разных предсказаний).
Таблица 3. Экзон-интронная структура гена HDAC1 (аннотация RefSeq).
AUGUSTUS делает 3 разных предсказания. Первый вариант является имеет наиболее длинные экзоны (лист AUGUSTUS1 в файле Excel) (координаты кодирующей части (CDS) - от 32292169 до 32333493), так как во втором варианте (координаты кодирующей части (CDS) - от 32292169 до 32333560) экзон №14 намного короче (лист AUGUSTUS2), а в третьем (координаты кодирующей части (CDS) - от 32292169 до 32333493) короче экзон №8 (лист AUGUSTUS3).
В Таблицах 4а-с красным цветом выделены отличия 2 и 1 вариантов, синим - 1 и 3, хаки - краевые экзоны, вишневым - последний интрон.
Таблица 4а. Экзон-интронная структура гена HDAC1 (аннотация AUGUSTUS, вариант 1).
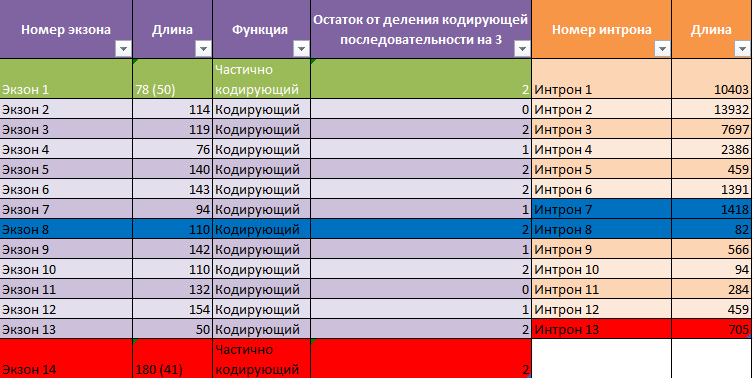
Таблица 4b. Экзон-интронная структура гена HDAC1 (аннотация AUGUSTUS, вариант 2).
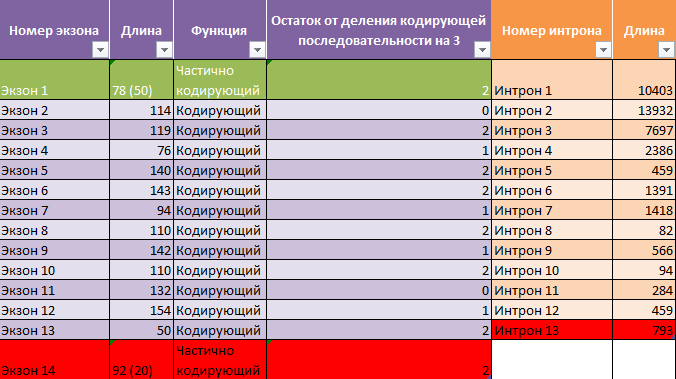
Таблица 4c. Экзон-интронная структура гена HDAC1 (аннотация AUGUSTUS, вариант 3).
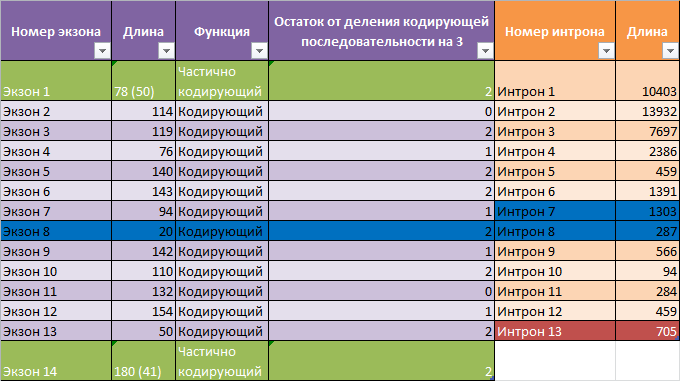
Главные отличия между аннотациями RefSeq и AUGUSTUS касаются первого и последнего экзонов и последнего интрона (все длины в п.н.):
- Первый экзон длиннее в аннотации RefSeq (113 против 78), при этом кодирующая часть одинаковая (50); первые интроны одинаковы по длине (10403); из всего этого следует, что RefSeq определил более длинную 5'-нетранслируемую область.
- Длина последнего экзона в предсказании RefSeq (608 с кодирующей частью длиной 29) отличается о предсказанных AUGUSTUS: 180 с кодирующей частью длиной 41 в 1 и 3 вариантах и и 92 (20) во 2 варианте. Длины интронов тоже отличаются: 268 (RefSeq), 793 (2 вариант AUGUSTUS) и 705 (1 и 3 вариант AUGUSTUS). Несомненно, в аннотации RefSeq снова 3'-натранслируемая область определена намного более длинной, но при этом сам экзон расположен намного раньше, чем в аннотациях AUGUSTUS. По всей видимости, определение последнего экзона является самым неоднозначным, так как различия в аннотациях крайне велики.