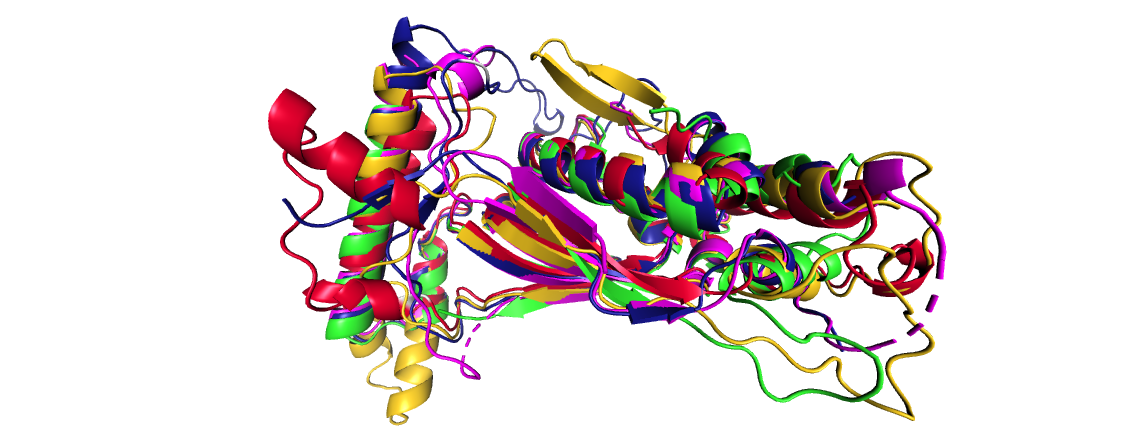
Совмещение структур гомологов вирусной тимидилатсинтазы ThyX
Для выполнения этого задания снова использовался белок вирусная флавин-зависимая тимидилатсинтаза ThyX из вируса Paramecium bursaria Chlorella virus 1 (PDB ID: 2CFA). С помощью сервиса PDBeFold были найдены структурные гомологи этого белка. Поиск выполнялся по цепи А тимидилатсинтазы (всего структура имеет две цепи). Для дальнейшей работы было выбрано четыре разных белка-гомолога с RMSD в диапазоне от 0.8 до 2.5 и длиной выравнивания более 50% от длины Query. В Таблице 1 перечислены отобранные гомологи.PDB ID:chain |
Организм |
RMSD |
Длина выравнивания, % от длины Query |
4gtd:D |
Thermotoga maritima |
1.58 |
89 |
4p5a:C |
Streptomyces cacaoi |
1.51 |
89 |
2af6:B |
Mycobacterium tuberculosis |
1.99 |
90 |
3ah5:E |
Helicobacter pilori |
2.07 |
83 |
На Рис. 1 изображено структурное совмещение цепи А исходного белка и его четырех структурных гомологов. Структурное совмещение было выполнено с помощью сервиса PDBeFold (результат в формате Rasmol, в формате PDB). Структурное выравнивание можно посмотреть в формате fasta. Можно заметить, что структуры совместились не слишком хорошо, так как несовпадения есть не только в области свободных петель. Скорее всего, это связано с дефектами расшифровки структур.
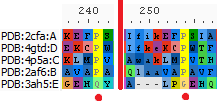
Далее требовалось сравнить структурное выравнивание (Рис. 2) и выравнивание по последовательностям (Рис. 3). Последнее я строила с помощью алгоритма Muscle.


Выравнивания различаются между собой. Однако продемонстрировать конкретное различие было довольно трудно, так как, во-первых, длины белков в fasta-файле со структурным выравниванием и в структурах, подгружаемых в PyMOL, различаются. Это сильно осложнило поиск "интересующих мест" на самих структурах. Далее, большинство несовпадений находятся в начале или конце выравниваний, где структуры слишком различаются, поэтому нельзя сделать вывод о том, какое выравнивание более правильное. Еще один осложняющий фактор - наличие нерасшифрованных участков в структурах (особенно в исходной), они как раз часто оказываются в местах несовпадений выравниваний.
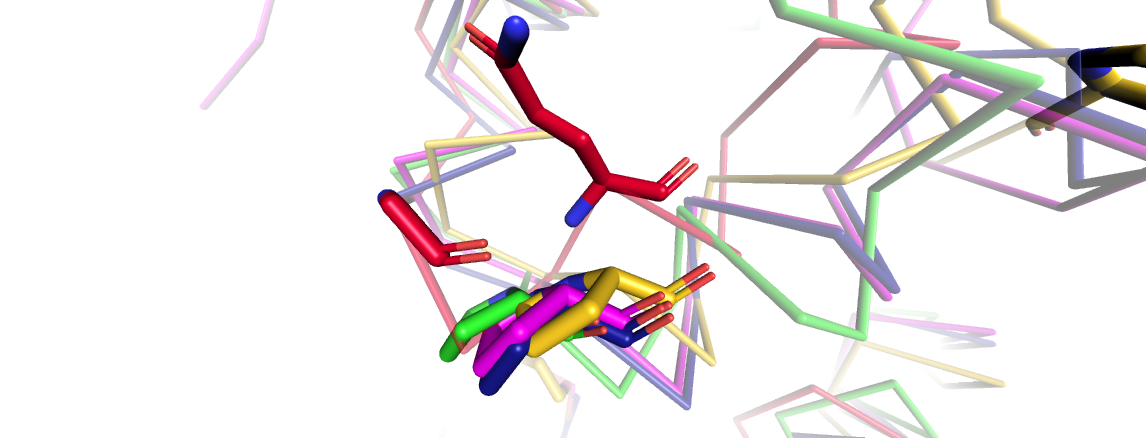
На Рис. 4 показан пример несоответствия между выравниваниями (до красной черты - выравнивание последовательностей, позиция 241; после черты - структурное, позиция 254). Для того чтобы выяснить, какое выравнивание ближе отражает действительность, я отобразила интересующие остатки на структурах (Рис. 5). Видно, что 4 пролина совмещены хорошо, а вот глицин (кандидат от партии структурного выравнивания) и глутамин (надежда выравнивания последовательностей) цепи 3ah5:E (остатки красного цвета) одинаково далеки от пролинов. По всей видимости, цепь 3ah5:E в этом месте отличается от общего структурного паттерна, поэтому ни один из двух способов с выравниванием не справился.
Построение совмещения по заданному выравниванию
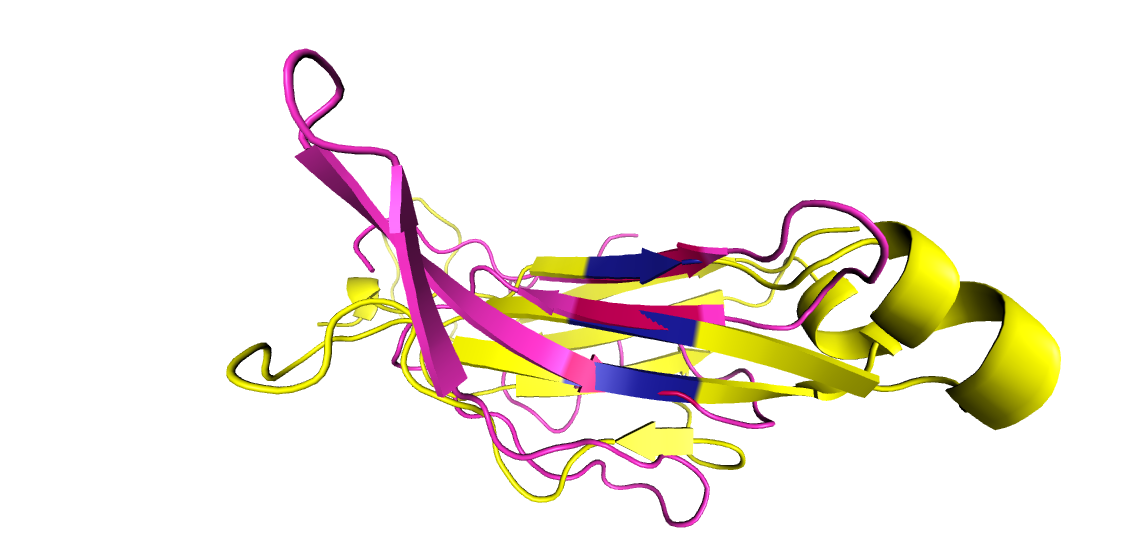
Для выполнения этого задания я использовала одну из структур константного домена T-клеточного рецептора из цепи альфа (PDB ID: 1MI5, участок цепи D: 118 - 206) и одну — из цепи бета (PDB ID: 1FYT, участок цепи Е: 119 - 246).
С помощью сервиса SheeP были получены карты бета-листов для цепи альфа (Рис. 6а) и цепи бета (Рис. 6b). Для того чтобы ориентация бета-листов совпадала, к карте для цепи бета была применена команда flip columns. Из двух бета-листов на карте для цепи бета верхний (Рис. 6b) совпадает с листом на карте для цепи альфа (Рис. 6а).
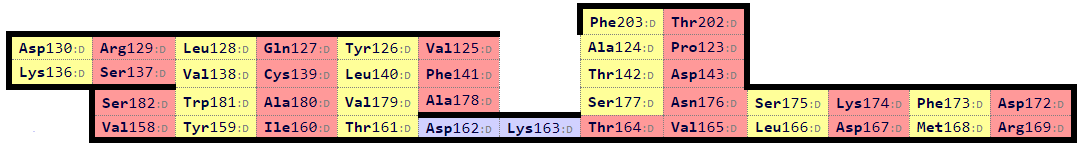

Затем я построила выравнивание этих структур (файл с совмещением). Для этого я нашла в каждой из структур консервативные цистеины, образующие дисульфидные мостики: Cys139 в цепи альфа и Cys148 в цепи бета. Выравнивание структур проводилось по этим цистеинам и окружающим их остаткам с помощью следующих команд:
select alpha, 1mi5 and chain D and resi 126-128+138-140+179-181
select beta, 1fyt and chain D and resi 129-131+147-149+194-196
pair_fit alpha, beta
Совмещение структур представлено на Рис. 7. Альфа-цепь покрашена сиреневым цветом, бета - желтым. Остатки, по которым производилось совмещение, выделены красным и синим цветами на цепях альфа и бета, соответственно. Видно, что структуры совпадают довольно неплохо, общий ход полипептидной цепи одинаков для большинства петель (говоря о бета-цепи, подразумевается только тот бета-лист, который выравнивается с листом на альфа-цепи). Однако в правой части Рис. 7 видно, что в одном месте там, где в альфа-цепи идет свободная петля, в бета-цепи расположена альфа-спираль. Также в альфа-цепи присутствует небольшой бета-тяж, не имеющий совпадения в бета-цепи. Эти различия не позволяют говорить об идентичности топологий у рассматриваемых участков.