| Главная страница | Семестры | О себе | Ссылки |
Cоздание изображения белка LigA с помощью Jmol
Jmol - бесплатная программа, которая позволяет визуализировать трехмерную структуру какой-либо биоиолекулы по файлу в формате pdb, содержащему информацию о координатах составляющих молекулу атомов и их химической природе (получается эта информация методом рентгеноструктурного анализа). Jmol cпособен отображать молекулу и отдельные группы атомов в ее составе в разных режимах, что очень удобно для анализа ее химического строения и биогических функций.
Идентификатор PDB ДНК лигазы - 4GLW. Соответственно, все последующие изображения были получены после открывания в Jmol скачанного отсюда файла.
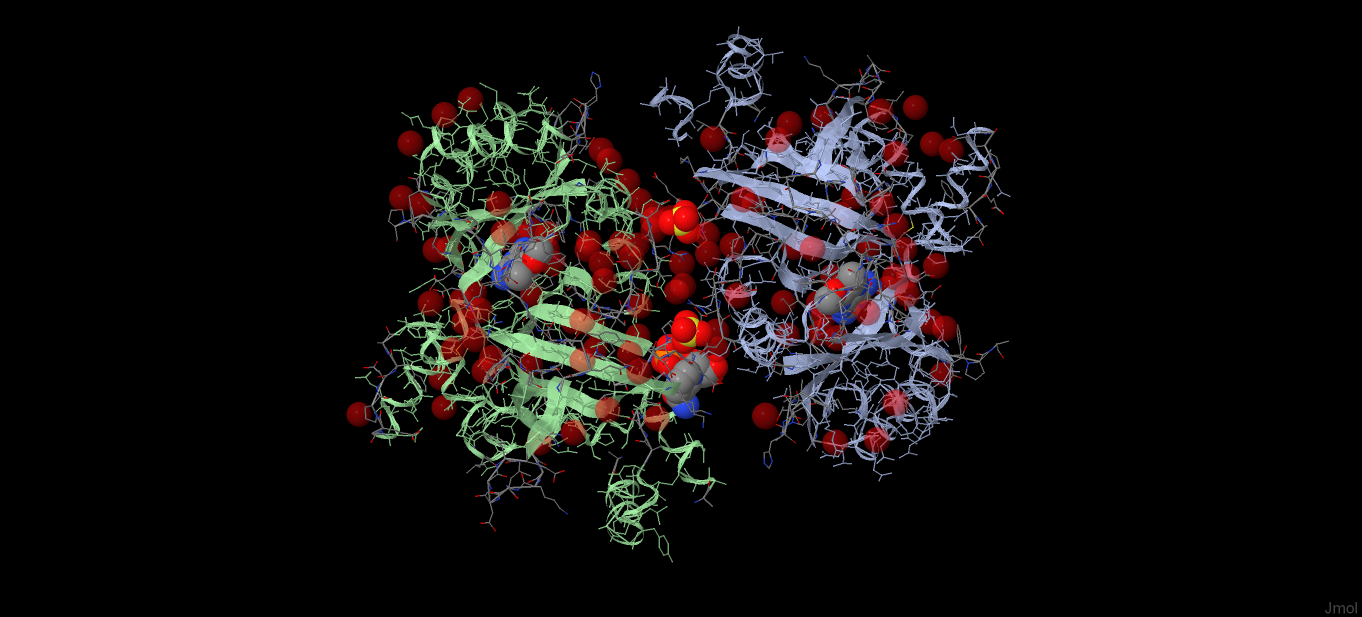
Рис. 1. Схема белка LigA, полученная с помощью Java-программы Jmol
Как видно из рисунка 1, белок LigA является гомомерным димером (состоит из двух одинаковых цепей). α-cпирали (узкие ленты, ribbons) и β-складки (широкие ленты, ribbons) цепи А окрашены светло-лиловым цветом, а цепи В - салатовым. Атомы белка, не включенные в спирали и складки, окрашены в соответствии с химической природой (cpk). Полупрозрачные шарики (cpk) - это молекулы воды. Атомы, изображенные непрозрачными шариками, образуют 5 лигандов. Радиусы шариков соответствуют Ван-дер-Ваальсовым радиусам атомов.
Скрипт, который позволяет воссоздать рисунок 1 в Jmol:
load 4GLW.pdb
background black
select all
cartoons off
ribbons off
backbone off
cpk off
wireframe 10
color cpk
select *.CA
backbone 35
select helix
ribbons 75
select sheet
ribbons 300
select helix or sheet
color chain
color translucent 0.2
select water or ligand
cpk
select water
color translucent 0.6
rotate Y 82
rotate X 312
rotate Z 150
rotate X 65
rotate Y 335
По этой ссылке можно скачать pdb-файл с информацией о трехмерной структуре LigA.
Информация о белке, составе молекул, методе решения пространственной структуры
В pdb-файле с информацией о третичной структуре белка содержится, кроме того, некоторая дополнительная информация. Ниже представлены некоторые выжимки из нее.
TITLE DNA LIGASE A IN COMPLEX WITH INHIBITOR
COMPND MOL_ID: 1;
COMPND 2 MOLECULE: DNA LIGASE;
COMPND 3 CHAIN: A, B;
COMPND 4 SYNONYM: POLYDEOXYRIBONUCLEOTIDE SYNTHASE [NAD(+)];
COMPND 5 EC: 6.5.1.2;
COMPND 6 ENGINEERED: YES
SOURCE 2 ORGANISM_SCIENTIFIC: STREPTOCOCCUS PNEUMONIAE;
EXPDTA X-RAY DIFFRACTION
REMARK 2 RESOLUTION. 2.00 ANGSTROMS.
HETNAM SO4 SULFATE ION
HETNAM 0XT 7-METHOXY-6-METHYLPTERIDINE-2,4-DIAMINE
HETNAM NMN BETA-NICOTINAMIDE RIBOSE MONOPHOSPHATE
HETSYN NMN NICOTINAMIDE MONONUCLEOTIDE
FORMUL 3 SO4 2(O4 S 2-)
FORMUL 4 0XT 2(C8 H10 N6 O)
FORMUL 7 NMN C11 H16 N2 O8 P 1+
FORMUL 8 HOH *110(H2 O)
Примерный перевод
Название: ДНК лигаза в комплексе с ингибитором
Компоненты-молекулы: ДНК лигаза
Компоненты-цепи: А и В
Cиноним назавания: НАД-зависимя полидеоксирибонуклеотидная синтаза
Код фермента: 6.5.1.2
Источник: пневманийный стрептококк
Метод получения трехмерной структуры: рентгеноструктурный анализ
Разрешение: 2.00 ангстрем
Малые молекулы: сульфат-ион, 7-метокси-6-метилптеридин-2,4-диамин, β-никотинамид рибоза-монофосфат, никотинамид-мононуклеотид
Формулы малых молекул: сульфат-ион - 2(O4S2-); диамин - 2(С8H10N6O); β-NMN - C11H16N2O8P1+; 110 H2O
Сравнение pdb-файла белка LigA и его биологической единицы
Первое отличие двух файлов - их расширения (.pdb у первого файла с белком, .pdb1.gz - у файла с биологической единицей).
Для выявления различий в структуре белков я получила их изображения в Jmol с помощью одного и того же приведенного выше скрипта, меняя только название исходного файла. Результат на рисунке 2.
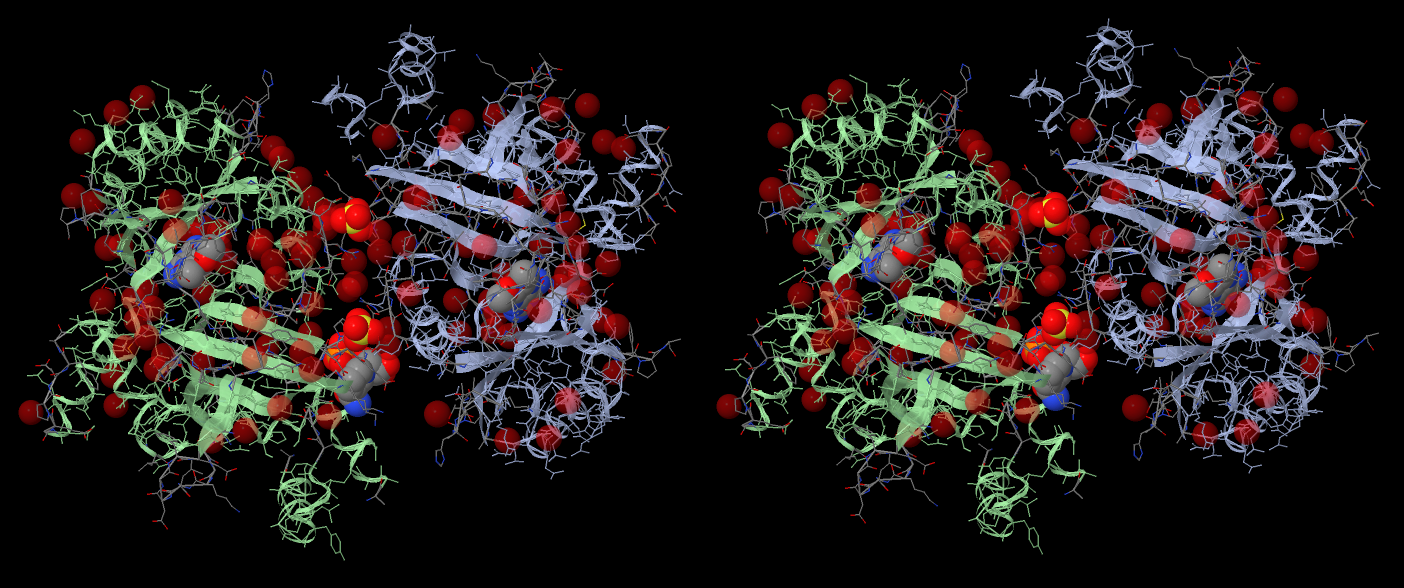
Рис. 2. Сравнение пространственной структуры белка LigA (справа) и его биологической единицы (слева)
Из рисунка 2 видно, что пространственные структуры белков очень похожи, если не идентичны.
© Дарья Горбачева | изменено 8.08.2014 |

