| Главная страница | Семестры | О себе | Ссылки |
Описание ДНК-лигазы и ортологов
С помощью ID Mapping, зная идентификатор PDB для своего белка, я узнала его код доступа в банк Uniprot - C1CKI0.
Ортолог белка - это белок, выполняющий ту же функцию, что и данный, но принадлежащий другому организму. Для поиска ортолоков ДНК-лигазы пневмококка я воспользовалась функцией Advanced Search, составив такой запрос:
taxonomy:Streptococcus NOT taxonomy:pneumoniae AND name:"DNA Ligase" |
Запрос выдал мне 554 записи из банка TrEMBL и 27 - из SwissProt. Из проверенных записей я выбрала две, информация о которых представлена в таблице 1.
Таблица 1. Сравнение НАД-зависимой ДНК-лигазы пневмококка и ее ортологов
Метка поля |
Информация о ДНК-лигазе из разных организмов |
|||
Streptococcus pneumoniae |
Streptococcus suis |
Streptococcus thermophilus |
||
Идентификатор записи |
ID |
DNLJ_STRZP |
DNLJ_STRSY |
DNLJ_STRT1 |
Код первый доступа |
AC |
C1CKI0 |
A4VVK7 |
Q5LYL9 |
Остальные коды доступа |
AC |
нет |
нет |
нет |
Дата создания документа |
DT |
28-JUL-2009 |
15-JAN-2008 |
15-JAN-2008 |
Дата последнего исправления аннотации |
DT |
16-APR-2014 |
16-APR-2014 |
16-APR-2014 |
Названия белка |
DE |
DNA ligase Polydeoxyribonucleotide synthase [NAD(+)] EC=6.5.1.2 (по класс. ферментов) |
DNA ligase Polydeoxyribonucleotide synthase [NAD(+)] EC=6.5.1.2 (по класс. ферментов) |
DNA ligase Polydeoxyribonucleotide synthase [NAD(+)] EC=6.5.1.2 (по класс. ферментов) |
Название организма |
OS |
Streptococcus pneumoniae (strain P1031) |
Streptococcus suis (strain 05ZYH33) |
Streptococcus thermophilus (strain CNRZ 1066) |
Таксономия |
OC |
Bacteria; Firmicutes; Bacilli; Lactobacillales; Streptococcaceae; Streptococcus |
Bacteria; Firmicutes; Bacilli; Lactobacillales; Streptococcaceae; Streptococcus |
Bacteria; Firmicutes; Bacilli; Lactobacillales; Streptococcaceae; Streptococcus |
Названия гена |
GN |
ligA SPP_1122 |
ligA SSU05_1180 |
ligA str1554 |
№ публикации |
RX |
нет |
PubMed=17375201; DOI=10.1371/ journal.pone.0000315 |
PubMed=15543133; DOI=10.1038/nbt1034 |
Авторы публикации |
RA |
Hotopp J.D., Censini S., Masignani V., Covacci A., Tettelin H. |
Chen C., Tang J., Dong W., Wang C., Feng Y., Wang J., Zheng F., Pan X., Liu D., Li M., Song Y., Zhu X., Sun H., Feng T., Guo Z., Ju A., Ge J., Dong Y., Sun W., Jiang Y., Wang J., Yan J., Yang H., Wang X., Gao G.F., Yang R., Wang J., Yu J. |
Bolotin A., Quinquis B., Renault P., Sorokin A., Ehrlich S.D., Kulakauskas S., Lapidus A., Goltsman E., Mazur M., Pusch G.D., Fonstein M., Overbeek R., Kyprides N., Purnelle B., Prozzi D., Ngui K., Masuy D., Hancy F., Burteau S., Boutry M., Delcour J., Goffeau A., Hols P. |
Название публикации |
RT |
"Complete genome sequence of Streptococcus pneumoniae strain P1031." |
"A glimpse of streptococcal toxic shock syndrome from comparative genomics of S. suis 2 Chinese isolates." |
"Complete sequence and comparative genome analysis of the dairy bacterium Streptococcus thermophilus." |
Журнал |
RL |
Submitted (DEC-2007) to the EMBL/GenBank/DDBJ databases |
PLoS ONE 2:E315-E315(2007) |
Nat. Biotechnol. 22:1554-1558(2004) |
Чем обосновано существование белка |
PE |
Evidence at protein level |
Inferred from homology |
Inferred from homology |
Ссылки на 3D структуры PDB |
DR |
PDB; 4GLW; X-ray; 2.00 A; A/B=1-305 |
нет |
нет |
Ионы-кофакторы |
СС |
COFACTOR: Magnesium or manganese (By similarity) |
COFACTOR: Magnesium or manganese (By similarity) |
COFACTOR: Magnesium or manganese (By similarity) |
FT METAL |
395 395 Zinc (By similarity) 398 398 Zinc (By similarity) 413 413 Zinc (By similarity) 418 418 Zinc (By similarity) |
395 395 Zinc (By similarity) 398 398 Zinc (By similarity) 413 413 Zinc (By similarity) 418 418 Zinc (By similarity) |
395 395 Zinc (By similarity) 398 398 Zinc (By similarity) 413 413 Zinc (By similarity) 418 418 Zinc (By similarity) |
|
Активный центр |
FT ACT_SITE |
109 109 N6-AMP-lysine intermediate (By similarity) |
109 109 N6-AMP-lysine intermediate (By similarity) |
109 109 N6-AMP-lysine intermediate (By similarity) |
Как видно из таблицы, ДНК-лигаза из организма пневмококка изучена гораздо лучше ортологов: расшифрована ее трехмерная стуктура, доказательства существования более весомые. Скорее всего, это связано с высоким практическим значением бактерии-возбутителя отита и бактериальной пневмонии.
Ответы на вопросы
Мне предлагалось ответить на несколько вопросов из данного списка.
Какие ионы связываются с белком?
Как указано в таблице 1, с ДНК-лигазой (и ее ортологами) связываются ионы магния и марганца, улучшая ее ферментативную активность. Также все три белка связывают цинк (цифры в таблице обозначают начало и конец участка связывания). Отсутствие ионов в PDB-файле можно объяснить удалением во время подготовки пробы белка к рентгеноструктурному анализу.
Последовательности большинства белков начинаются с метионина. Почему? После биосинтеза в процессе созревания белка метионин может быть удален. Указан ли метионин в начальной позиции заданного белка? А удаляется ли он потом?
Метионин кодируется наиболее распространенным старт-кодоном AUG (триплет ДНК ATG). Из полного генома Streptococcus pneumoniae (база данных NCBI) я узнала, что ген ligA действительно начинается с ATG (рисунок 1).
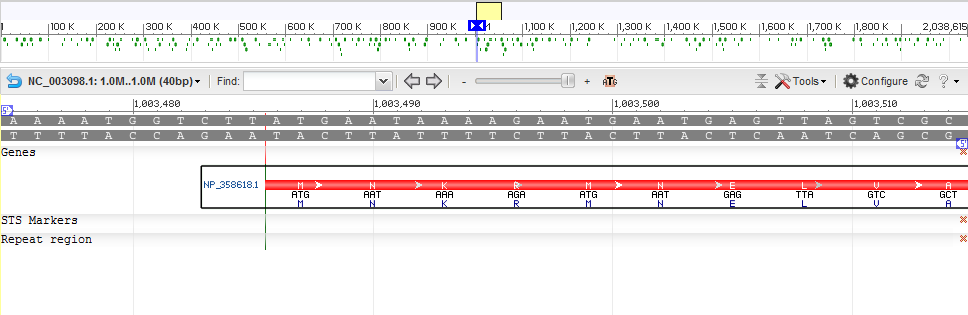
Рис. 1. Начало гена ligA в геноме пневмококка. Рисунок получен с помощью геномного браузера на сайте NCBI
На трехмерных структурах, задаваемых PDB-файлом с белком и его биологической единицей, первый остаток ни для одной из субъединиц не изображен, однако в поле PEMARK PDB-файла также сказано, что 1-36 субъединицы A и остатки 1-35 и 305 субъединицы В отсутствовали во время эксперимента (скорее всего, были удалены на этапе пробоподготовки). В качестве первых остатков действительно указаны метионины, значит, начальные участки субъединиц не подвергаются посттрансляционным изменениям. Все остатки, кроме первых двух метионинов и последнего аланина субъединицы В, изображены на трехмерной структуре, значит, их положение было предсказано. Но тогда я не понимаю, почему нельзя было изобразить и три отсутствующих остатка.
Какие аминокислотные остатки участвуют в образовании активного центра?
Из таблицы 1 ясно, что активный центр ДНК-лигазы образован 109-ми остатками лизина. К лизину присоединяется аденозинмонофосфат, что в дальнейшем делает возможным связывание участков ДНК.
© Дарья Горбачева | изменено 8.08.2014 |

