| Главная страница | Семестры | О себе | Ссылки |
Предсказание генов X5 с помощью AUGUSTUS
В задании требовалось предсказать гены в одном из скэффолдов сборки генома Х5. Для поиска подходящего скэффолда я с помощью команды seqretsplit разбила X5.fasta на множество файлов-скэффолдов (рисунок 1) и выбрала себе один на основании размера, остальные удалила. Результат: scaffold-262.fasta, длина 48647 нуклеотидов (узнала с помощью infoseq - рисунок 2).
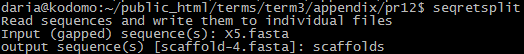
Рис. 1. Команда seqretsplit
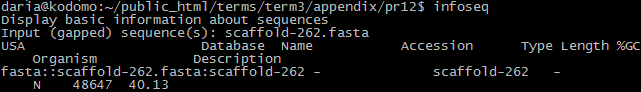
Рис. 2. Команда infoseq
Далее нужно было выбрать организм-эталон из списка таковых для работы AUGUSTUS. Для этого я запустила по скэффолду BLAST, a точнее blastx - транслирует последовательность в шести рамках и использует результаты для поиска похожестей. Хорошо то, что такой подход учитывает интроны. Результат на рисунке 3.
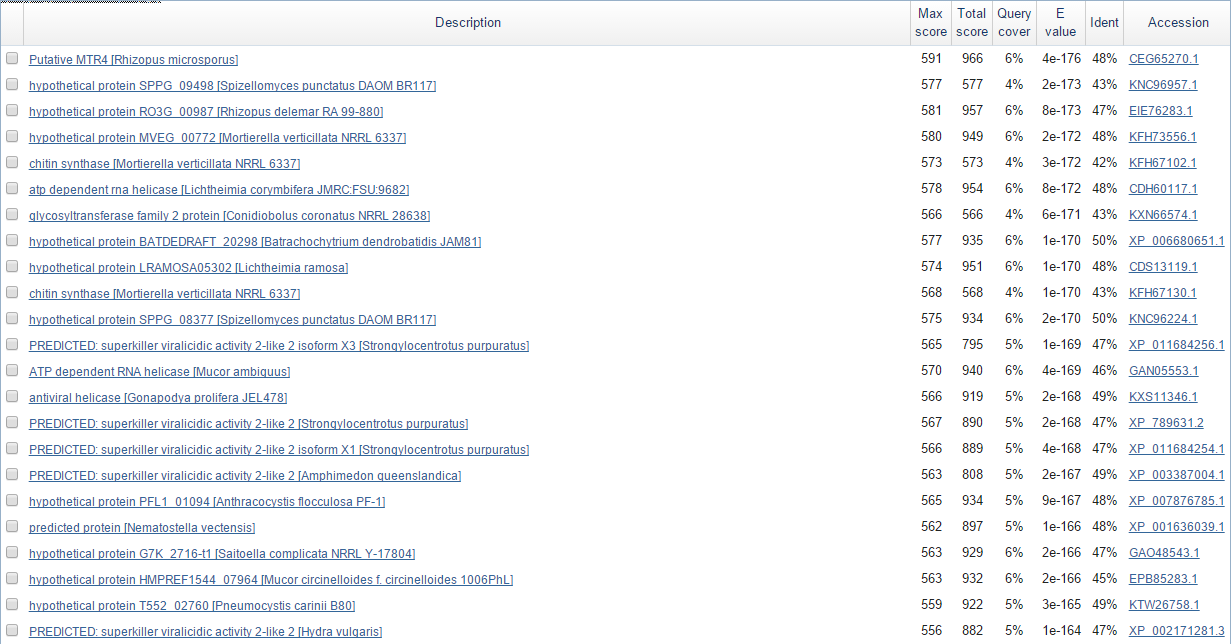
Рис. 3. Выдача BLAST по последовательности скэффолда
По счастью две из трех лучших находок принадлежат роду Rhizopus, представитель которого есть в эталонах - Rhizopus oryzae. Его и выберу.
Поля, кроме организма-эталона и файла с самой последовательностью, остались по умолчанию. Страница задания. В итоге выдача алгоритма составила шесть файлов:
augustus.aa - аминокислотные последовательности продуктов предсказанных генов в формате .fasta.
augustus.cdsexons - нуклеотидные последовательности предсказанных экзонов в формате .fasta.
augustus.codingseq - кодирующие последовательности каждого гена в формате .fasta (включая старт- и стоп-кодоны).
augustus.gbrowse - информация о трансляции: инициатор, терминатор, направление цепи, инторы, экзоны, старт- и стоп-кодоны.
augustus.gff - вся вышеперечисленная информация в одном формате.
augustus.gtf - вся вышеперечисленная информация в другом формате.
Проверка предсказаний
Судя по augustus.aa, в скэффолде было предсказано 18 белок-кодирующих генов, для проверки я случайно выбала g5, g6, g8, g11 и g16.
g5
Много хороших похожих находок (доказательство - рисунок 4), многие из которых относятся к одному и тому же белку - цистеин-протеиназе, поэтому возможно, что g5 имеет к ней отношение. Однако выравнивания находок начинаются не с начала белка, а на несколько сот нуклеотидов позже начала. Типичное выравнивание на рисунке 5. Делаю вывод, что была близко к истине предсказана экзон-интронная структура, но неверно определены координаты начала.
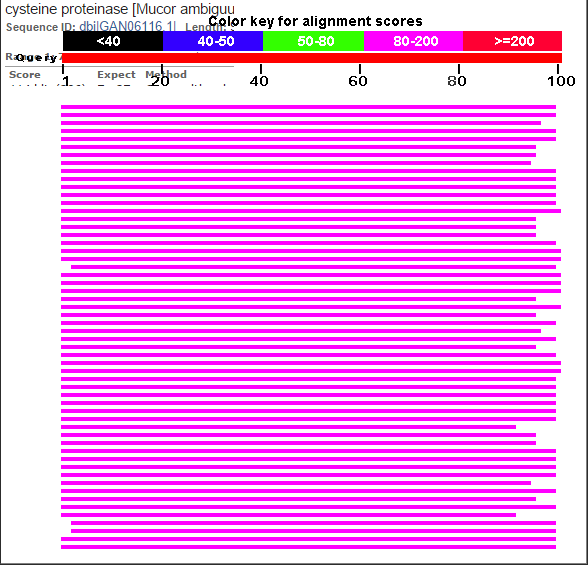
Рис. 4. Резюме выдачи blastp на g5
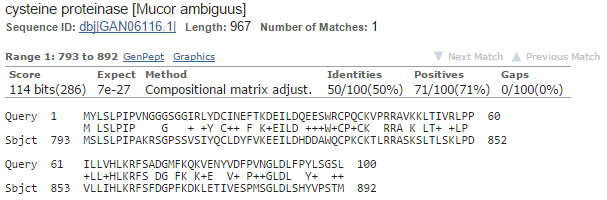
Рис. 5. Типичное выравнивание g5 и находки
g6
Тоже много хороших находок (на рисунке 6). Да и координаты начала, в отличие от предыдущего случая, похоже, определены верно (на рисунке 7 типичное выравнивание с более-менее совпадающими номерами остатков). О функции g6 тоже можно судить - белок cdc4, скорее всего. AUGUSTUS сработал на ура.
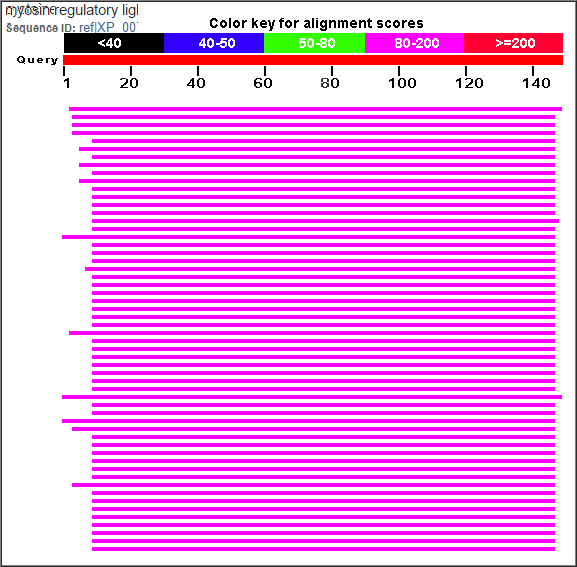
Рис. 6. Резюме выдачи blastp на g6
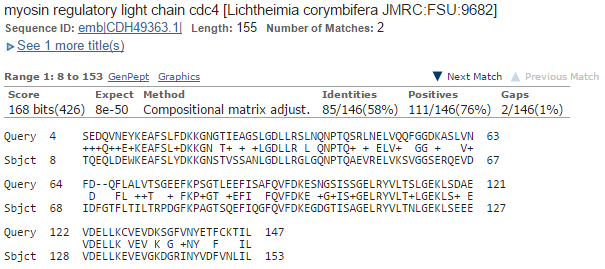
Рис. 7. Типичное выравнивание g6 и находки
g8
"No significant similarity found." - BLAST ничего не нашел. Значит, белок-кодирующий ген был предсказан неверно.
g11
BLAST выдал один недостоверный результат (рисунок 8). Видимо, этот соответствующий g11 ген тоже был неверно предсказан.

Рис. 8. Выдача BLAST по последовательности g11
g16
Странный результат. Вроде бы находок много, но они плохи. Но и плохи как-то одинаково (рисунок 9). Выравнивания все не хорошие, но похожи и относятся в подавляющем большинстве к белку Daam2 (одно на рисунке 10). Скорее всего, AUGUSTUS правильно предсказал ген, но ошибся и с началом, и с экзон-интронной структурой.
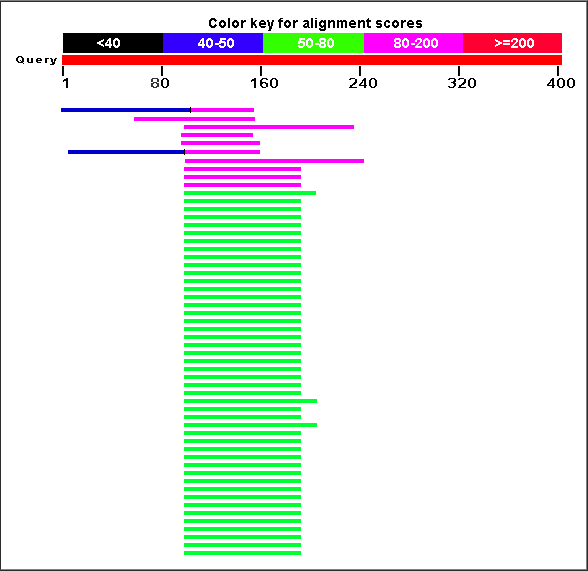
Рис. 9. Резюме выдачи blastp на g16
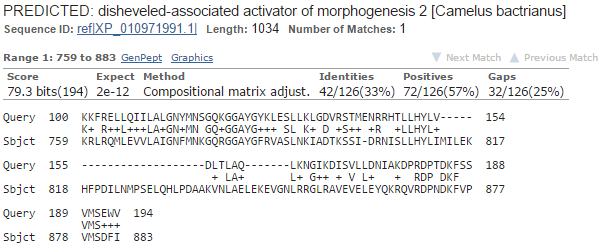
Рис. 10. Типичное выравнивание g16 и находки
Сравнение аннотации RefSeq и AUGUSTUS гена человека
Для работы был выбран pax9 - ген, важный в эмбриогенезе. Влияет на развитие многих органов - например, зубов.
Этот ген я нашла в браузере UCSC, в частности, в последней сборке генома h38. В выдаче оставила только три трэка: base position, Refseq и AUGUSTUS, результат можно видеть на рисунке 11.
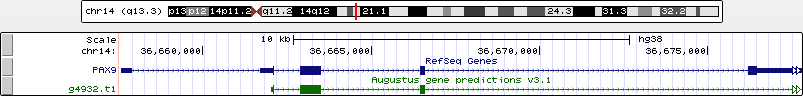
Рис. 11. Аннотации pax9, полученные с помощью UCSC Genome Browser
Судя по UCSC, ген находится в 14-ой хромосоме, координаты: 36657568-36677804, соотсетственно, длина: 20237 п.о., на прямой цепи.
С помощью Tools => Table Browser я получила таблицу, следуя указаниям в задании (конечно, кое-как обработав данные руками). Результат не впечатляет. Если постулировать истинность аннотации RefSeq, то AUGUSTUS ошибся сильно: верно определил только 2 из 5 экзонов (но к чести AUGUSTUS - определил абсолютно верно), длину самого гена предсказал в 3 раза больше истинной.
© Дарья Горбачева | изменено 10.03.2016 |

