| Главная страница | Семестры | О себе | Ссылки |
Аннотирование генома бактерии с помощью RAST
Требовалось аннотировать прокариотическую последовательность с помощью ресурса RAST.
Я выбрала бактерию, с которой всегда работаю - пневмококк. Необходимая в отчете и использованная при формировании запроса информация о ней представлена в таблице 1.
Таблица 1. Информация о запросе на RAST
Таксономия |
cellular organisms; Bacteria; Firmicutes; Bacilli; Lactobacillales; Streptococcaceae; Streptococcus; Streptococcus pneumoniae strain R6 |
Taxonomy ID |
171101 |
Название генома |
Streptococcus pneumoniae R6 chromosome, complete genome |
Хромосома |
Кольцевая, полный геном |
GeneBank AC |
В качестве входного файла я подала RAST'у последовательность генома .fasta. Указала таксономию, параметры оставила по умолчанию.
Результат я скачала в формате книги Excel - 171101.14.xls.
Для сравнения результатов RAST'a и проверенных аннотаций я объединила некоторую информацию из полученной таблицы (оставив в ней только белок-кодирующие) с таблицей аннотированных генов из практикума "EMBOSS". Результат - таблица rastandannot.xlsx.
В общем, анннотации очень сходны, абсолютное большинство предсказаний генов одинаковы. RAST производит впечатления более точного источника информации.
C помощью разнообразных и нудных использований функции ЕСЛИ в Excel были получены следующие данные о сходстве аннотаций:
Одинаково аннотированные гены: 1365
Аннотированы только RAST'ом: 465
Аннотированы только RefSeq'ом: 169
Не совпадают границы: 280
Сравнение с аннотацией генов в записи GeneBank
Ген аннотирован только RAST'ом
Из таковых был случайно выбран ген, информация о котором в таблице 1.
Таблица 1. Аннотированный только RAST'ом ген
Начало |
Конец |
Предсказанный продукт |
Последовательность |
687590 |
688018 |
Гидролаза белков α/β |
Результат странный. Вроде бы blastx хорошо все нашел (рисунок 1), даже функции подходящие. Интересно, что находка на третьей позиции интуитивно кажется "следующей за" одним из аннотированных RefSeq'ом генов из таблицы (фрагмент с ним на рисунке 2). Поэтому складывается впечатление, что RAST верно предсказал ген.
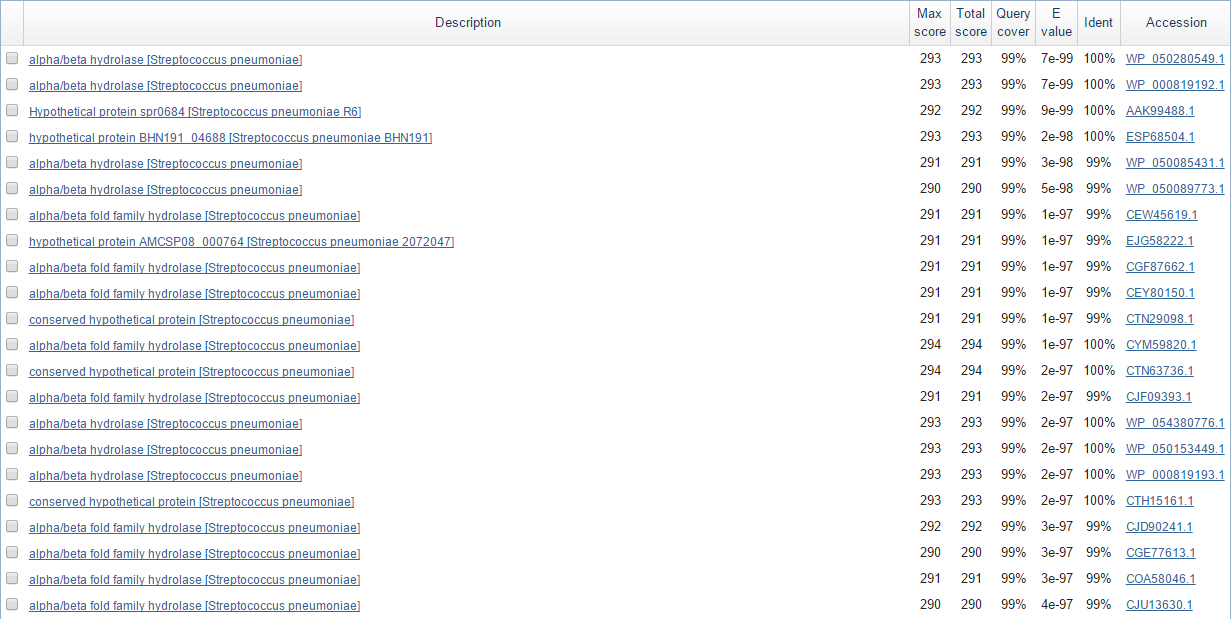
Рис. 1. Часть выдачи BLAST по последовательности предсказания

Рис. 2. Фрагмент общей таблицы аннотаций
Ген аннотирован только в RefSeq'е
Из таковых был случайно выбран ген, информация о котором в таблице 2.
Таблица 2. Аннотация гена существует только в RefSeq
Название |
Начало |
Конец |
Предсказанный продукт |
Последовательность |
867410 |
867559 |
L33 - рибосомальный белок |
По последовательности запустила blastx. Выдача (на рисунке 3) содержит очень много находок с огромной вероятностью гомологии. Последовательность отмечена как крайне консервативный домен бактерий, поэтому почти наверно ген был аннотирован правильно. Остается только гадать, как такой вроде бы самый очевидный ген проглядел RAST.
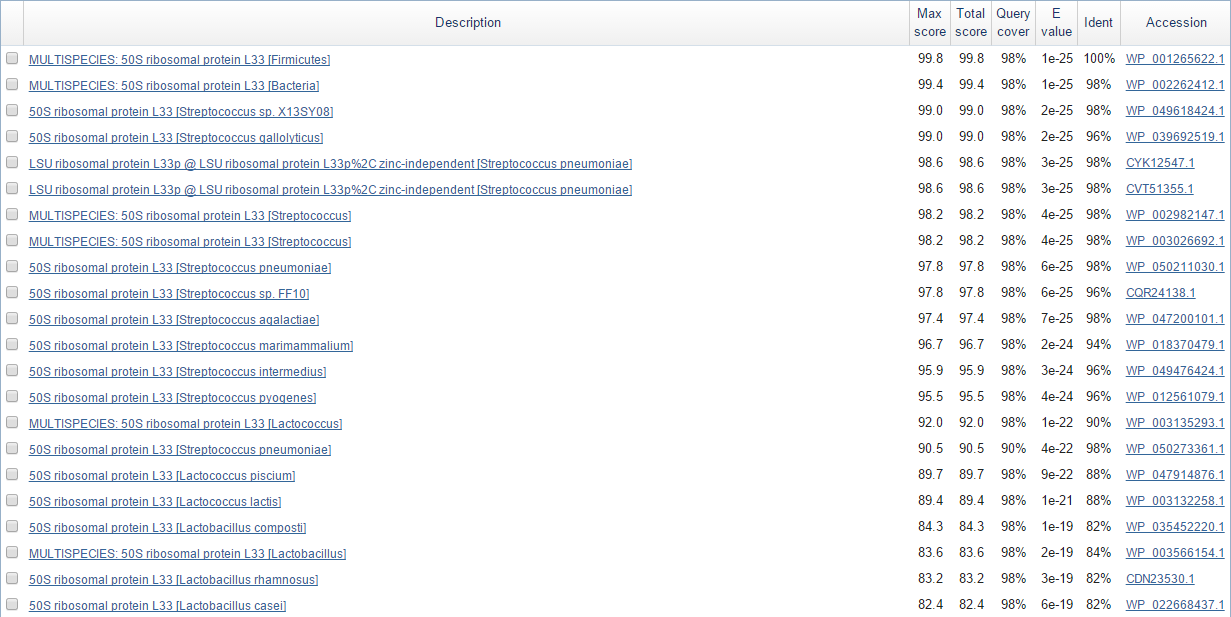
Рис. 3. Часть выдачи BLAST по rpmG
RAST предположил функцию гена c неизвестной функцией
Информация об одном таком гене в таблице 3.
Начало |
Конец |
Продукт по RefSeq |
Продукт по RAST/b> |
Последовательность |
1645644 |
1646180 |
Гипотетический белок |
SczA - регулятор транскрипции из семейства TetR |
И опять же выдача blastx говорит сама за себя (рисунок 4). Очень сложно предположить, что функция рассматриваемого гена - не белок из семейства TetR (хотя по находкам и нельзя судить, что это именно SczA). Опять же, удивительно, что такой ген до сих пор не получил подтверждения.
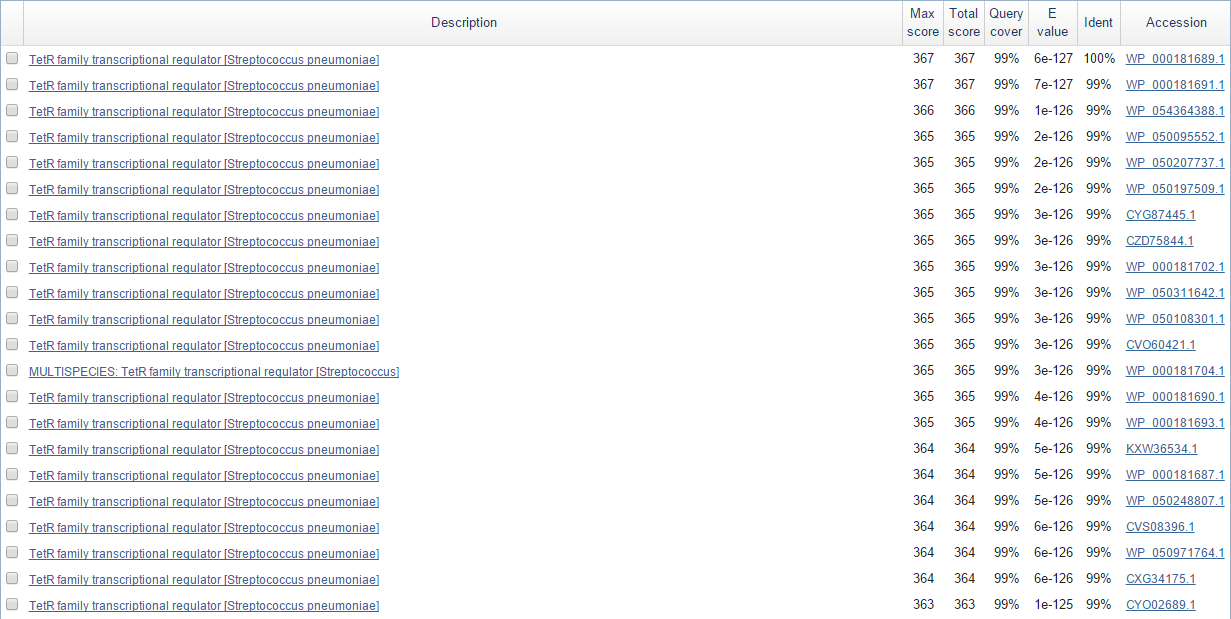
Рис. 4. Часть выдачи BLAST по предполагаемому sczA
© Дарья Горбачева | изменено 11.03.2016 |

