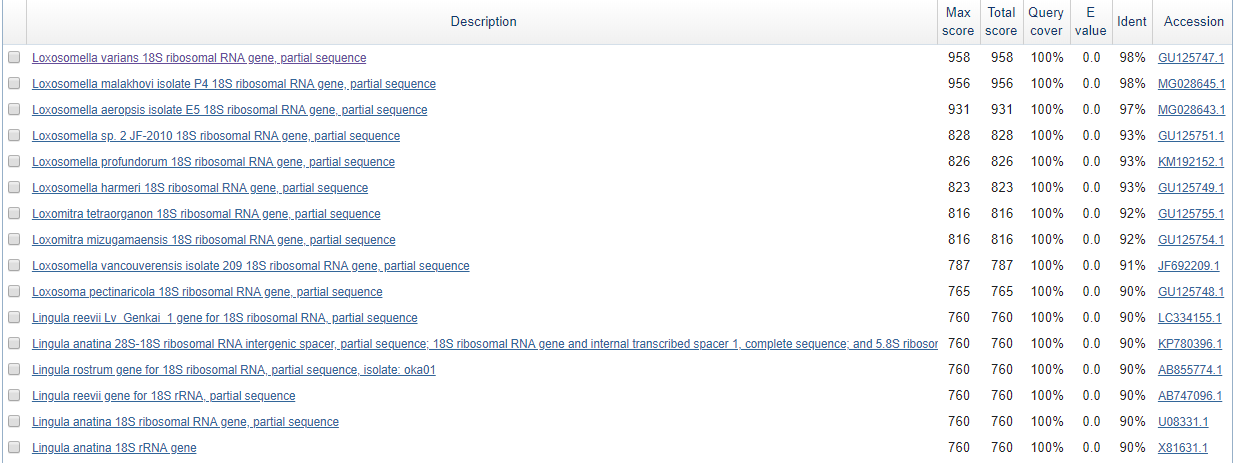
 Рис.1.1
Рис.1.1 Задания по BLAST
1. Таксономия и функции прочтённой последовательности
Прочтённая последовательность является частью гена, кодирующего рибосомальную 18S РНК. Вероятно, последовательность получена из представителя рода Loxosomella, полная таксономия которого, согласно NCBI: cellular organisms; Eukaryota; Opisthokonta; Metazoa; Eumetazoa; Bilateria; Protostomia; Lophotrochozoa; Entoprocta; Loxosomatidae; Loxosomella
Принадлежность последовательности к генам рибосомальной 18S РНК не вызывает сомнений, так как все находки BLAST относятся к этому типу генов.
 Рис.1.1
Рис.1.1
Принадлежность к роду Loxosomella можно предположить, исходя, например, из дерева, построенного blast по находкам:
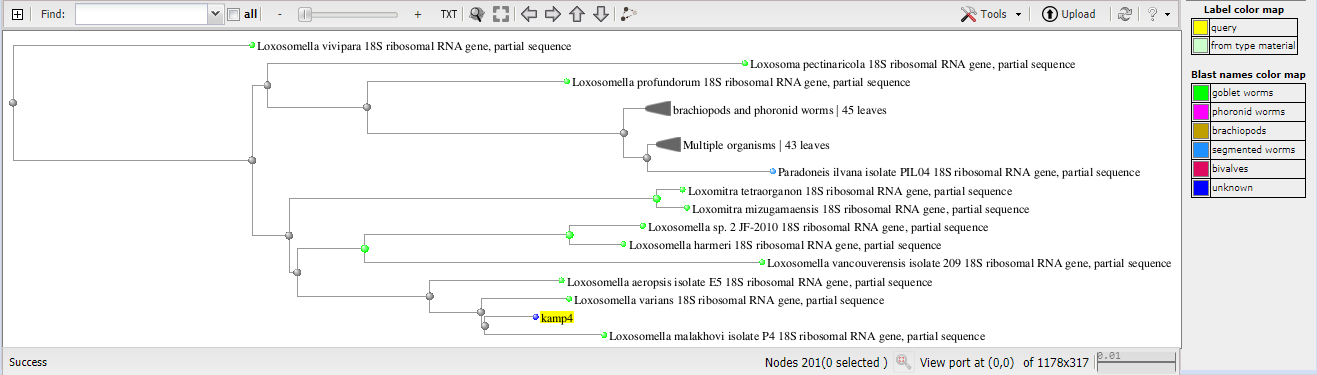 Рис.1.2.
Рис.1.2.
Дерево отражает расстояние между различными последовательностями (эволюционное расстояние, измеряемое количеством замен, вставок и делеций, отделяющих последовательности друг от друга). Видно, что наша последовательность располагается где-то внутри веера веток, ведущих к различным представителям рода Loxosomella.
2. Сравнение списка находок нуклеотидных последовательностей тремя разными вариантами BLAST
Сначала возьмём в качестве запроса последовательность из первого задания. Плодотворной идеей для снижения числа находок до обозримого оказалось ограничить поиск таксоном Bacteria. Поиск по базе nr (в Refseq ничего не ищется), галочки в полях "Exclude Uncultured/environmental sample sequences" и "Exclude Models (XM/XP)". Проделываем три поиска: Megablast со стандартными настройками, blastn co стандартными настройками, blastn с длиной слова 7 и Match/mismatch scores = 1;-1. На всякий случай везде меняем максимальное количество находок на 500. Получаем вот что:
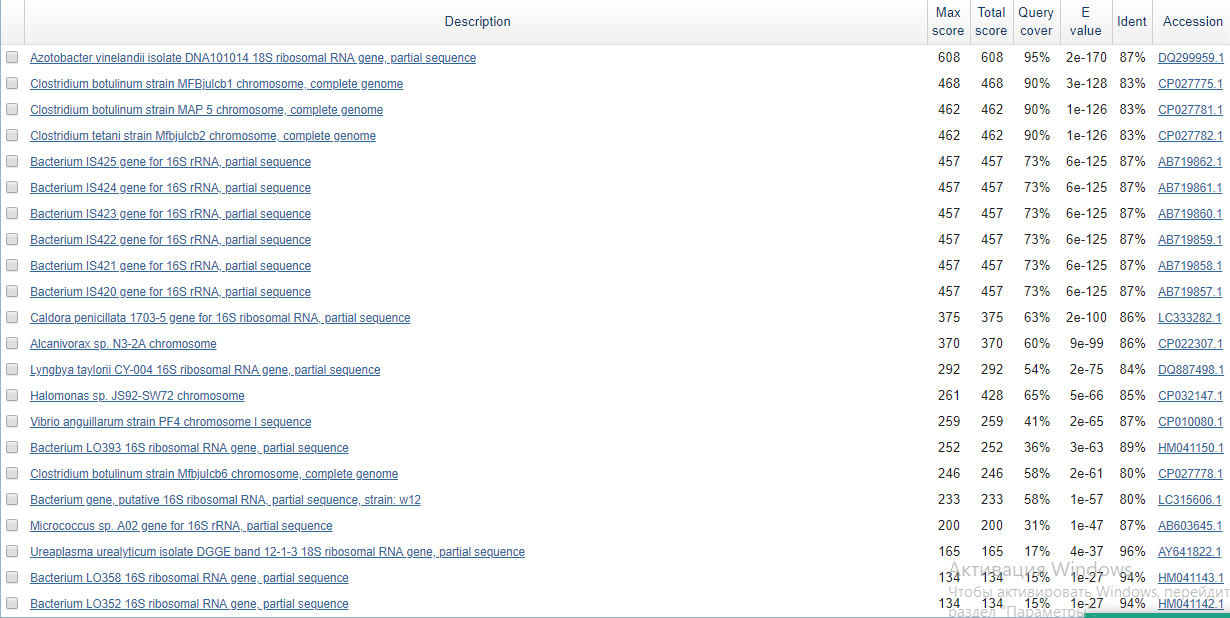 Рис.2.1. Megablast. 22 находки
Рис.2.1. Megablast. 22 находки
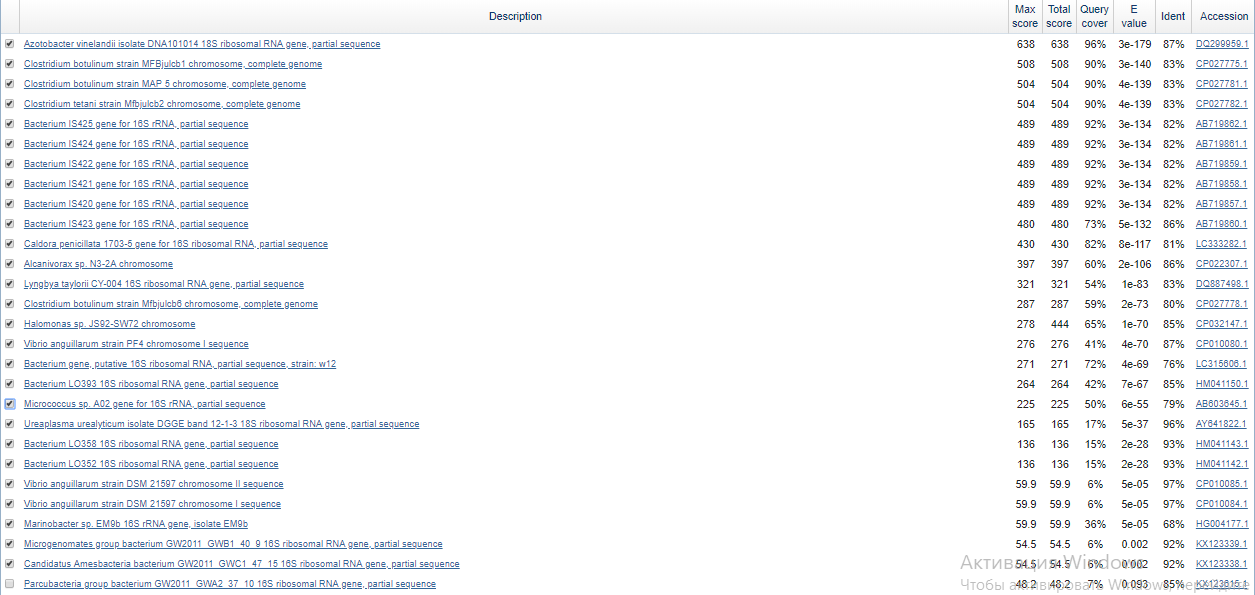 Рис.2.2. Часть выдачи blastn, стандартные настройки. Всего 66 находок,
27 из них - с e-value < 0.01
Рис.2.2. Часть выдачи blastn, стандартные настройки. Всего 66 находок,
27 из них - с e-value < 0.01
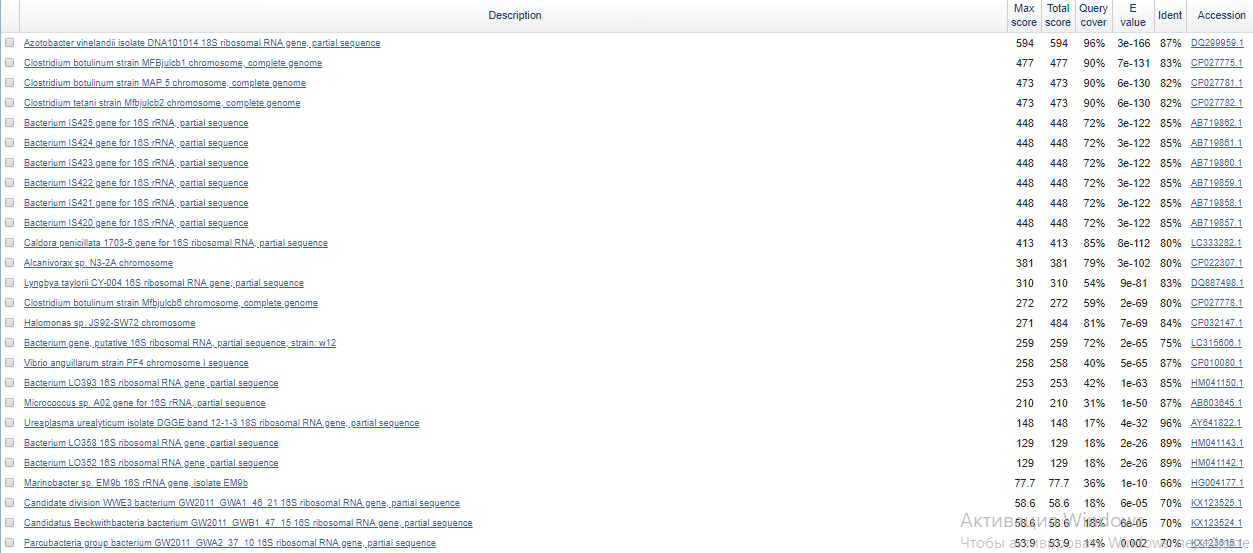 Рис.2.3. Часть выдачи blastn, чувствительные настройки. Всего находок 499, из
них 92 имеют e-value < 0.05
Рис.2.3. Часть выдачи blastn, чувствительные настройки. Всего находок 499, из
них 92 имеют e-value < 0.05
В следующей таблице приведено сравнение выдачи разных вариантов blast (рассмотрены находки с малым e-value). Голубым выделены находки, встретившиеся во всех трёх вариантах, а розовым - в обоих вариантах blastn. Видно, что самые значимые находки одинаковы во всех трёх случаях. С другой стороны, при переходе от blastn со стандартными настройками к более чувствительному blastn замечаем, что при длине слова 11, по-видимому, теряем некоторые находки из-за их "разреженности" (в целом, выравниваются неплохо, но нет достаточно длинных слов).
Таблица с выдачей разных вариантов blast
3. Гомологи трёх белков в неаннотированном геноме
Поиск гомологов проводился при помощи tblastn с порогом на evalue - 0.01.
HSP71_YEAST (шаперон HSP70, белок теплового шока) имеет гомологи в геноме Amoeboaphelidium protococcarum (по крайней мере, один - выравнивание с транслированным scaffold-199 хорошее и не содержит стоп-кодонов):
Score E
Sequences producing significant alignments: (Bits) Value
scaffold-199 920 0.0
scaffold-96 744 0.0
scaffold-423 737 0.0
unplaced-999 540 8e-171
unplaced-980 461 9e-142
scaffold-157 285 1e-81
scaffold-693 281 2e-80
unplaced-804 264 2e-74
scaffold-499 262 6e-74
unplaced-959 231 1e-63
scaffold-469 150 5e-43
scaffold-418 150 5e-43
unplaced-113 122 1e-32
scaffold-138 78.6 3e-17
scaffold-61 78.6 3e-17
unplaced-721 43.9 2e-05
Исходя из результатов BLAST по refseq-proteins, ограниченного таксоном Fungi, данный белок достаточно консервативен и представлен во многих таксонах ( см. таблицу). Самая худшая находка (из 250) - из организма Candida pseudohaemulonis, Score = 694, Query coverage = 93%, e-value = 0.0, Identity = 60%.
Тубулин (TBB_NEUCR) также имеет гомологи.
Score E
Sequences producing significant alignments: (Bits) Value
unplaced-665 742 0.0
scaffold-26 693 0.0
unplaced-5 348 1e-105
scaffold-57 348 1e-105
scaffold-423 161 6e-49
Тубулин - основной компонент микротрубочек. Микротрубочки у данного рассматриваемого организма имеются (ссылка). Логично, что есть и тубулин.
А вот фактор инициации трансляции eIF3g (EIF3G_SCHPO) гомолога в рассматриваемом геноме, видимо, не имеет:
Score E
Sequences producing significant alignments: (Bits) Value
scaffold-20 95.5 2e-21
scaffold-444 92.0 2e-20
scaffold-170 38.9 0.002
scaffold-17 37.4 0.008
РНК-узнающий мотив (RNA recognition motif, RRM) приходится, согласно Uniprot, в этом белке на 202-280 аминокислоты, которые ни в одном из случаев не попадают в выравнивание. Ни одно из полученных выравниваний не выглядит достаточно убедительным и, согласно результатам BLAST (параметры те же, что и в случае белка теплового шока), данный белок не является очень консервативным. Худший результат из 250 имеет Rhizopus microsporus ATCC 52813: Score = 35.4, Query coverage = 37%, e-value = 2e-04, Identity = 32%
4. Гены белков в контигах
При выполнении этого задания было просмотрено множество контигов. Во многих из них при помощи blastx, сделанного на сайте NCBI (поиск по refseq protein, таксон ограничен Fungi), с высоким весом и низким evalue находились гены каких-то белков. Однако во многих случаях результат выглядел неправдоподобно по двум причинам: маленький процент Identity (большой вес набирался просто за счёт большой длины участка) и, что более существенно, - большое количество стоп-кодонов в данной рамке-считывания контига. В процессе поиска мне так и не удалось подобрать пример, где внутри предполагаемого гена не нашлось бы стоп-кодонов. Понятно, что они все теоретически могли бы оказаться в интронах, однако в приведённых ниже участках контигов их больше пяти, что снижает правдоподобность полученных результатов.
 Рис.4.1. В контиге unplaced-10 нашёлся участок ABC-транспортера
Рис.4.1. В контиге unplaced-10 нашёлся участок ABC-транспортера
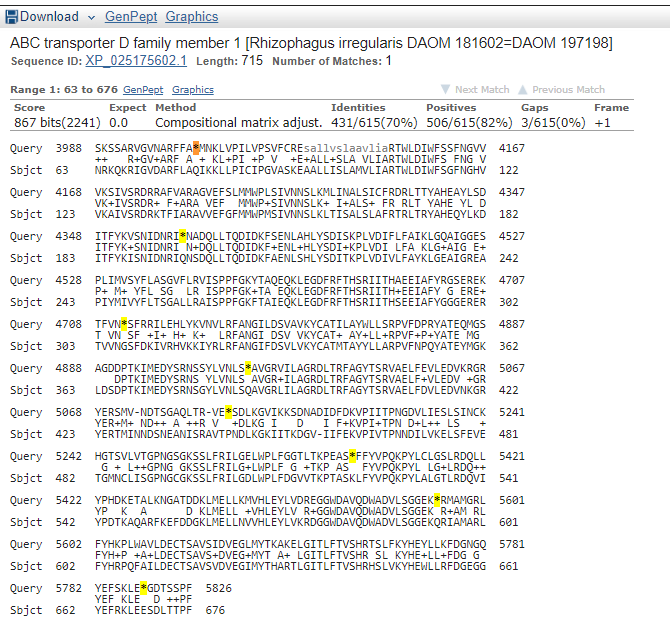 Рис.4.2. Транслированный участок контига выравнивается хорошо, однако
в нём много стоп-кодонов(*) и выравнивание не захватывает начало ABC-транспортера, поэтому начало гена в контиге
не определить
Рис.4.2. Транслированный участок контига выравнивается хорошо, однако
в нём много стоп-кодонов(*) и выравнивание не захватывает начало ABC-транспортера, поэтому начало гена в контиге
не определить
 Рис.4.3. В контиге unplaced-1034 нашлись гены белков:
фосфоманномутазы, контролирующего клеточное деление белка, протеинкиназы. За достоверность находки первого и
третьего генов говорит то, что рядом независимо находятся кодирующие последовательности сразу нескольких
кусочков белка
Рис.4.3. В контиге unplaced-1034 нашлись гены белков:
фосфоманномутазы, контролирующего клеточное деление белка, протеинкиназы. За достоверность находки первого и
третьего генов говорит то, что рядом независимо находятся кодирующие последовательности сразу нескольких
кусочков белка
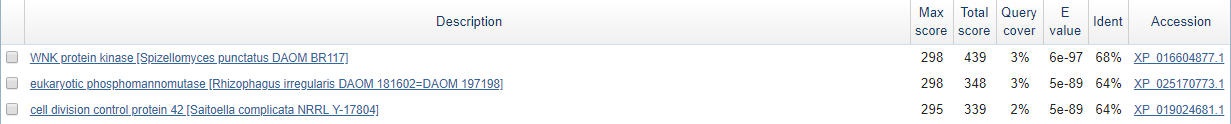 Рис.4.4
Рис.4.4
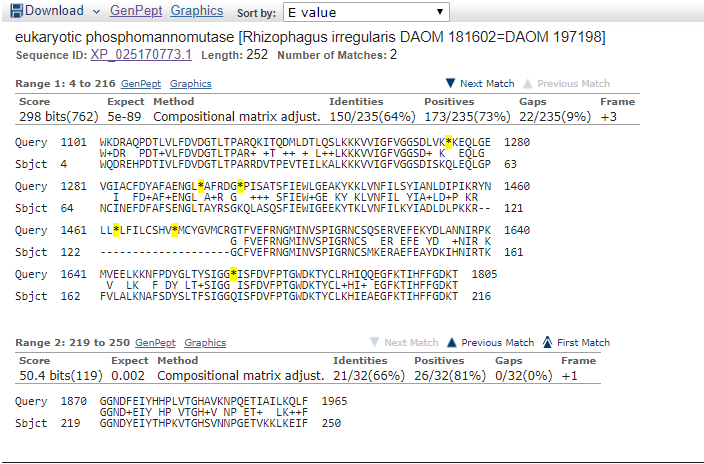 Рис.4.5. Выравнивание со вторым в списке белком (фосфоманномутаза).
Выравнивание хорошо тем, что в транслированном участке контига относительно мало стоп-кодонов (меньше, чем в лучшей
находке). Помимо этого, фосфоманномутаза нашлась почти целиком (всего в этом белке 252 аминокислоты). Таким образом,
можно предположить, что примерные координаты гена этого белка на контиге - 1090-1810. Стоит отметить, что
фосфоманномутаза принадлежит тому же организму, что и ABC-транспортер в первом примере.
Рис.4.5. Выравнивание со вторым в списке белком (фосфоманномутаза).
Выравнивание хорошо тем, что в транслированном участке контига относительно мало стоп-кодонов (меньше, чем в лучшей
находке). Помимо этого, фосфоманномутаза нашлась почти целиком (всего в этом белке 252 аминокислоты). Таким образом,
можно предположить, что примерные координаты гена этого белка на контиге - 1090-1810. Стоит отметить, что
фосфоманномутаза принадлежит тому же организму, что и ABC-транспортер в первом примере.
5. Карта локального сходства геномов двух бактерий
Для рассмотрения был выбран род Mycoplasma. Найти двух подходящих бактерий оказалось нетривиальной задачей, потому что для геномов бактерий разных видов BLAST зачастую выдавал примерно такую картину:
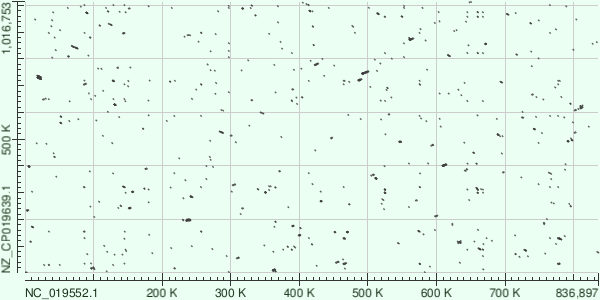 Рис.5.1. BLAST(blastn) для свиной и бычьей микоплазм
(Mycoplasma hyorhinis SK76, Mycoplasma bovis strain 08M). Есть общие гены, но их порядок полностью
нарушен.
Рис.5.1. BLAST(blastn) для свиной и бычьей микоплазм
(Mycoplasma hyorhinis SK76, Mycoplasma bovis strain 08M). Есть общие гены, но их порядок полностью
нарушен.
В то время как для геномов штаммов одного вида результатом было примерно следующее:
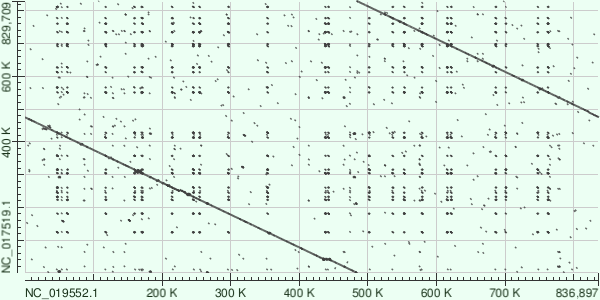 Рис.5.2. BLAST для двух штаммов свиной микоплазмы.
Учитывая, что геномы кольцевые, такая карта означает почти 100% сходство (нет даже перестроек между
участками).
Рис.5.2. BLAST для двух штаммов свиной микоплазмы.
Учитывая, что геномы кольцевые, такая карта означает почти 100% сходство (нет даже перестроек между
участками).
И, наконец, удалось получить некий промежуточный вариант:
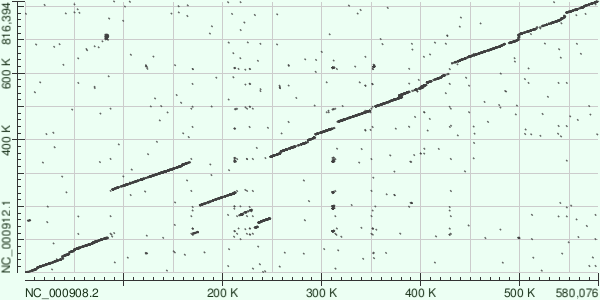 Рис.5.3. BLAST(megablast) для
Mycoplasma genitalium G37 и Mycoplasma pneumoniae M129 (AC NC_000908 и NC_000912 соответственно).
Рис.5.3. BLAST(megablast) для
Mycoplasma genitalium G37 и Mycoplasma pneumoniae M129 (AC NC_000908 и NC_000912 соответственно).
Последний BLAST выполнен со стандартными параметрами megablast. Рассмотрим карту локального сходства этих организмов. Геном Mycoplasma pneumoniae M129 примерно на 250 тыс. пн длиннее, чем геном Mycoplasma genitalium G37. Судя по карте локального сходства, либо у Mycoplasma pneumoniae M129 имеются вставки, либо, что более вероятно в процессе эволюции паразитического организма, у Mycoplasma genitalium G37 произошли делеции ненужных участков ("лестничный" вид, начиная примерно с 250К по горизонтальной оси). Рассмотрим теперь левую половину карты и поймём, как из M. genitalium получить M. pneumoniae: участки 80-180K и 210-250K меняем местами, первый из них затем оставляем нетронутым, а второй делим пополам и половинки меняем местами. Вряд ли именно такой сценарий имел место в живой природе, но зато он даёт описание расположения предположительно гомологичных участков в данных бактериях.
© Быкова Даша, 2018