Поиск сигнала посадки σ-субъединицы РНК-полимеразы
Подготовка данных
Для выполнения практикума выбрана Escherichia coli штамма K-12 подштамма MG1655. Для нее были скачаны последовательность хромосомы и аннотация в формате GFF. С помощью веб-приложения Operon-mapper на основе fasta-файла с последовательностью генома и таблицы особенностей были определены координаты оперонов. Выдача содержала информацию о 4445 оперонах, что как раз примерно соответствует числу генов E.coli
Далее с помощью скрипта (автор Георгий Муравьев) были подготовлены следующие данные:
- материал обучения housekeeping.fasta — промоторы генов домашнего хозяйства, поиск таких генов осуществлялся по ключевым словам в колонке Function выдачи Operon-mapper (рибосомальный, полимераза, транскрипция, трансляция, АТФ-синтаза). В качестве промотора выбиралась область 100 нуклеотидов (нт) перед началом оперона.
- материал тестирования promoters.fasta — промоторы всех оперонов
- негативный контроль negative.fasta — участки длиной 100 нт, вырезанные из генома случайным образом
Запуск MEME
Для поиска сигналов в последовательностях был использован локальный MEME со следующими параметрами: поиск в ДНК (-dna), число мотивов для поиска — 3 (-nmotifs 3), длина находки от 6 до 50 нуклеотидов (-minw 6 -maxw 50), остальное по умолчанию
meme housekeeping.fasta -dna -nmotifs 3 -minw 6 -maxw 50
| Находка | E-value |
|---|---|
| GCRYMHKDTGYGCDTYSHGCYTCGMCGWYYTGWYSGCGYTS |
8.9e-009 |
| TAGCABTACSCCYKCGTGAHDCCACCTTYYAKCGWDTVAGDGSBTGCCA |
1.9e-002 |
| WTSGACRCCTYTTTCWSTTT | 1.3e-001 |
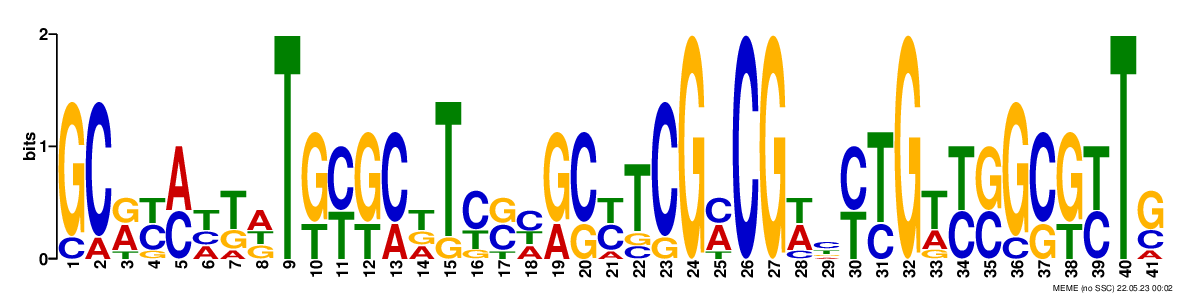


Выбрал мотив c наименьшим E-value — GCRYMHKDTGYGCDTYSHGCYTCGMCGWYYTGWYSGCGYTS.
Его паттерн (пункт "regular expression" в текстовой выдаче) - GC[AG][CT][AC][TAC][TG][AGT]T[GT][CT][GT][CA][TAG]T[CT][CG][CAT][GA][CG][CT]TCG[AC]CG[AT][CT][CT][TC]G[TA][TC][GC]G[CG][GT][TC]T[GC]
Запуск FIMO
Для поиска мотива в положительном и отрицательном контроле были запущены следующие команды:
fimo --norc -motif GCRYMHKDTGYGCDTYSHGCYTCGMCGWYYTGWYSGCGYTS -thresh 0.0125 ./meme_out/meme.txt promoters.fasta
fimo --norc -motif GCRYMHKDTGYGCDTYSHGCYTCGMCGWYYTGWYSGCGYTS -thresh 0.0125 ./meme_out/meme.txt negative.fasta
Поиск проводился только по одной цепи (--norc), сначала порог на p-value (-thresh) был взят 0.001, получилось 416 находок (fimo_0001.tsv) в материале тестирования, маловато, ведь всего в нем 4445 промоторных последовательностей. С порогом 0.01 получилось 3368 находок (fimo_001.tsv), уже лучше, но еще не все сайты посадки нашлись. С "классическим" порогом на p-value до 0.05 находок было уже 15285 (fimo_005.tsv), это избыточно. Думаю, наилучший вариант порога 0.0125 – 4175 находок (fimo_00125.tsv), примерно столько же, сколько и промоторов.
В негативном контроле с теми же параметрами было найдено всего 449 последовательностей (fimo_00125_negative.tsv)