| AC | PF01018 | ||||||||
| ID | GTP1_OBG | ||||||||
| Функция | N-терминальный домен ГТФазы obgE, Spo0B-ассоциированного белка. ObgE ГТФаза играет важную роль при переходе от вегетативного роста к споруляции у бактерий. Есть данные об взаимодействии данного белка с рибосомами. Ингибируется повышением содержания ГДФ в клетке. Локализован в цитоплазме. | ||||||||
| Структура | Имеет уникальную третичную структуру, образованную сэндвич-структурой из 8 бета-тяжей и трех богатых глицином регионов, которые формируют 6 левозакрученных аналогичных коллагену спиралей. | ||||||||
| Число разных доменных архитектур с этим доменом | 22 | ||||||||
| Число представителей в разных таксонах высшего порядка |
|
| Архитектура | Число представителей | |
| 1: GTP1_OBG + MMR_HSR1 + DUF1967 |  | 1312 |
| 2: GTP1_OBG + MMR_HSR1 |  | 1789 |
| AC | PF01926 | ||||||||
| ID | MMR_HSR1 | ||||||||
| Функция | Взаимодействует с 50S рибосомальной субчастицей и связывает пуриновые нуклеотиды (гуанин предпочтительнее). | ||||||||
| Число разных доменных архитектур с этим доменом | 150 | ||||||||
| Число представителей в разных такосонах высшего порядка |
|
| AC | PF09269 | ||||||||
| ID | DUF1967 | ||||||||
| Функция | DUF = domain of uknown function, но в статье упоминается, что DUF1967 похож по структуре на домен, обнаруженный в стрессовых белках, например в SpoT – белке, который участвует в метаболизме нуклеотида ppGpp. Было установлено, что OBG действительно может связывать ppGpp. | ||||||||
| Число разных доменных архитектур с этим доменом | 8 | ||||||||
| Число представителей в разных такосонах высшего порядка |
|
Таблица Excel.
Выравнивание 31 выбранной последовательности
Последовательности были зашифрованы: первая цифра в названии - номер архитектуры (1 или 2), буква следующая за цифрой - таксон (В - бактерии, Е - эукариоты).
Для построения дерева использовались алгоритмы Neighbor joining (fneighbor) , Maximum parsimony (fprotpars) , UPGMA , а также bootsrap анализ. Наибольший интерес вызывает дерево, полученное с помощью UPGMA.
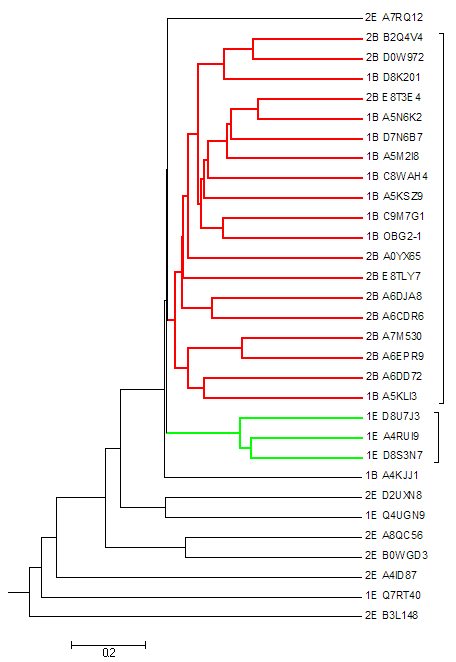
На нем четко выделилась клада, объединяющая бактерий (не попала в нее только одна последовательность).
Зная, что UPGMA строит дерево “сверху вниз”, объединяя сначала две ближайших последовательности и считая их далее одним кластером, есть основания полагать, что бактериальные последовательности действительно чрезвычайно близки между собой. При этом дерево укоренено совершенно неверно. Это говорит нам о том, что на каком-то этапе постройки расстояние от кластера с бактериальными последовательностями до некоторой эукариотической оказалось ближе чем до любого другого OBG белка эукариот и причем ситуация повторилась несколько раз. Это странно, потому что у эукариот система репарации эффективнее. Я думаю, что у эукариот OBG белки уже не имеют особого значения, что и отражается в числе мутаций. Различия между OBG белками эукариот оказались заметны и при построении профиля.
Определенное любопытство вызвала клада, содержащая последовательности Selaginella moellendorffii (плаунок) и водорослей родов Ostreococcus и Volvox. В клетках селягинеллы OBG локализуется во внутренней мембране и матриксе хлоропластов. Есть данные о синтезе ppGpp в хлоропластах, что возможно связано с его функцией в растениях.
Итак, очевидно, что разделить дерево возможно только по таксономии, а не по архитектурам, поэтому нельзя делать выводы о том какая архитектура появилась раньше.
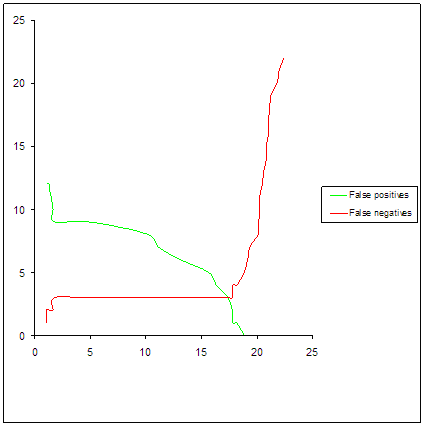
Для построения профилей я использовал два файла bac_input.msf (выбранные бактериальные последовательности) и eukaryo_input.msf(сначала использовал только выбранные последовательности, но затем расширил их число). Результаты поиска при помощи полученных профилей по исходным последовательностям – файлы result_bac.xls и result_eu.xls. Графики:
Бактериальный профиль:
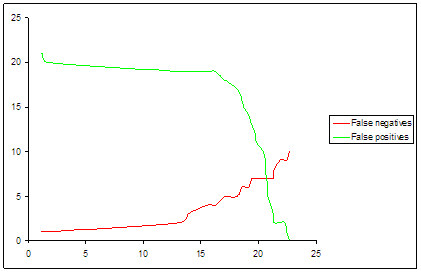
Эукариотический профиль:
Для бактериальных последовательностей получился хороший профиль. В качестве порога я выбрал значение, при котором сумма ошибок первого и второго рода минимальная (если суммы одинаковые, то выбираю минимумы каждого типа ошибок).
При поиске по Swissprot было найдено 700 бактериальных последовательностей – из них 11 не принадлежать ни к первой, ни ко второй архитектуре. Эукариотичские в выдачу не попали.
У эукариот нормальный профиль создать не получилось. При пороге 21.3 минимальное число ошибок первого и второго, однако при поиске по Swissprot найдено всего 5 последовательностей из них 3 эукариотических.
Вообще еще на этапе выравнивания была заметно большее число консервативных позиций у бактерий, по сравнению с эукариотами. Вероятно по ним и отсеиваются OBG эукариот при поиске бактериальным профилем, а эукариотический профиль их не содержащий, “пропускает” бактериальные последовательности.
статья