По заданию нам было дано дерево, в виде сценария эволюции нуклеиновой последовательности.
((A:80,B:80):20, (((C:20,D:20):10,E:30):80, F:80):10);
По этому сценарию, при помощи автофигур редактора Word, я построил графическую визуализацию дерева:

Затем мне было дано последовательность гена для которого были проведены последующие действия.
Его последовательность иожно най ти пройдя по следующей ссылке: BAA16466
Нами была проведена мутация данной последовательности. При мутации мы учитывали что:
Мутация может пройти в любой аминокислоте
Мутация может пройти в обратном направлении
При повторении мутации два раза (или более) мы не получим одинаковые последовательности
Для проведения мутации мы воспользовались программой msbar пакета программ EMBOSS.
Для облегчения работы был создан следующий скрипт:
msbar BAA16466 mut1 -point 4 -count 138 -auto
msbar BAA16466 mut2 -point 4 -count 69 -auto
msbar mut1 A -point 4 -count 552 -auto
msbar mut1 B -point 4 -count 552 -auto
msbar mut2 mut3 -point 4 -count 552 -auto
msbar mut3 mut4 -point 4 -count 69 -auto
msbar mut4 C -point 4 -count 138 -auto
msbar mut4 D -point 4 -count 138 -auto
msbar mut3 E -point 4 -count 207 -auto
msbar mut2 F -point 4 -count 552 -auto
|
В этом скрипте каждая строчка - мутация соответствующей последовательности.
Для незнающих принципов работы UNIX'а:
msbar BAA16466 mut1 -point 4 -count 138 -auto
msbar - команда в UNIX'е для мутации последовательности
BAA16466 - мутируемая последовательность
mut1 - результат мутации
-point 4 -count 138 -auto - параметры по которым проводилась мутация
|
Более подробная информация об анализе прведённх мутаций вы можете найти здесь, также там, если постараться,
можно найти сравнение программ по построению деревьев.
Перейти на файл про сравнение деревьев (doc)
| Истинные расстояния в моей модели: число точечных замен на 100 нуклеотидов |
|  |
 |  |
| Матрица попарного сходства:среднее число совпадающих нуклеотидов на 100 позиций |
|  |
 | 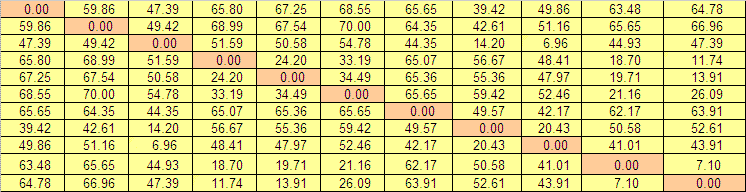 |
| Матрица попарных расстояний, вычисленных по методу Джукса - Кантора |
|  |
 |  |
Для того, чтобы определить наиболее подходящий метод для построения дерева было произведено сравнение
вышележащих таблиц (для этого был построен график матриц попарных растояний (сходств)):


