Учебный сайт Дюгая Ильи
Домены. Профили
Я исследовал домен с неизвестной функцией DUF559 (PF04480), он содержит 91 архитектуру (фрагмент описания см. рис. 1). Я рассматривал первую и вторую архитектуры. Далее "1" - архитектура с одним доменом PF04480, "2" - архитектура с одним доменом PF04480 и одним - PF13338.
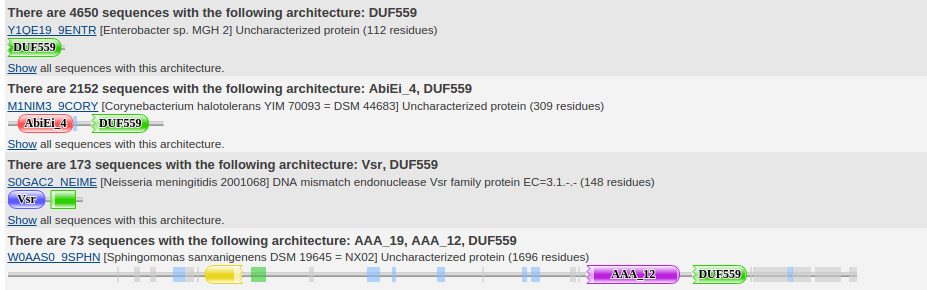
Рис. 1. Некоторые архитектуры, содержащие домен PF04480.
Я скачал с сайта PFAM полное выравнивание всех последовательностей домена. Проект выравнивания здесь. Далее я получил таблицу с доменной архитектурой всех последовательностей с моим доменом. Команда: "python swisspfam-to-xls.py -z /srv/databases/pfam/swisspfam.gz -m ac.txt -o PF04480.txt".
Из uniprot я скачал записи, соответствующие белкам с моим доменом (файл) и получил для них таксономию (команда: "python uniprot_to_taxonomy.py -i PF04480_uniprot.txt -o PF04480_taxonomy.txt").
С помощью ресурсов Excel и Pthon получил сводную таблицу, содержащую список последовательностей с указанием их доменной архитектуры, длины домена PF04480 и таксономии.
В качесте таксона я выбрал тип Actinobacteria, в качестве подтаксонов классы Micrococcales и Corynebacteriales. И выбрал 77 представителей этих подтаксонов, их последовательности вырезал из общего выравнивания, открыл в Jalview и удалил плохо выравненные повледовательности и небольшие участки с C и N концов. Проект выравнивания: Project_PF04480.jvp. Фаста: PF04480_seqs_final.fasta.
Затем по данному выравниванию в программе MEGA построил дерево (см. рис. 2) методов Neighbor Joining с 100 бутстреп репликами. Скобочная формула дерева:
(((((((((((('M_1_H0QQD8/28-91','M_1_A0JZN7/46-109'),('M_2_B8HDS1/36-99','M_2_F0MBC5/34-97')),'M_1_A9WRS7/36-99'),'M_1_A1RB14/43-107'),('M_1_H0QP99/46-109','M_1_B8H768/38-101')),('M_1_B8HDF6/34-98','M_2_A1R3N2/35-99')),('C_2_D0L3S2/12-77',('C_1_C0WFH9/29-87','C_1_E0MX03/28-86'))),'M_1_C7NJ62/8-72'),('M_1_B8HHV3/22-65',('M_2_E6SAC0/9-74',('M_2_A3TPI5/32-97','M_2_E6SDF2/23-82')))),(('M_1_C7MAX6/40-92','C_1_E4W8S8/14-77'),(('C_1_F5YZ79/13-76','C_1_A1TBP8/7-72'),('C_1_F5Z313/8-73',('C_1_I0PQG6/11-76','C_1_H0IUG1/12-77'))))),((('M_1_A9WMW6/8-67','M_2_F0M716/7-67'),'M_1_H0QH88/7-65'),('C_1_B1VIW1/5-65',(('M_1_H8E834/21-83','M_1_E8N6J6/2-46'),('M_1_H8E4Q9/27-91',('M_1_H8E268/22-85',('M_1_E8ND14/25-89','M_1_H8E171/43-107'))))))),((('M_1_E3BAQ3/5-69','M_2_C7R3L8/19-70'),('M_2_D3LLB7/10-59','M_2_C5C714/7-56')),((('C_1_G4HYA9/17-64','C_2_F6EJW0/12-74'),('C_1_I0RY54/19-83','C_1_I0RM27/12-65')),((('M_2_F6FTB7/10-75','C_2_D5PHS9/19-68'),('C_1_G4HW48/5-65','C_2_I0RD18/19-82')),(((('C_2_B1MIL3/24-83','C_2_I0PXH2/24-83'),'C_2_F5Z2E1/11-59'),('C_2_B1MN46/8-70','C_2_H0IMX9/8-70')),((('C_2_Q740U5/6-70','C_2_A1TF66/9-74'),'C_1_A1U957/14-71'),('C_2_I0RVG6/20-81',(('C_2_A2VPW6/12-63','C_2_P96837/12-63'),('C_2_A5U8M9/12-63','C_2_G0TJI7/12-70')))))))));
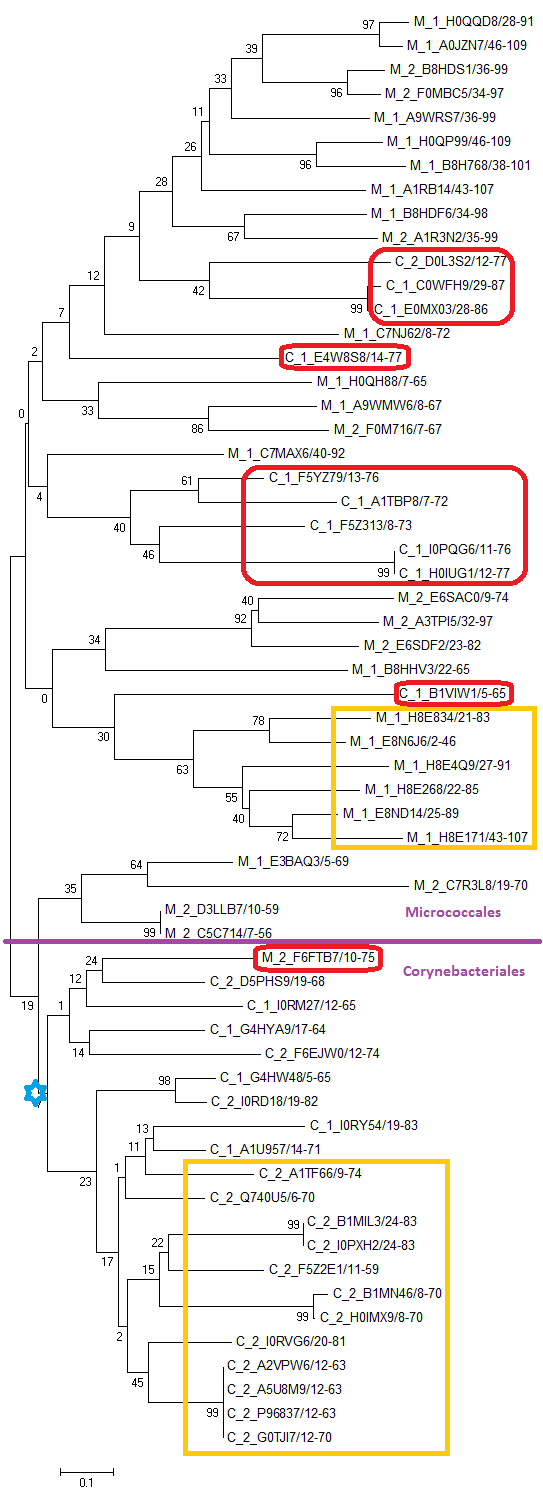
Рис. 2. Филогенетическое дерево домена PF04480. Построено методом Neighbor Joining.
На дереве "М" в названии последовательности означает, что она принадлежит Micrococcales, "С" - Corynebacteriales. Красными рамочками выделены выбивающиеся последовательности. Желтыми - клады подсемейств. Видно, что дерево можно попробовать разделить на 2 большие клады, соответствующие подсемействам, но при этом наблюдается дольшое количество выбивающихся последовательностей.
Я выбрал последовательности С_2_****** из клады, выделенной желтой рамочкой. Их выравнивание здесь. По ним с помощью программы hmm2biuld был построил профиль последовательностей, откалибровал hmm2calibrate. С помощью этого профиля провёл поиск по всем белкамс моим доменом с помощью программы hmm2search. Результат поиска можно увидеть здесь.
С помощью Excel построил ROC-кривую (рис. 3). Таблица с расчетами - PF04480_roc_1.xlsx.
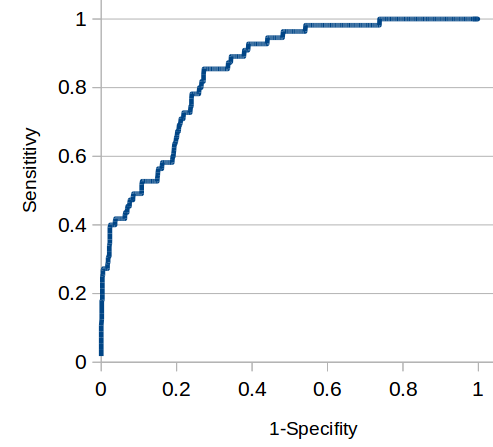
Рис. 3. ROC-кривая. Получена с помощью Excel.
Я выбрал порог E-value 1.30Е-04, при нём чувствительность составляет 71,4%, специфичность - 78,2%. Результаты при этом пороге - в таблице 1.
Таблица 1. Результаты поиска по профилю при выбранном пороге E-value 1.30Е-04
| На самом деле | принадлежит подсемейству | не принадлежит | сумма |
| Выше порога по профилю | 40 | 142 | 182 |
| Ниже порога | 16 | 510 | 526 |
| сумма | 56 | 652 | 708 |
Видно, что не удалось выбрать порог e-value с большой специфичностью и чувствительностью, профиль находит мало последовательностей подсемейства и довольно много ненужных нам последовательностей. Возможно это связано с тем, что выбранный домен довольно вариабелен.
Дата последнего обновления: 28.05.2014
Copyright © Дюгай Илья, 2013.