



 |  |  |  |
HMM профиль, ROC-кривая |
Вернуться на страницу семестра Задание 1. Определить целевое семейство белков. Составить список белков семейства из `SwissProt`Для работы был выбран домен Sec23/Sec24 trunk. Ссылка на страницу в pfam. AC Seq23_trunk. ID PF04811. С ним был связан предыдущий практикум, и дополнительную информацию о домене можно узнать из него. В базе данных Pfam HMM профиль любого домена можно скачать, но я наложу дополнительные условия - белок должен быть у представителя млекопитающих. Запрос в банке Uniprot следующий: database:(type:pfam id:PF04811) taxonomy:"Mammalia [40674]" AND reviewed:yes. Находок 14. Они вынесены на лист 1 окончательного файла Excel. Задание 2. Построение и откалибровка профиляИз Uniprot скачены fasta последовательности, в JalView они выровнены алгоритмом Muscle. Удалена 1 последовательность O94855. Ссылка на проект выравнивания. Рисунок 1. Выравнивание последовательностей
Пользуясь программами, установленными на kodomo, получаю из выравнивания профиль и калибрую его. Профиль: hmm2build profile.txt pr8_align.mfa - построение профиля. Калибровка: hmm2calibrate profile.txt - калибровка профиля. Полученный профиль - profile.txt Задание 3. Получение результатов поиска по профилю на множестве последовательностей SwissProtC помощью программы, установленной на kodomo, среди записей банка данных Swissprot были найдены последовательности, соответствующие построенному профилю. Замечание по работе программы:
по умолчанию отбираются находки с E-value < 10. Получится, что большинство находок правильные, всего несколько штук - возможно, неправильные. Для подбора порога надо чтобы неправильных
тоже было много - сравнимо с числом правильных. Поэтому порог E-value для доменов убран --domE 1000 (т.е. порог по E равен 1000). Снижен порог для веса --domT -50.
Задание 4. Выбор порога нормализованного веса для находок по профилю, ROC-кривая, гистограммаГистограмма весов находокПостроена гистограмма весов всех находок, хороших и плохих. Видим 2 ступеньки нормализованного веса (резкое падение) - позиция 7 и 19 - вес падает с 2122,3 до 1303 и с 1012 до 540 соответственно. Известны представители семейства и то, что они заканчиваются как раз на второй ступеньке. Поэтому порог будем считать 1012. Рисунок 2. Гистограмма весов находок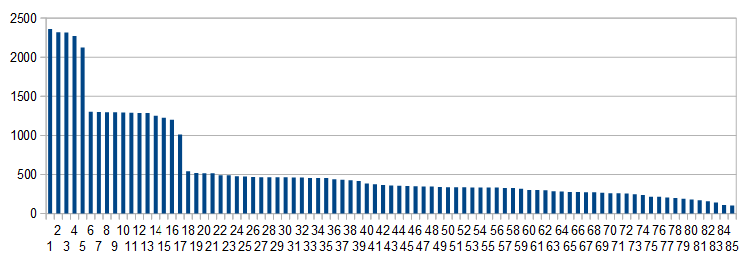
ROC-криваяROC-кривая (Receiver Operator Characteristic) показывает зависимость количества верно классифицированных положительных примеров от количества неверно классифицированных отрицательных примеров (SE от 1-SP). Определения TP, TN, FP, FN изображены на примере диагностики заболеваний на рисунке 3. Если развить пример с пациентами, то чувствительность (sensitivity) - это доля позитивных результатов теста в группе больных пациентов, а специфичность (specificity): доля негативных результатов теста в группе здоровых пациентов. Формулы - SE = TP / (TP + FN), SP = TN / (TN + FP). На основе вычислений, приведённых в файле ExcelРисунок 3. Пояснение к статистической обработке, в таком же порядке вычисления в Excel-файле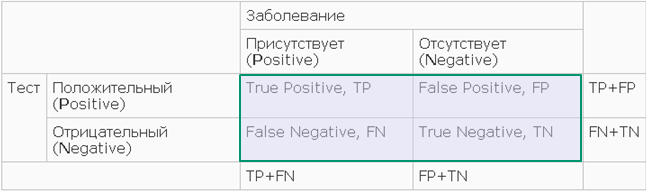
Рисунок 4. ROC-кривая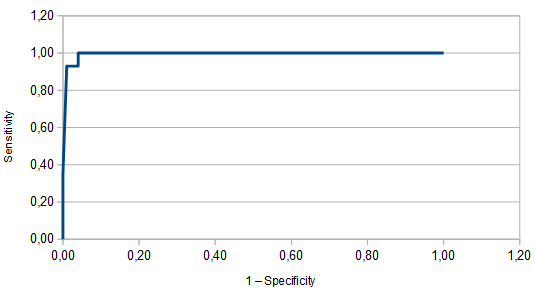
Файл Excel со всеми расчётами и таблицами. Вывод: профиль точен для определения принадлежности белка к семейству. Все белки из Swissprot были найдены и чтобы убрать излешек вводим порог 1012. Для него Sensitivity = 1, Specificity = 0,96, Precision = 0,96, Precision = 0,96 Выставленный порог веса позволяет точно выявлять белки искомого семейства и отвергать ложные находки: если вес >= порогу, то белок принадлежит семейству, если <, то гипотеза отвергается. Источники |