Практикум 7. Сигналы и мотивы (часть 2)

Практикум является продолжением работы с вирусом MERS, начатой в предыдущем практикуме. Мы строго подтверждали принадлежность найденных ранее мотивов классу TRS для двух штаммов - England 1 и референса HCoV-EMC.
England 1
Требовалось строго подтвердить, что полученный ранее мотив является TRS. Для этого мы сначала выполнили программу meme для 100b upstream sequences еще раз с параметром строгой длины мотива 6.
meme all_MERS_us.fasta -oc for_meme_5/ -dna -nmotifs 3 -w 6
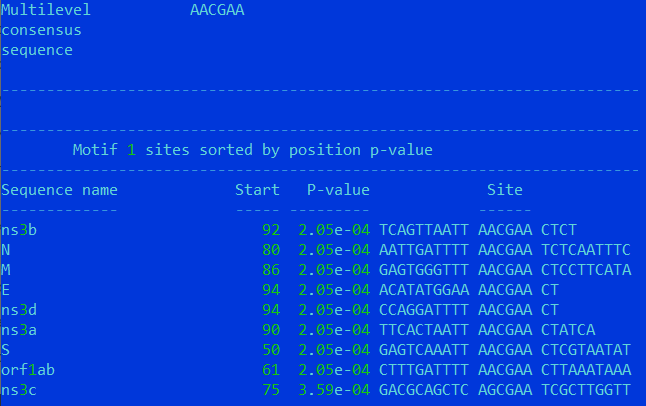
Подробнее вывод meme описывался в предыдущем практикуме.
Найденный нами мотив был в этом выводе, но он был "смещен" влево на 1 основание относительно известного мотива ACGAA[CT]. Остальные выведенные meme мотивы имели плохой evalue.
Потом мы воспользовались программой fimo, ищущей мотивы, найденные meme в небольших последовательностях, в геноме. Выполнили ее для нашего мотива в геноме выбранного коронавируса (штамм England 1) (первого по выдаче мотива в meme) с ограничением на pvalue не более 0.0004 и только на прямой цепи.
fimo --motif 1 --norc --verbosity 1 --output-pthresh 4.0E-4 for_meme_5/meme.html MERS_genome.fasta
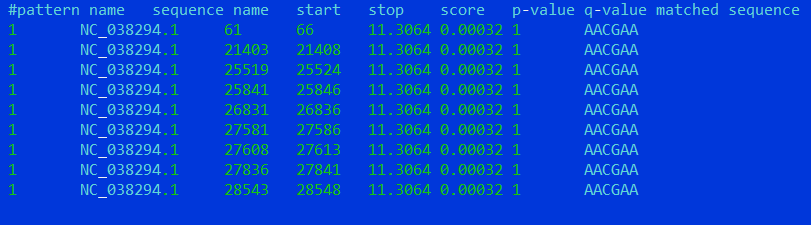
Вывод FIMO подтвердил предположение о том, что найденный мотив является TRS. Из свойств идеального мотива: встретился перед всеми генами вируса, включая orf1ab и поздние гены, длина равна 6, между сигналом и старт-кодоном нет ORF (просмотрено вручную), нет других сигналов, соответствующих мотиву (мы так подобрали pvalue). Вероятность встретить такой мотив случайно мала: вероятность вычисляется на основе распределения Пуассона для разных отрезков генома по схеме:
Параметр лямбда можно высчитать как матожидание найти такой мотив в случайной нуклеотидной последовательности данной длины (для конкретного слова длины n: N/4^n). Исходя из дальнейших ограничений высчитываем POIS (0) (в данном отрезке не встретилось последовательности; для последовательностей генов) или POIS (1) (встретилось ровно 1 раз; для upstream regions). При перемножении наверняка получается очень маленькое число.
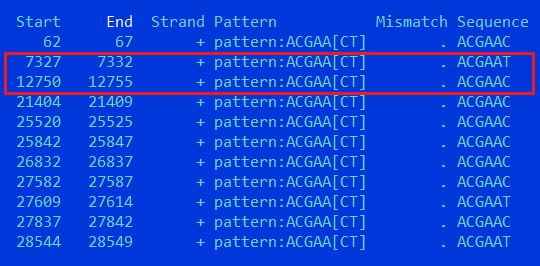
Признаемся, что когда мы решили проверить встречаемость консенсусного мотива (добытого из статьи, указанной в предыдущем практикуме) ACGAA[CT] программой fuzznuc:
fuzznuc -sequence MERS_genome.fasta -pattern ACGAA[CT] -outfile my_pattern.fuzznuc
то программа нашла еще несколько фрагментов генома, где встречается подобный паттерн (внутри гена orf1ab).
HCoV-EMC
Те же действия мы повторили для другого штамма (Human betacoronavirus 2c EMC/2012).
Сначала мы запустили программу meme со строгим ограничением на длину мотива, используя файл all.fasta, получение которого было описано в предыдущем практикуме. Длина мотива 6 была выбрана исходя из того, что в статье Zuniga et al., Journal of Virology, 2004 указывается, что это его свойство (длина 6 соответствует самой консервативной части - core sequence).
meme all.fasta -dna -nmotifs 3 -w 6
Мотивом с лучшим E-value (4.0e-003) был фрагмент AACGAA большого мотива TTAACGAACT, найденного в предыдущем практикуме. p-value для восьми генов 2.04e-04, для гена NS4B 5.24e-04, для orf8b 1.04e-03.
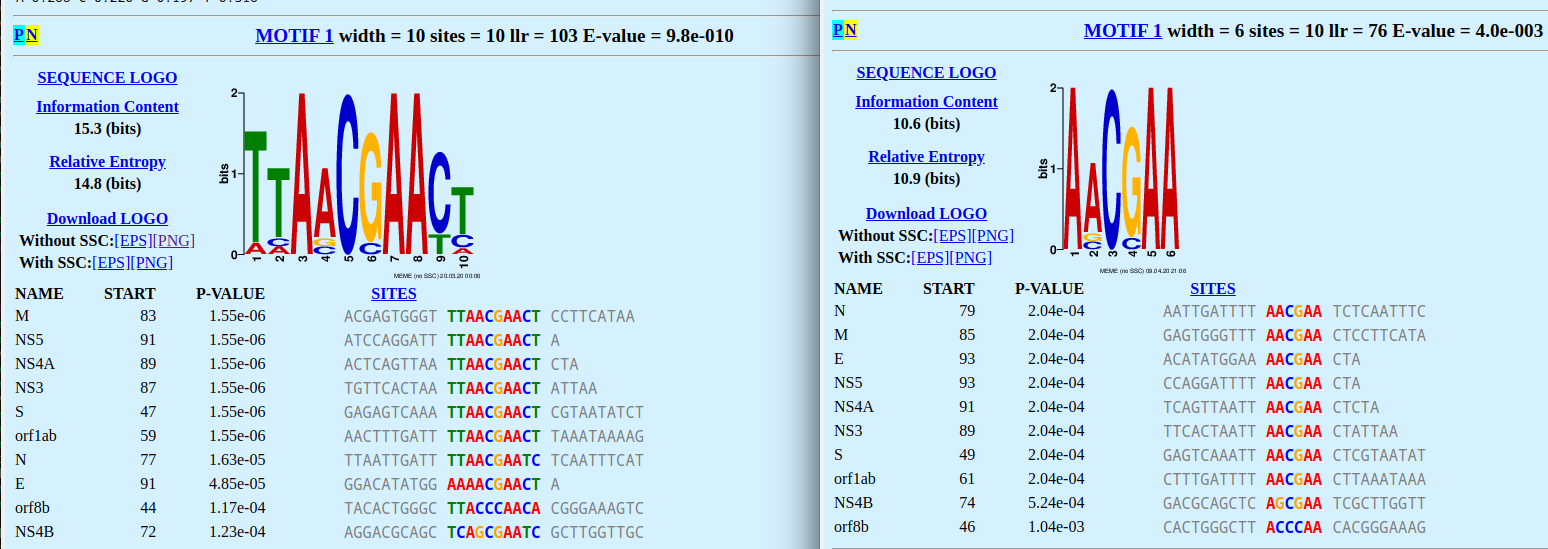
Два других мотива (CGAGCT и CAGGGT) имели плохие Evalue (5.8e+003 и 8.4e+004 соответственно).
Далее для всего генома штамма была запущена программа fimo (find individual motif occurences) пакета meme с ограничением на p-value. Она искала 3 мотива, найденных meme в коротких вырезанных последовательностях, по всему геному.
$ fimo --motif 1 --norc --verbosity 1 --output-pthresh 4.0E-4 meme_out/meme.html sequence.fasta
В одном из полученных файлов - fimo.html - в секции HIGH-SCORING MOTIF OCCURENCES можно увидеть таблицу:

Видно, что именно первый мотив, то есть консервативный участок мотива, предполагаемого в 6 практикуме как сигнал TRS, найден fimo 9 раз в геноме штамма.
Для построения LOGO последовательности Козак поздних генов коронавируса мы вырезали фрагменты -9..4 для каждого из них. Для этого, как и в предыдущем практикуме, скачали полный геном вируса в формате fasta, удалили первую строчку, слили все строки в одну строку с помощью xargs, удалили пробелы и дальше вырезали так. Если ген имеет начальную координату 279, то мы вырезали кусок от 270 (279-9=270) до 283 (279+9=283) нуклетида, то есть в команде cat 270-284, так как она не учитывает левую границу:
$ cat sequence2.txt | cut -c 270-284 > orf1ab.fasta
Для всех генов были выполнены такие команды:
$ cat sequence2.txt | cut -c 270-284 > orf1ab.fasta $ cat sequence2.txt | cut -c 21447-21461 > S.fasta $ cat sequence2.txt | cut -c 25523-25537 > orf3.fasta $ cat sequence2.txt | cut -c 25843-25857 > orf4a.fasta $ cat sequence2.txt | cut -c 26084-26098 > orf4b.fasta $ cat sequence2.txt | cut -c 26831-26845 > orf5.fasta $ cat sequence2.txt | cut -c 27581-27595 > E.fasta $ cat sequence2.txt | cut -c 27844-27858 > M.fasta $ cat sequence2.txt | cut -c 28557-28571 > N.fasta $ cat sequence2.txt | cut -c 28753-28767 > orf8b.fasta
Далее был создан файл mylist c названиями файлов, файлы были слиты в один командой
seqret @mylist all.fasta
Meme был запущен командой
meme all2.fasta -dna -nmotifs 3 -minw 14
Полученное LOGO:
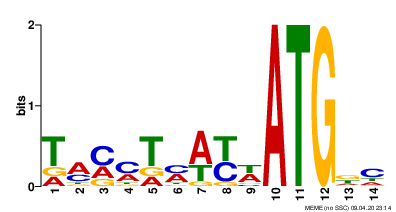
LOGO для человека (https://wikipedia.org/wiki/Консенсусная_последовательность_Козак):
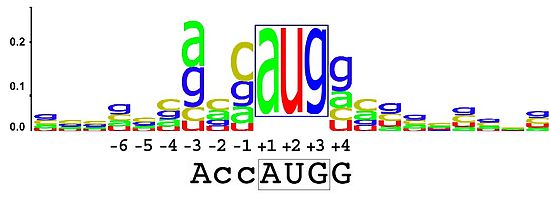
Повышенная встречаемость G сразу после старт-кодона и повышенная встречаемость A на 2 нуклеотида ниже старт-кодона у вируса чем-то напоминают последовательность у человека.
Кто выполнил работу
Елена Птицына выполнила строгое подтверждение принадлежности мотива классу TRS для референса, построила LOGO для последовательности Козак в референсе и сравнила его с LOGO для человека.
Елизавета Зайцева строго подтвердила принадлежность мотива классу TRS для штамма England 1 и разместила отчет о работе на страничке.









