Построение дерева по нуклеотидным последовательностям
Для рассмотренных в предыдущих практикумах бактерий были получены последовательности РНК малой субъединицы рибосомы (16S rRNA). Для этого использовась база данных полных геномов NCBI[1], из которой были извлечены файлы [frn], содержащие последовательности тРНК и рРНК. Создание единого файла, в который входят именно необходимые нам 16S рРНК последовательности, было автоматизировано при помощи cкрипта на Python. Он извлекает по заданному списку мнемоник нужные последовательности из соответствующих файлов [frn] и записывает их в объединенный файл формата [fasta].
Выравнивание нуклеотидных последовательностей выполнялось при помощи онлайн-сервиса Muscle[2], после чего построенное выравнивание переводилось в [fasta]-формат и методом Analyze открывалось в программе MEGA.
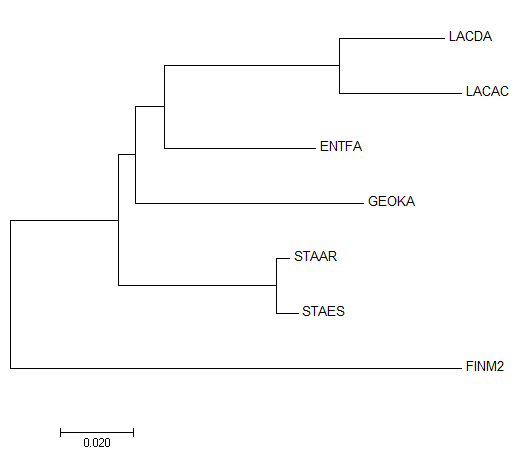
Для построения дерева в MEGA был выбран метод Neighbor-Joining, результат работы которого приведен на Рис. 1. Для сравнения с эталонным деревом, изображенным на Рис. 2, также приведены соответствующие нетривиальные ветви каждого дерева.
Построенное по нуклеотидной последовательности дерево обладает четырьмя нетривиальными ветвями, из которых три соответствуют аналогичным ветвям на эталонном дереве. Для ветви, создающей различие данных деревьев, разбиение выделено оранжевым цветом.
Рис. 1. Дерево, построенное на основе 16S рРНК методом NJ
 |
|
Рис. 2. Эталонное дерево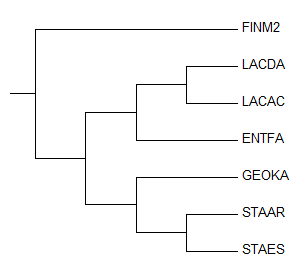
|
|
В целом, сравнивая дерево с тем, что было получено в ходе выполнения Практикума 3, стоит отметить приближение топологии дерева к эталонной: при использовании белковых последовательностей дерево вообще получалось неразрешенным, что не позволяет адекватно провести его сравнение с эталонным. Здесь же эта проблема не возникла, и, хоть это и можно было бы списать на случайность, на самом деле можно уверенно говорить о том, что построение деревьев на основе 16S рРНК является более надёжным методом реконструкции. Это связано с повышенной консервативностью соответствующих генов рРНК, что и позволяет в конечном итоге более точно определить систематическое положение организмов и оценить время расхождения близких видов.
Построение и анализ дерева, содержащего паралоги
Файлы, относящиеся к данному заданию, находятся в директории emkeller/term4/.
В данном задании требовалось построить дерево гомологов белка CLPX_BACSU[3], имеющихся у рассматриваемых бактерий. Этот белок является АТФ-связывающей субъединицей Clp-протеаз и выполняет функцию фактора специфичности, обеспечивающего соединение протеазы с нужным субстратом[4]. В этом качестве зачастую, помимо белков с неправильным фолдингом, выступают белки, помеченные ssrA-хвостом в результате процесса транс-трансляции и, таким образом, подлежащие деградации. В отсутствие протеолитической субъединицы (CLPP) CLPX также может выступать в роли шаперона.
Из файлов, содержащих полные протеомы исследуемых бактерий, был собран единый файл proteomes.fasta. На его основе далее была создана база данных db.fasta:
makeblastdb -in proteomes.fasta -out db.fasta -dbtype prot
Далее был запущен blastp последовательности белка CLPX_BACSU.fasta по этой базе данных с порогом evalue = 0.001. Вывод осуществлялся в текстовый файл res.txt:
blastp -query CLPX_BACSU.fasta -db db.fasta -evalue 0.001 -out res.txt
Выдачу blastp можно видеть на Рис. 3.
Рис. 3. Находки blastp
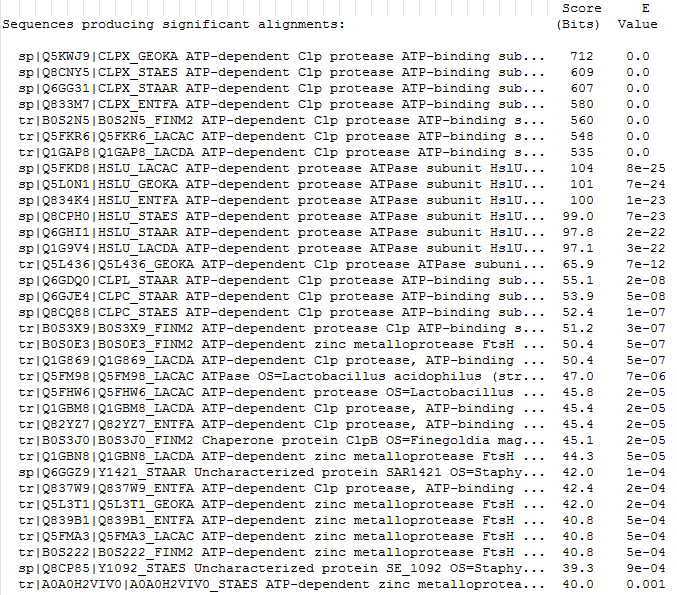
После этого для удобства представления результатов и дальнейшей работы с ними был создан файл [xlsx], и из него извлечены мнемоники белков, соответствующие найденным гомологам. Также на отдельном листе составлена таблица, наглядно представляющая группы мнемоник, соответствующих одной и той же функции белка.
Рис. 4. Функции белков-гомологов
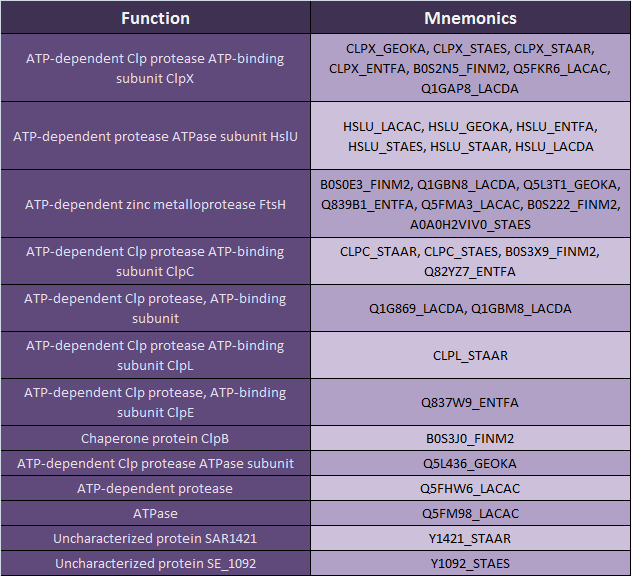
Список, включавший в себя 34 мнемоники, был использован для получения последовательностей белков средствами Jalview. Далее 34 полученные последовательности были сохранены в отдельный файл и затем выровнены средствами веб-сервиса Muscle[2].
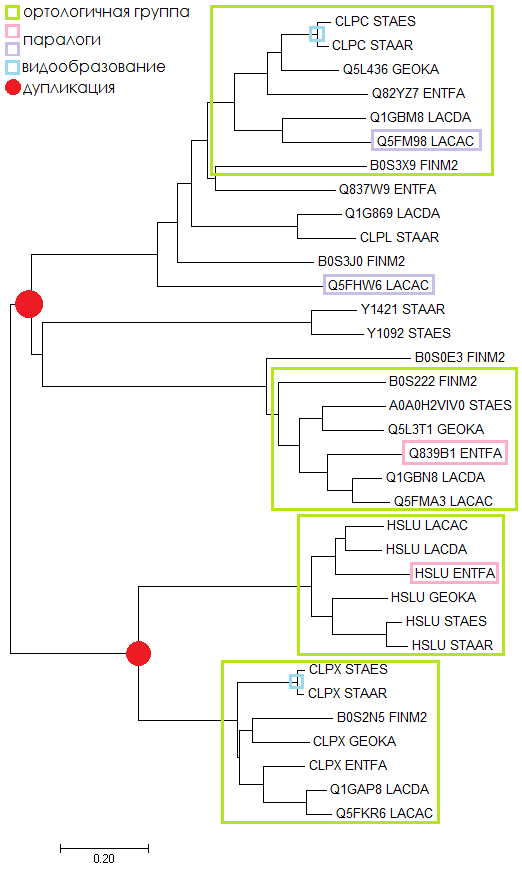
Для построения дерева был использован метод Neighbor-Joining в программе MEGA. Полученное дерево приведено ниже на Рис. 5. Также показаны ортологичные группы, пары паралогов в двух разных организмах, некоторые примеры актов видообразования и дупликаций, произошедших в ходе эволюции.
Рис.5. Паралоги, ортологи и эволюционные события на древе гомологов
[1] NCBI Genomes
[2] MUSCLE Web Service
[3] Uniprot: CLPX
[4] Uniprot: CLPP