МНОЖЕСТВЕННЫЕ ВЫРАВНИВАНИЯ |
Назад |
В данном занятии будут предприняты попытки найти, выровнять и частично проанализировать гомологи белка YokD_BacSu
Для начала я провёл поиск при помощи программы BLASTP среди эукариот и прокариот.
Подробную информацию по поиску гомологов среди эукариот и прокариот вы можете просмотреть в сохранённых отчётах о поиске:
bact.search; euk.search
В следующей таблице (1) находится краткая информация о финальных запросах, которые были составлены после серии гораздо менее точных прикидочных:
| Поиск | Алгоритм BLAST | Название базы данных | Ограничения по таксонам | Порог e-value | Максимальное количество хитов |
| По прокариотам | BLASTP 2.2.28+ | nr | exclude:Firmacutes exclude: Eukarya |
1x10-10 | 250 |
| По эукариотам | BLASTP 2.2.28+ | nr | Eukarya | 6x10-51 | 10 |
| табл.1 – параметры финальных поисков | |||||
Я выбрал такие достаточно узкие рамки поиска, поскольку прикидочные поиски выдавали слишком много результатов, не имеющих однозначного сходства с исходной последовательностью, например, имели высокий вес и е-value меньше единицы, но слишком маленькую идентичность, или же идентичность только по одному блоку, или же были высоко идентичны YokD_BacSu, но были гораздо короче, или же имели слишком много посторонних вставок.
NCBI, на котором я и пользовался сервисом BLAST, предоставляет возможность выстраивать филогенетическое древо гомологов белка (см.фиг 1-3). При построение деревьев отличие от YokD_BacSu было установлено на уровне не больше 85%.
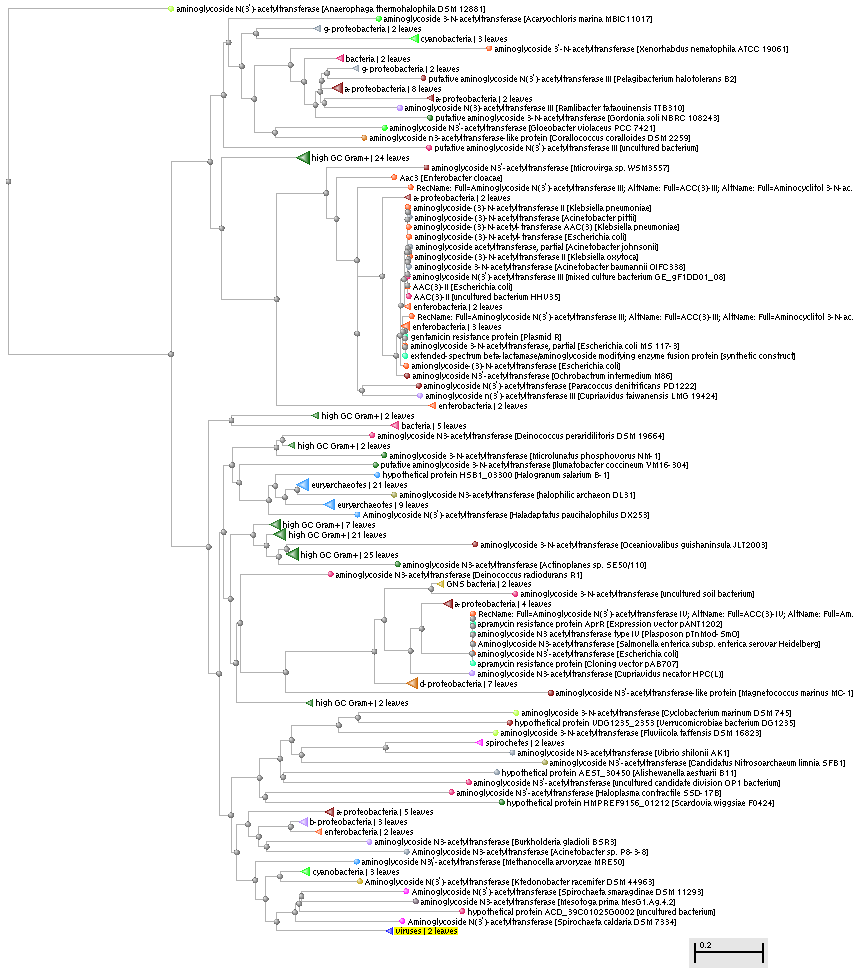 |
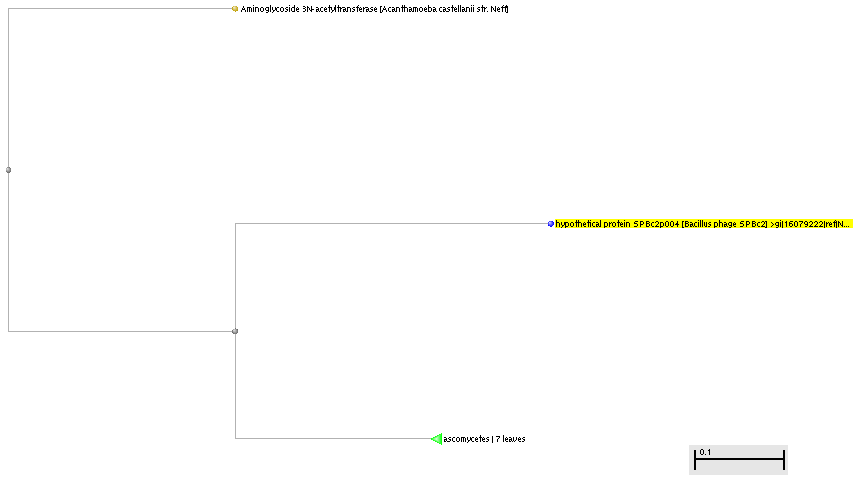 |
| фиг.1 – филогенетическое древо бактериальных гомологов | фиг.2 – филогенетическое древо эукариотических гомологов |
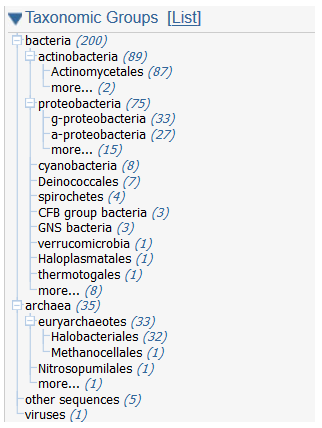 |
|
| фиг.3. – поиск гомологов по таксонам, цифрами обозначены количество находок в данном таксоне |
|
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
По следующей ссылке расположено множественное выравнивание гомологов из всех вышеприведённых организмов. Ниже на фиг.4 приведёно изображение этого выравнивания. 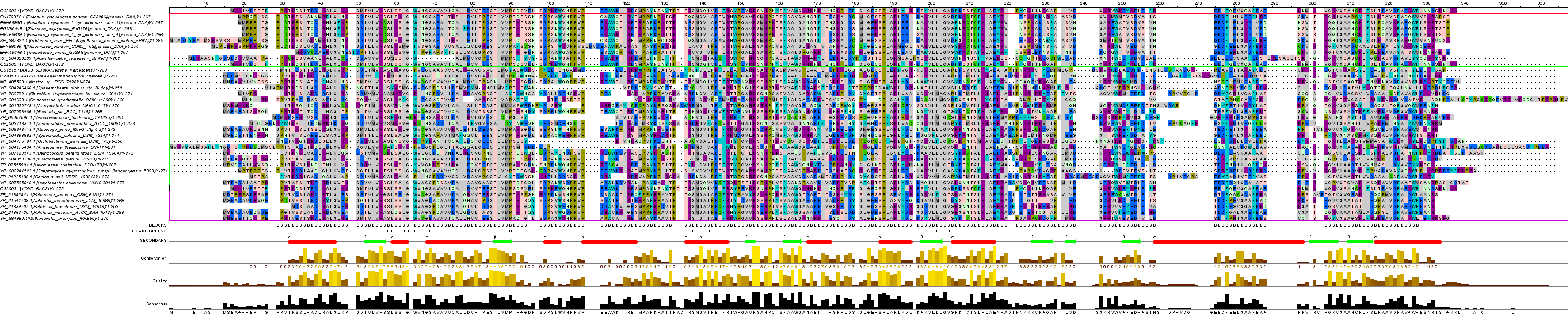 фиг.4 – множественное выравнивание YokD_Bacsu c гомологами (щёлкните на любое изображение, чтобы открыть его в этом окне) Легенда: алифатические неполярные аминок-ты - серый (метионин - тёмно-серый) ароматические - бирюзовый алифатические полярные незаряженные - дымчатый розовый отрицательно заряженые - синий положительно - фиолетовый цистеин - зелёный пролин - оливковый |
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Рассмотрение множественного выравнивания гомологов YokD_BacSu |
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
Как нетрудно заметить, в целом выравнивание высококонсервативно (см.фиг.5-7). Можно увидеть, что АО в тех же позициях, в которых они связывают лиганд
у YokD_BacSu, довольно консервативны. Причём, если не соблюдается идентичность лиганд-связывающих АО, то в примерно половине таких случаев происходит
замена на АО со сходными свойствами: Ser на Cys (№57 в выравнивании), Val на Ile (66). В позиции 141 в YokD_BacSu стоит Gln, что не вполне нормально
для этой позиции. Впрочем, если учесть, что лиганд COA находится в гидрофобном "чехле", АО которого суперконсервативны, то можно предположить, что
именно благодаря нему и возможны некоторые перестройки сайте связывания СОА. Таким образом, все гомологи в выборке должны также свзявать лиганд.
Сайт связывания СОА см.задание первого семестрана и фиг.8.
фиг.5 – выравнивание с порогом идентичность 34% (при наведении курсора изображения меняют вид) фиг.6 – выравнивание с порогом идентичность 53% фиг.7 – выравнивание с порогом идентичность 100% 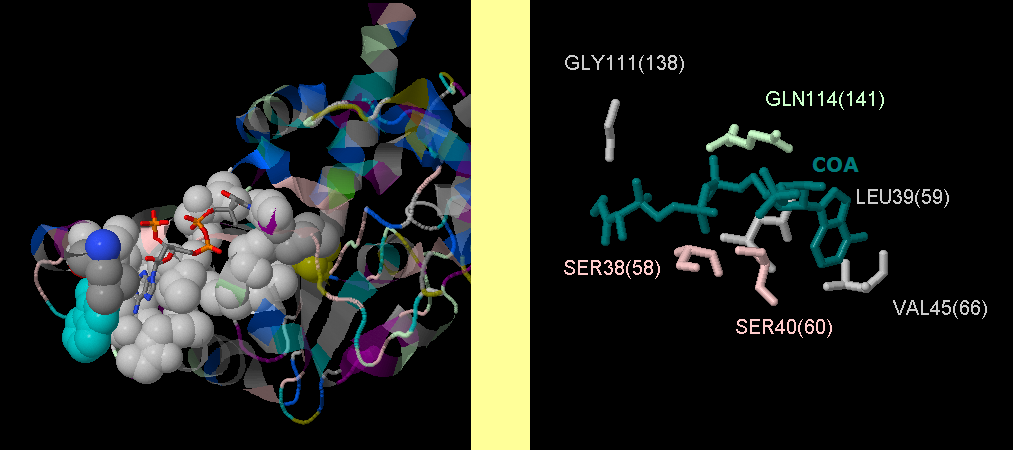 фиг.8 – слева гидрофобный "чехол", справа расположение лиганд связывающих остатков вокруг СОА, в скобках – номер АО в выравнивании |
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
Наиболее различны начала и концы белков, что естественно, поскольку они меньше всего влияют на общую структуру белка. Колонки пробелов приходятся в основном на участки, свободные от участков со вторичной структурой в YokD_BacSu. Что интересно, у нескольких белков какие-то некрупные потенциальные участки со вторичной структурой вообще отсутствуют. В целом же структура гомологов не должна значительно отичаться от YokD_BacSu. |
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||