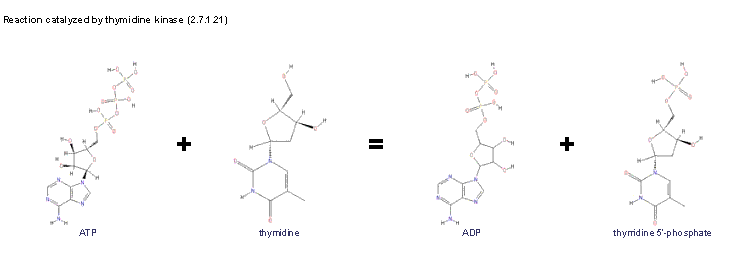
Было найдено 180 ферментов с таким кодом.
А вот полученная схема ферментативной реакции:

На этой странице приведено полное описание фермента тимидинкиназы
(с кодом 2.7.1.21).
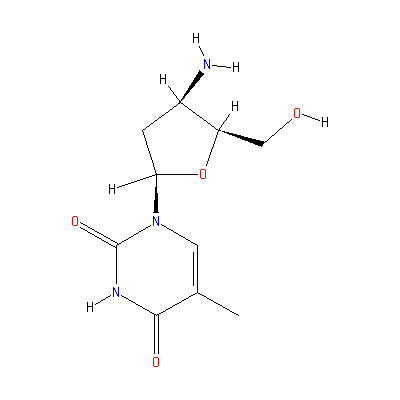 Это 2'-деокси-3'-аминотимидин.
Это 2'-деокси-3'-аминотимидин.
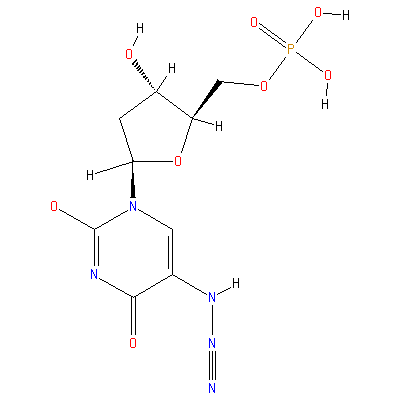 Это 5-ацидо-2'-деоксиуридин-5'-монофосфат.
Это 5-ацидо-2'-деоксиуридин-5'-монофосфат.
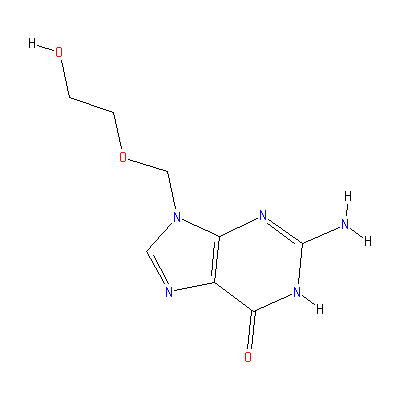 Это ацикловир.
Это ацикловир.
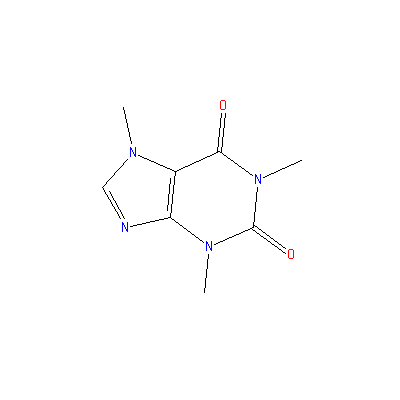 Это кофеин.
Это кофеин.
Это заболевание вызывается ретровирусом — ВИЧ. Наблюдения показали, что
содержание тимидинкиназы возрастает при развитии заболевания. Из этого делается
вывод, что исследование этого фермента может помочь в поиске средств, которые
будут способствовать лечению этого смертельного (на данный момент) заболевания.
Подробнее об этом можно прочитать здесь.
Вирус герпеса, как известно, встречается примерно у 90% людей и обитает в
клетках нервной системы человека. При ослабленном иммунитете его наличие в
организме проявляется в виде высыпаний на коже (в основном на носу и на губах).
При наличии вируса герпеса очень часто выявляется мутантная форма фермента
тимидинкиназы. Следовательно, наличие вируса в организме может как-то влиять на
работу этого фермента. И наоборот: большое количество мутантных форм может
уменьшать способность организма к борьбе с вирусом. Но это всего лишь гипотезы,
а ссылка на материалы находится здесь.
Некоторые гормоны (инсулин, глюкагон, глюкокортикоиды) регулируют экспрессию фосфоенолпируваткарбоксикиназы — регуляторного фермента. В статье сообщается о том, что ретиноевая кислота также может регулировать экспрессию этого фермента. Об участии тимидинкиназы в этих процессах можно посмотреть здесь.
Было обнаружено, что недостаток некоторых витаминов (к примеру, В12) ингибирует синтез тимидилата. Описана сложная цепочка реакций, связанных с этим процессом, и найти это можно здесь.
Нужно было найти в SWISS-Prot ферменты с данным кодом из трёх организмов
— кишечной палочки Escherichia coli K-12, археи Methanococcus
jannaschii и человека. Поиск осуществлялся в системе SRS с помощью
следующего запроса:
([swissprot-ECNumber:2.7.1.21*] & (([swissprot-EntryName:*_Ecoli*] |
[swissprot-EntryName:*_Human*]) | [swissprot-EntryName:*_Metja*]))
В итоге было найдено 3 белка: один у кишечной палочки и два у человека. У организма Methanococcus jannaschii не было найдено фермента с кодом 2.7.1.21. Поэтому, чтобы получить эволюционно дальний как от бактерий, так и от позвоночных белок (для более эффективного сравнения), был проведён ещё один поиск по данному коду — среди архей (к которым и относится Methanococcus jannaschii). Структура запроса:
([swissprot-ECNumber:2.7.1.21*] & [swissprot-Taxonomy:Archaea*])
Было найдено три белка, из которых был взят один — KITH_PYRKO (организм
Pyrococcus kodakaraensis). Он исследовался дальше вместе с тремя найденными
ранее белками.
Для сравнения доменной структуры найденных белков был использован вариант
просмотра результата SW_InterProMatches. Его преимущество состоит в том, что он
позволяет увидеть домены в последовательностях.
Ниже в таблице приведены результаты сравнения доменов Pfam.
| ID белка | AC Number | Число доменов Pfam | Идентификатор Pfam | Положение домена в последовательности |
| KITH_ECOLI | P23331 | 1 | PF00265 | 2 — 186 |
| KITH_HUMAN | P04183 | 1 | PF00265 | 19 — 189 |
| KITM_HUMAN | O00142 | 1 | PF01712 | 113 — 257 |
| KITH_PYRKO | Q5JGT7 | 1 | PF00265 | 8 — 192 |
Теперь нам нужны аминокислотные последовательности описанных доменов для
получения их попарной идентичности. Последовательности получены с помощью
программы seqret пакета EMBOSS (начало и конец доменов были
указаны по "требованию" программы в ходе её исполнения). Сделано это было
следующим образом:
seqret sw:P23331 -sask seqret sw:P04183 -sask seqret sw:O00142 -sask seqret sw:Q5JGT7 -saskКак видно, три из четырёх белков являются гомологичными (и к тому же имеют одинаковый идентификатор Pfam). Проведём попарное выравнивание последовательностей доменов гомологичных белков. Для этого используем следующий скрипт.
Попарные идентичности, полученные в результате работы этого скрипта, сведены в одну таблицу.
| AC Numbers | P23331 | P04183 | Q5JGT7 |
| P23331 | 100% | 25.0% | 28.3% |
| P04183 | - | 100% | 36.0% |
| Q5JGT7 | - | - | 100% |
Далее можно посмотреть полученные выравнивания:
Kith_Ecoli и Kith_Human
Kith_Ecoli и Kith_Pyrko
Kith_Human и Kith_Pyrko
Попарная идентичность последовательностей получилась в примерном диапазоне
от 25 до 35%. Результат вполне приемлемый, если учесть, что рассматривались
очень далёкие организмы — кишечная палочка, архея и человек.