Для построения профиля я сохранил соответствующее выравнивание доменов, полученное в предыдущем задании в msf формате:
убираю символы возврата строки: noreturn -infile selected.msf -outfile selected2.msf добавление весов в выравнивание: pfw selected2.msf > weighted_selected.msf создание профиля: pfmake weighted_selected.msf /usr/share/pftools23/blosum62.cmp > my.prf
pfsearch -C 40 -f my.prf /srv/databases/uniprot/sprot_shuffled.fasta | sort -nr > selected_scores.txt команда нормализации профиля: pfscale scores.txt my.prf > my_scaled.prf
Команда поиска по профилю в SW pfsearch -C 200 -f my_scaled.prf /srv/databases/uniprot/sprot.fasta | sort -nr > selected_result_5.5.xlsПорог был выбран таким, поскольку порог в 5.5 давал очень много результатов (более 300 000). Полученный файл, вместе с графиком весов полученных находок
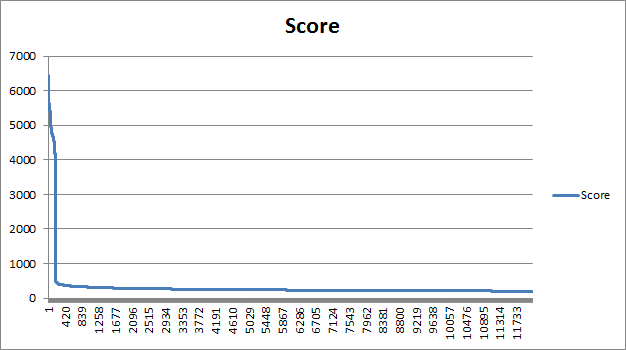
Построим график весов находок pfsearch, отсортированных по убыванию. Ступенька на этом графике можно интерпретировать как порог нормализованного веса для находок из семейства. Средствами Excel для полученного списка находок был построен график нормализованного веса находок, «ступеньку» на котором можно интерпретировать как порог нормализованного веса для находок из семейства.

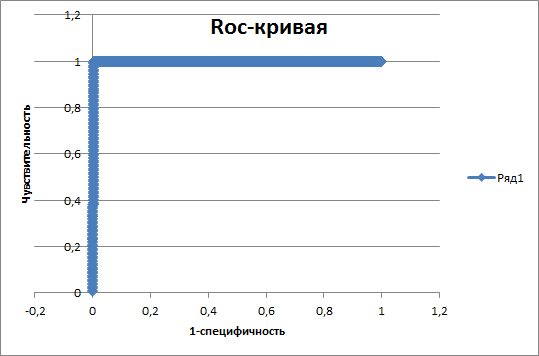
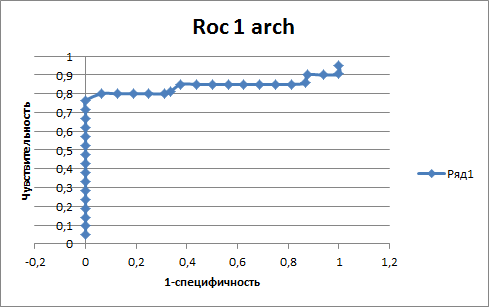
ROC-кривая представляет собой зависимость чувствительности алгоритма классификации (true positive rate, TPR) от величины FPR (false positive rate), которую можно обозначить как 1 – специфичность. Построенная в Excel ROC-кривая по полученному списку находок.
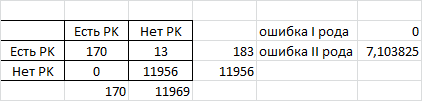
На основе данных, приведённых в данной таблице, можно установить порог нормализованного веса, равный 1233, который даёт 0 ошибок I рода (0%; это число соответствует вероятности не определить последовательность, содержащую домен PK) и 13 ошибок II рода ( 7%; это значение соответствует вероятности определить последовательность, не имеющую домена PK, как принадлежащую семейству).
Я решил разделить выравнивание по доменной архитектуре, поскольку на мой взгляд по полученному дереву нецелесообразно делить по таксономии организмов.
Первой архитектурой является один домен Glutaminase. Вторая архитектура - домен Glutaminase и Ank_2.
Поскольку остальные попытки построения профиля со второй архитектурой были неудачными, я добавил ещё последовательностей, чтобы улучшить выравнивание, и снова провел поиск по профилю:
1arch.msf
2arch.msf
Новое общее выравнивание
Как не трудно догадаться, выравнивание в файле 1arch.msf содержит все последовательности с первой архитектурой, а в файле 2arch.msf - все последовательности со второй архитектурой.
noreturn 1arch.msf 11arch.msf pfw 11arch.msf > 11arch_weighted.msf pfmake 11arch_weighted.msf /usr/share/pftools23/blosum62.cmp > 1arch.prf
Далее, проведем cам поиск по профилю в исходных последовательностях. Порог веса поставим маленьким (-C 0.0), чтобы все последовательности оказались в выдаче. Файл all_seq.fasta содержит все 33 последовательности.
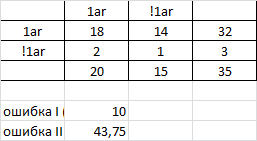
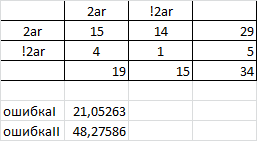
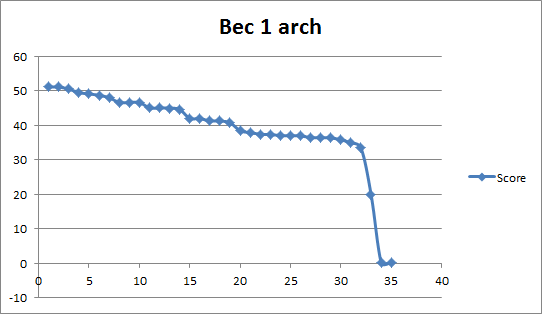
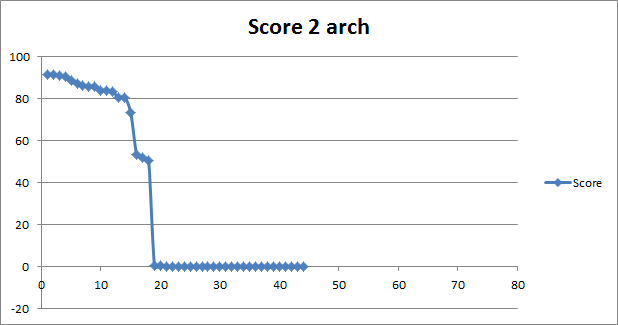
команды поиска по 1arch и 2arch профилям в исходных последовательностях pfsearch -C 0.0 -f 1arch.prf all_seq.fasta | sort -nr > 1arch.xls pfsearch -C 0.0 -f 2arch.prf all_seq.fasta | sort -nr > 2arch.xls Таблица для первой архитектуры Таблица для второй архитектуры
ROC-кривые:
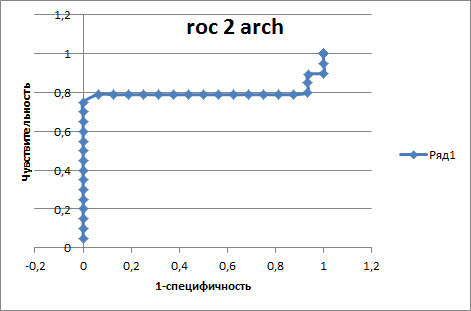
Основываясь на полученных графиках для весов, можно однозначно определить порог для 1 архитектуры - это 19. Для второй архитектуры порог вообще - 0.33. По таблицам видно, что ошибка первого рода очень высокая. Это говорит о том, что не имеет смысла применять подобный профиль для определения последовательностей с двумя разными доменными архитектурами так как результаты будут недостоверными.