Задание 1.Построить выравнивание представителей домена Pfam белков с разной доменной архитектуройЯ выбрала домен своего белка с идентификатором PF09279. Информация представлена по ссылке здесь
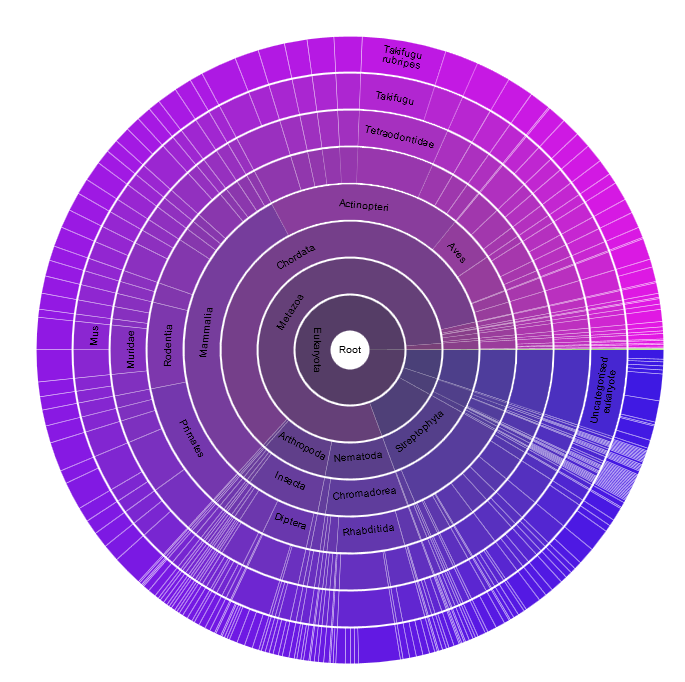
Рисунок 1.Принадлежность выбранного домена к разным таксономическим группам.Домен включает 64 доменные архитектуры. Его выравнивание представлено в программе JalView здесь.Выравнивание в фаста формате в файле Были выбраны 2 доменные архитектуры, которые представлены на рисунке 2.
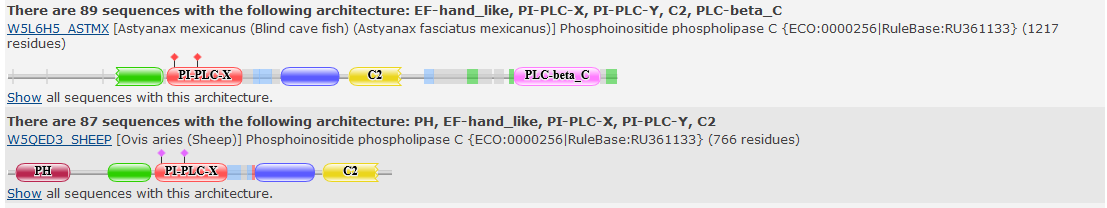
Рисунок 2.Выбранные доменные архитектуры, содержащие домен PF09279.Первая архитектура содержит 5 доменов - EF-hand_like, PI-PLC-X, PI-PLC-Y, С2, PLC-beta C terminal, вторая - 5 - EF-hand_like, PI-PLC-X, PI-PLC-Y, PH, С2. Далее получила таблицу с информацией о доменной структуре каждой последовательности, содержащей данный домен с помощью команды "python swisspfam-to-xls.py -z /srv/databases/pfam/swisspfam.gz -m domen.txt -o PF09279.txt". Командой "python uniprot-to-taxonomy.py -i uniprot.txt -o PF09279_taxonomy.txt" была получена таксономия для каждой последовательности. Данные представлены в сводной таблице здесь В качестве таксона был выбран Mammalia и 2 подтаксона Laurasiatheria и Euarchontoglires. Затем надо было выбрать последовательности, содержащих отобранные архитектуры. Далее были вырезаны их последовательности из общего выравнивания. Выраваниение выбранных последовательностей в Jalview здесь Выравнивание в фаста формате здесь
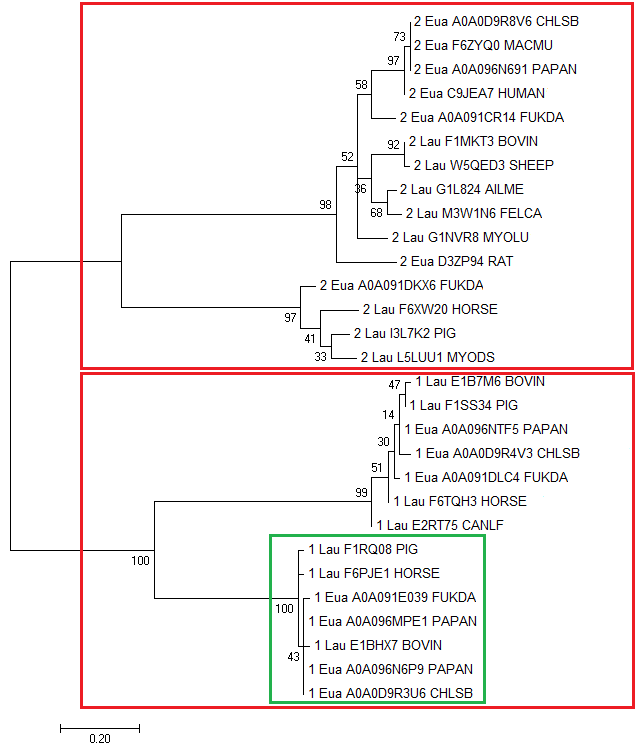
Задание 2. Построить филогенетическое дерево последовательностей ДОМЕНАНа основе полученного выравнивания было построено филогенетическое дерево, представленное на рисунке 3, при помощи программы MEGA. Метод построения дерева: метод максимального правдоподобия.
Рисунок 3.Филогенетическое деревоСкобочная структура дерева ссылка На рисунке 3 последовательности имеют особые названия. Цифра "1" означает, что последовательность имеет первую отобранную архитектурную структуру, цифра "2" - второй архитектуре. "Eua" означает принадлежность к Euarchontoglires, а "Lau" - Laurasiatheria. Как видно, дерево разделено на две четкие клады, которые заключены в красные рамки на рисунке 3. Мы хотели посмотреть, по какому принципу будет разделение на клады:по таксону или доменной архитектуре.На полученном дереве видно, что разделение произошло по доменным архитектурам. Задание 3.Построить профиль подсемейства и охарактеризовать качество его работы.Для построения профиля необходимо выбрать хорошее подсмемейство из выравнивания. Я выбрала последовательности, которые заключены в зеленую рамку на рисунке 3. Выравнивание отобранных последовательностей в этом файле С помощью программы hmm2biuld был построен профиль последовательностей. Программой hmm2calibrate он был откалиброван. Используя этот профиль, был проведен поиск по всем белкам программой hmm2search. Результат находится здесь
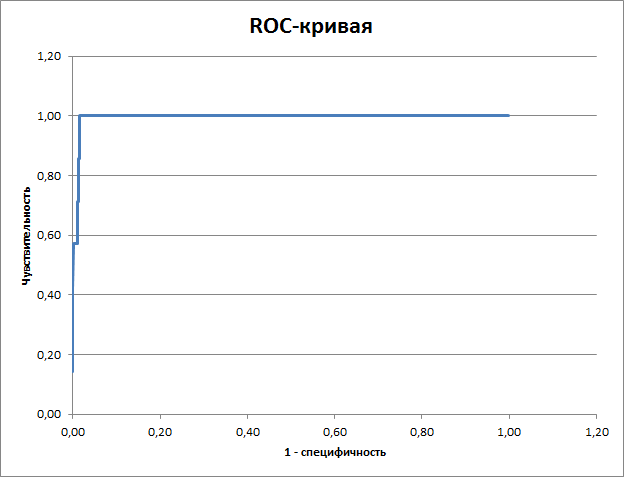
Данные были перенесены в Excel.Последовательности, которые были использованы для построения профиля были отмечены как "ИСТИНА", остальные - "ЛОЖЬ".Далее был построен график зависимости чувствительности от 1-специфичность.Результаты представлены в таблице Чувствительность поиска это доля достоверно предсказанных белков, содержащих домен к общему числу белков. Специфичность это доля достоверно предсказанных белков, которые не содержат домен к общему числу белков. Сам этот график есть ROC-кривая, которая представлена на графике 1.
График 1.ROC-криваяПри помощи ROC кривой был выбран порог e-value = 2e-79.
|