Выравнивание домена
Для выполнения проекта я выбрала семейство доменов Catalase (каталаза), так как в первом семестре я работала с каталазой. АС - PF00199, ID - Catalase. Каталаза отвечает за разложение пероксида водорода на кислород и воду и встречается практически во всех аэробных организмах. Согласно Pfam, этот домен содержится в 5410 последовательностях, в 2516 организмах, и образует 55 архитектур. Было скачано выравнивание всех последовательностей, содержащих домен. К одной из последовательностей, CATA_HUMAN, была найдена 3D структура, 1QQW. Все это можно увидеть в проекте Jalview (скриншот на рис.1).
Рис.1. Скриншот Jalview проекта.
Архитектуры домена
При помощи скрипта swisspfam-to-xls.py командой:
python swisspfam-to-xls.py -z /srv/databases/pfam/swisspfam.gz -p PF00199 -o PF00199.xlsбыла получена таблица с информацией oб архитектуре всех последовательностей, содержащих выбранный домен (лист "PF00199" в таблице). Чтобы узнать больше об архитектурах, была составлена сводная таблица (лист summary). Из нее можно узнать, что помимо собственно каталазы, в ее архитектурах часто встречается домен PF06628. Из Pfam узнаем, что это так называемый Catalase-related immune-responsive domain, который часто встречается в каталазах и несет октопептид, узнаваемый Т-лимфоцитами. Третий наиболее часто встречающийся домен - PF01965. Относится к семейству DJ-1/PfpI. По-видимому, это какие-то протеазы.
Вот 2 доменные архитектуры, содержащие данные домены (зеленый - catalase, красный - catalase-rel, фиолетовый - DJ-1/PfpI) :
 1. Эта архитектура соответствует 3816 последовательностям.
1. Эта архитектура соответствует 3816 последовательностям.
 2. Эта архитектура соответствует 512 последовательностям.
2. Эта архитектура соответствует 512 последовательностям.
Далее я скачала из Uniprot полные записи всех последовательностей. Получился файл uniprot-yourlist%3AM201705268A530B6CA0138AFAA6D2B97CE8C2A924D2A867G.txt.gz. Я его распаковала и применила скрипт uniprot-to-taxonomy.py:
gunzip uniprot-yourlist%3AM201705268A530B6CA0138AFAA6D2B97CE8C2A924D2A867G.txt.gz python uniprot-to-taxonomy.py -i uniprot-yourlist%3AM201705268A530B6CA0138AFAA6D2B97CE8C2A924D2A867G.txt -o taxonomy.xlsПолучился файл taxonomy.xls с таксономией всех последовательностей, который я скопировала в общую таблицу) на лист taxonomy.
Я выбрала таксон Dicarya и 2 субтаксона: Ascomycota и Basidiomycota. Я выбрала по 30 последовательностей из обоих субтаксонов (лист my taxon таблицы) и выписала их выравнивания в отдельные файлы с помощью скрипта filter-alignment.py:
python filter-alignment.py -i align.mfa -m a1.txt -o asco1.fasta -a "/" python filter-alignment.py -i align.mfa -m b1.txt -o basidio1.fasta -a "/" python filter-alignment.py -i align.mfa -m a2.txt -o asco2.fasta -a "/" python filter-alignment.py -i align.mfa -m b2.txt -o basidio2.fasta -a "/"a1.txt a2.txt, b1.txt и b2.txt - это списки выбранных последовательностей Ascomycota и Basidiomycota первой и второй архитектуры соответственно. Программа выдала 4 файла с выравниваниями, которые были слиты в один (можно было бы получить сразу 1 файл, но тогда было бы сложнее сортировать по субтаксонам и архитектурам). Чтобы можно было отличить субтаксон и архитектуру к каждой последовательности я приписала строчку вида А1, где буква - это таксон (А - Ascomycota, В - Basidiomycota), а цифра - номер архитектуры. Я выделила там группы по архитектурам, удалила плохо выровненные С и N концы и явные фрагменты, покрасила выравнивание Clustalx c консрвативностью 5% и сохранила проект.
Построение филогенетического дерева домена
Строилось дерево методом Neighbour Joining using PAM 250. С помощью сервиса ITOL дерево было визуализировано (рис.2).

Рис.2. Дерево последовательностей, содержащих домен catalase, укорененное в ветвь, разделяющую 2 архитектуры.
Как можно заметить, на дереве есть 2 хорошо выраженные ветви для архитектуры 1 и 2. В связи с чем можно предположить, что у общего предка Ascomycota и Basidiomycota существовали обе архитектуры.
Построение профиля подсемейства.
Я выбрала подсемейство из 10 белков, которые образуют кладу на дереве (самые верхние на рис. 2), и белки в нем имеют общую доменную архитектуру. Сохранив их в отдельном файле, я выстроила профиль используя пакет HMMER и откалибровала его.
hmm2build profile.txt profile.mfa hmm2calibrate profile.txtПолучился файл profile.txt. Затем был проведен поиск по профилю в Uniprot (в качестве базы данных был использован файл database.fasta, полученный из ранее скаченного uniprot-yourlist%3AM201705268A530B6CA0138AFAA6D2B97CE8C2A924D2A867G.txt):
seqret uniprot-yourlist%3AM201705268A530B6CA0138AFAA6D2B97CE8C2A924D2A867G.txt fasta::database.fasta hmm2search profile.txt database.fasta > found.txtПолучился файл found.txt со всеми находками, и их e-value. Я занесла их в новую таблицу Excel, и в отдельной колонке указала те последовательности, по которым строилось выравнивание. Далее на отдельном листе (ROC) я построила ROC-кривую (рис.3), а также нашла порог для score и evalue (порог в данном случае находился как такое значение, при котором сумма чувствительности и специфичности максимальна).
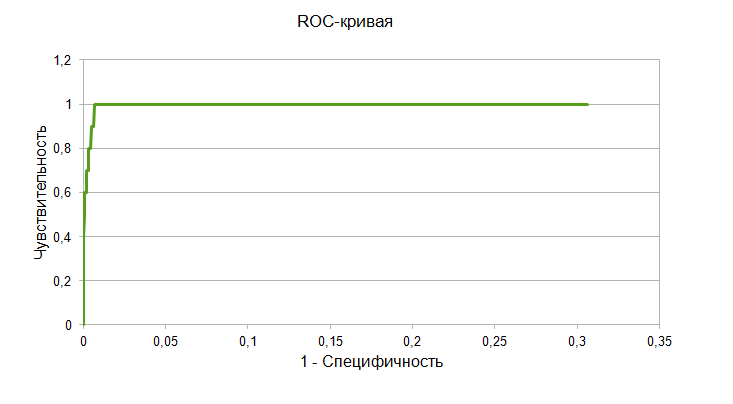 Рис.3. ROC-кривая для профиля. |
|