Практикум №10
Поиск нуклеотидных последовательностей
Поиск произвоидлся через сборный банк "Nucleotides" на сайте NCBI. Запроса было два - для каждого из организмов.
1.(Actinomyces naeslundii[Organism]) AND chromosome[Title], также был использован фильтр RefSeq. Первым выбранным организмом стала бактерия Actinomyces naeslundii strain CW; Accession: NZ_CP113787.1.
2.(Actinomyces [Organism]) AND chromosome[Title] , аналогично был использован фильтр RefSeq. По данному запросу я получил 36 результатов и выбрал один из них: Actinomyces trachealis strain zg-993; Accession: NZ_CP065027.1.
Dot-Plot
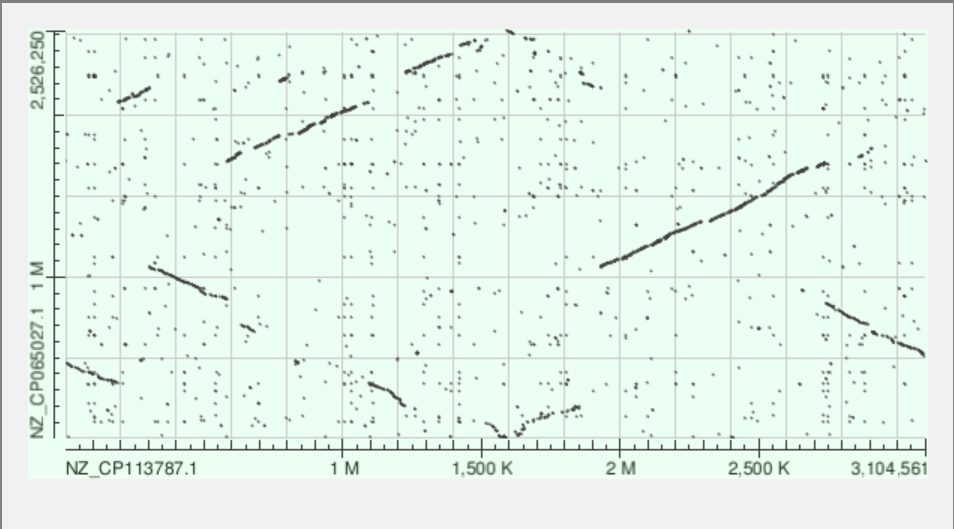
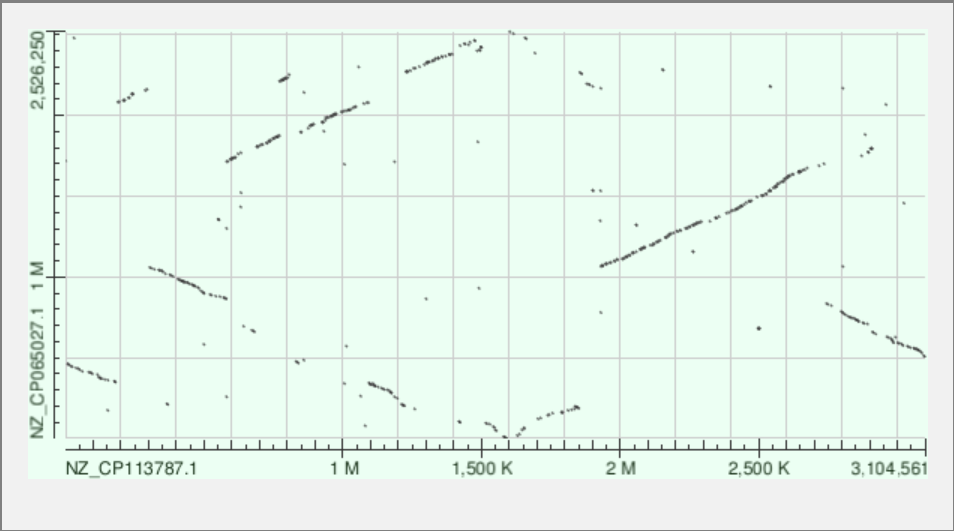
Параметры Blast взяты по умолчанию. Сравнивались два вида организмов одного рода Actinomyces. На картах dot-plot выдача двух различных алгоритмов Blast, а именно blastn и megablast.
Из выдачи megablast сделать какие-либо выводы сложно - разрывы и пропуски гораздо больше по сравнению с другим алгоритмом, потому что данные нуклеотидные последовательности не идентичны и имеют разную длину, а данный алгоритм лучше всего работает с практически одинаковыми последовательностями.
Из выдачи blastn видно, что на карте много единичных точек, это означает, что в последовательности много повторов. Также в верхне части карты видны зоны делеций и инсерций (небольшие разрывы в длинном участке, которые стоят не на одной линии). Вероятно, что имеются неконсервативные учатски - это такие же разрывы в длинных участках, которые далее продолжаются без сдвигов вверх и вниз. Blastn в данном случае более информативен, так как предназначен для поиска последовательностей, с меньшей идентичностью, каковыми и являются выбранные мной.