Практикум №7
1.Сравнение предсказаний трансмембранных участков в бета-листовом белке
В качестве исследуемого белка я выбрал Chitoporin - Хитопорин. PDB - 7vtz, UniProt - CHIP_ECOLI. Данный белок есть у организма Escherichia coli (strain K12), находится он в наружной мембране бактерий.
Координаты трансмембранных участков: 1( 9- 18), 2( 38- 48), 3( 57- 65), 4( 98- 110), 5( 113- 121), 6( 141- 148), 7( 156- 163), 8( 190- 196), 9( 203- 209),10( 217- 226),11( 232- 241),12( 261- 269),13( 276- 282),14( 324- 331),15( 343- 349),16( 376- 384),17( 395- 400),18( 424- 434)
Далее я сравнил координаты с координатами полученными при помощи DeepTMHMM, для этого я взял последовательность аминокислот в формате fasta из PDB: Beta sheet 12 20, Beta sheet 37 48, Beta sheet 56 67, Beta sheet 101 108, Beta sheet 113 120, Beta sheet 139 146, Beta sheet 156 164, Beta sheet 189 196, Beta sheet 203 210, Beta sheet 217 224, Beta sheet 234 241, Beta sheet 261 268, Beta sheet 275 282, Beta sheet 324 331, Beta sheet 341 348, Beta sheet 375 383, Beta sheet 393 401, Beta sheet 423 431.
Можно заметить, что практически все координаты соответсвуют (перекрываются) с погрешностью в +- 3 аминокислоты. Выдачу в текстовом виде со всеми участками (нас интересуют только "Beta sheet") можно посмотреть здесь.
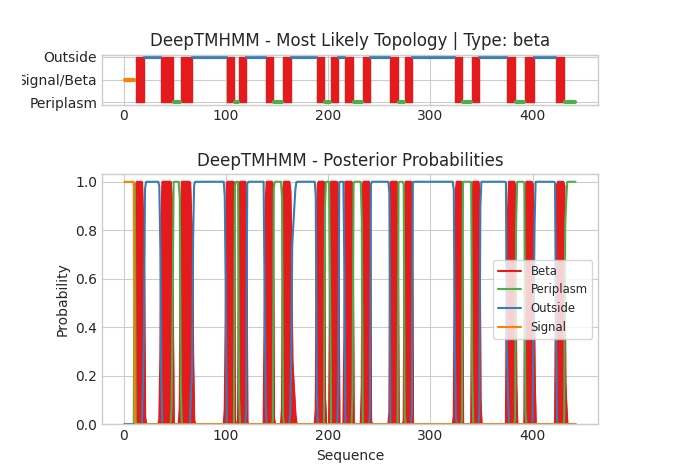
Верхний график указывает на координаты искомых участков. А нижний на вероятности предсказаний для каждой позиции, наши искомые позиции - покрашены в красный, для трансмембранных участков вероятности находятся в районе 0.95.
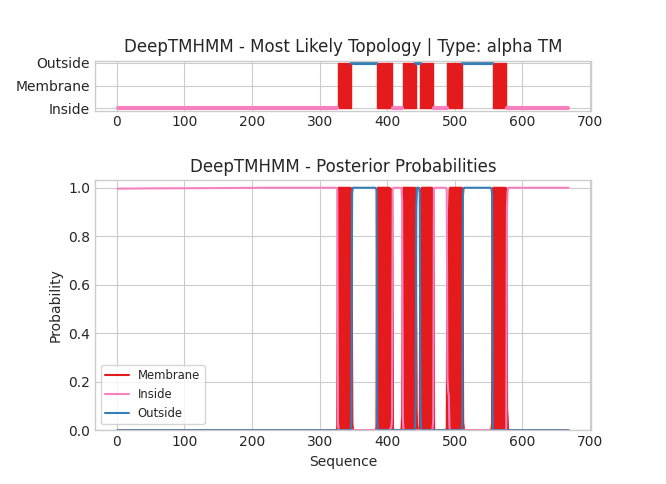
2. Сравнение предсказаний трансмембранных участков в альфа-спиральном белке
Мне был выдан белок TRPV6 channel, monomer, structure 3 - транзиторный рецепторный потенциальный канал ваниллоидного типа 6, PDB - 5wo6, UniProt - TRPV6_RAT. Данный белок находится в организме Rattus norvegicus. Данный белок участвует в трансклеточной абсорбции кальция в эпителиальных клетках.
Координаты трансмембранных участков: 1( 328- 347), 2( 382- 404), 3( 423- 444), 4( 450- 468), 5( 484- 507), 6( 555- 578)
Координаты, полученные из DeepTMHMM: TMhelix (327 347), TMhelix (385 407), TMhelix (423 442), TMhelix (449 468), TMhelix (489 511), TMhelix (556 576), полная выдача, мы смотрим на TMhelix.
Здесь ситуация такая же, как и с предыдущим белком, есть небольшая погрешность, но в целом все координаты были прдесказаны достаточно точно, с небольшой погрешность в +- 3 аминокислоты.