Сигналы. OriC
Основная информация
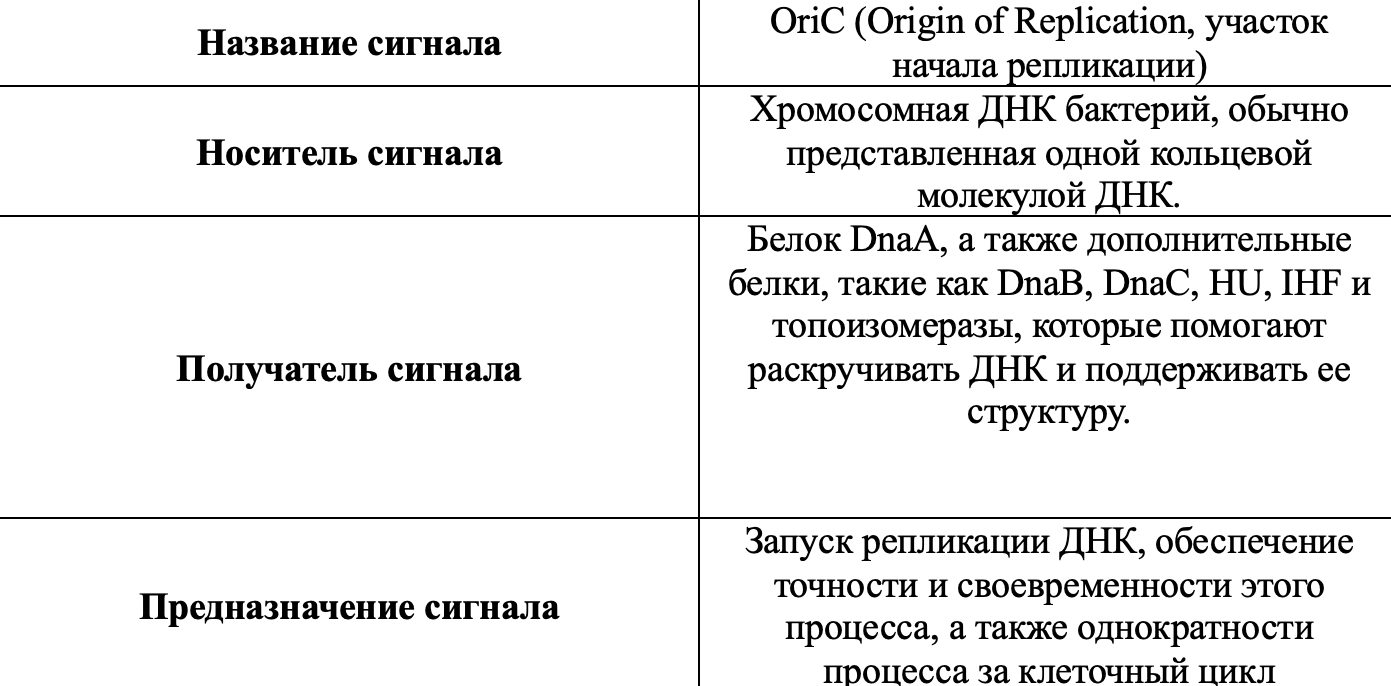
Структура
DnaA-боксы - Короткие последовательности ДНК (обычно 9 пар оснований), которые специфически связываются с белком DnaA. Например, у Escherichia coli консенсусная последовательность DnaA-бокса — 5’-TTATCCACA-3’ или немного от нее отличающиеся.
АТ-богатый регион - Участок, облегчающий раскручивание ДНК благодаря более слабым водородным связям между аденином и тимином. У E.coli АТ-богатый регион содержит три таких 13-мерных повтора.
Сайты связывания дополнительных белков - Например, белок IHF может связываться с OriC, помогая изгибать ДНК и облегчать доступ DnaA к ДНК.
Механизм действия
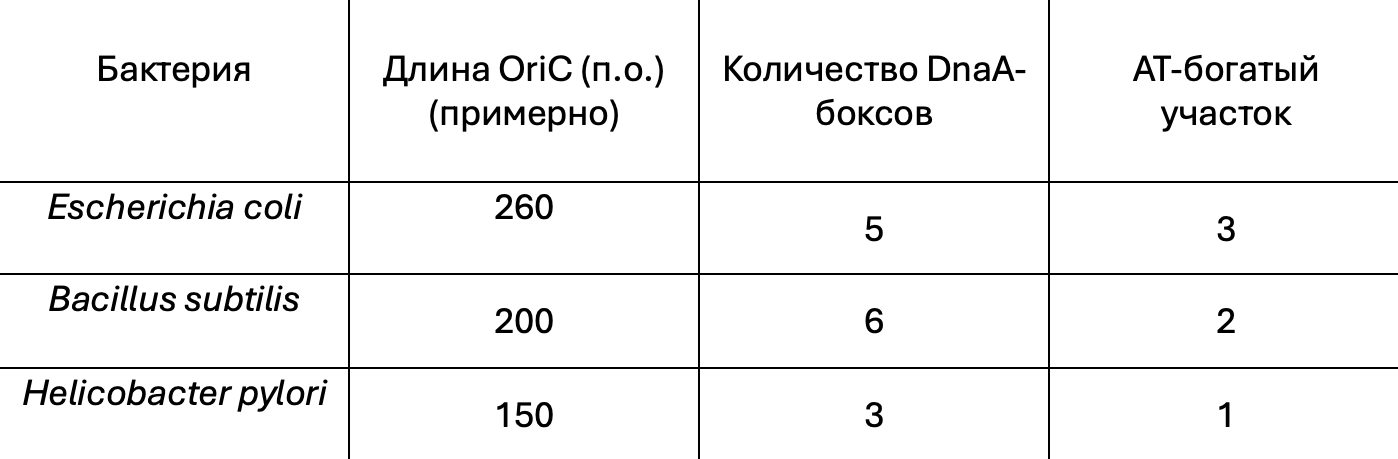
Сравнение OriC разных бактерий
Поиск OriC
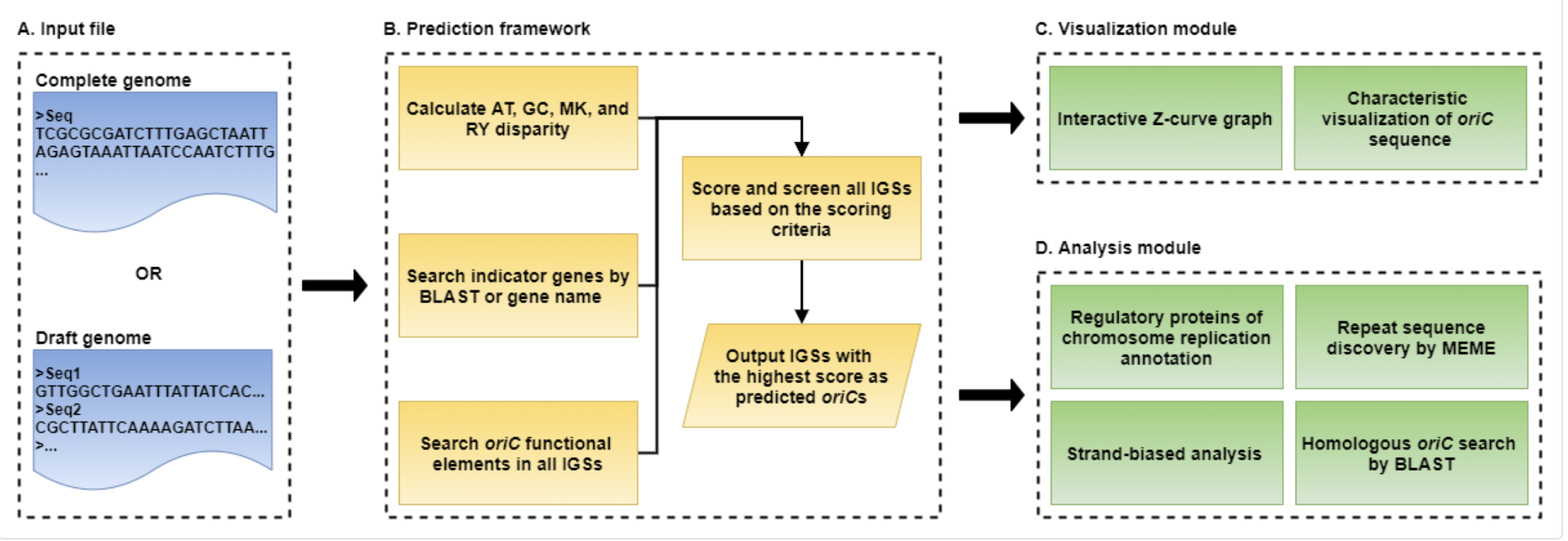
Изображение 1 Схема работы Ori-Finder
выдачу целиком можно увидеть здесь
Сервис предсказал один OriC (хотя в результате работы Ori-Finder у другой сборки генома этой же бактерии нашлось 2, но мы рассматриваем непосмредственно DoriC и непосредственно вышеупомянутую сборку). Длина полученного ориджина составляет 522 нуклеотида. Также можно увидеть его координаты (2720195..2720716 nt), а также координаты генов, белковые продукты котоорых взаимодействуют с ориджином. Например, ген DnaA действительно расположен вблизи OriC (относительно всего генома). Помимо этого, ресурс также предоставляет последовательность аннотированного OriC с отмеченными функциональными участками.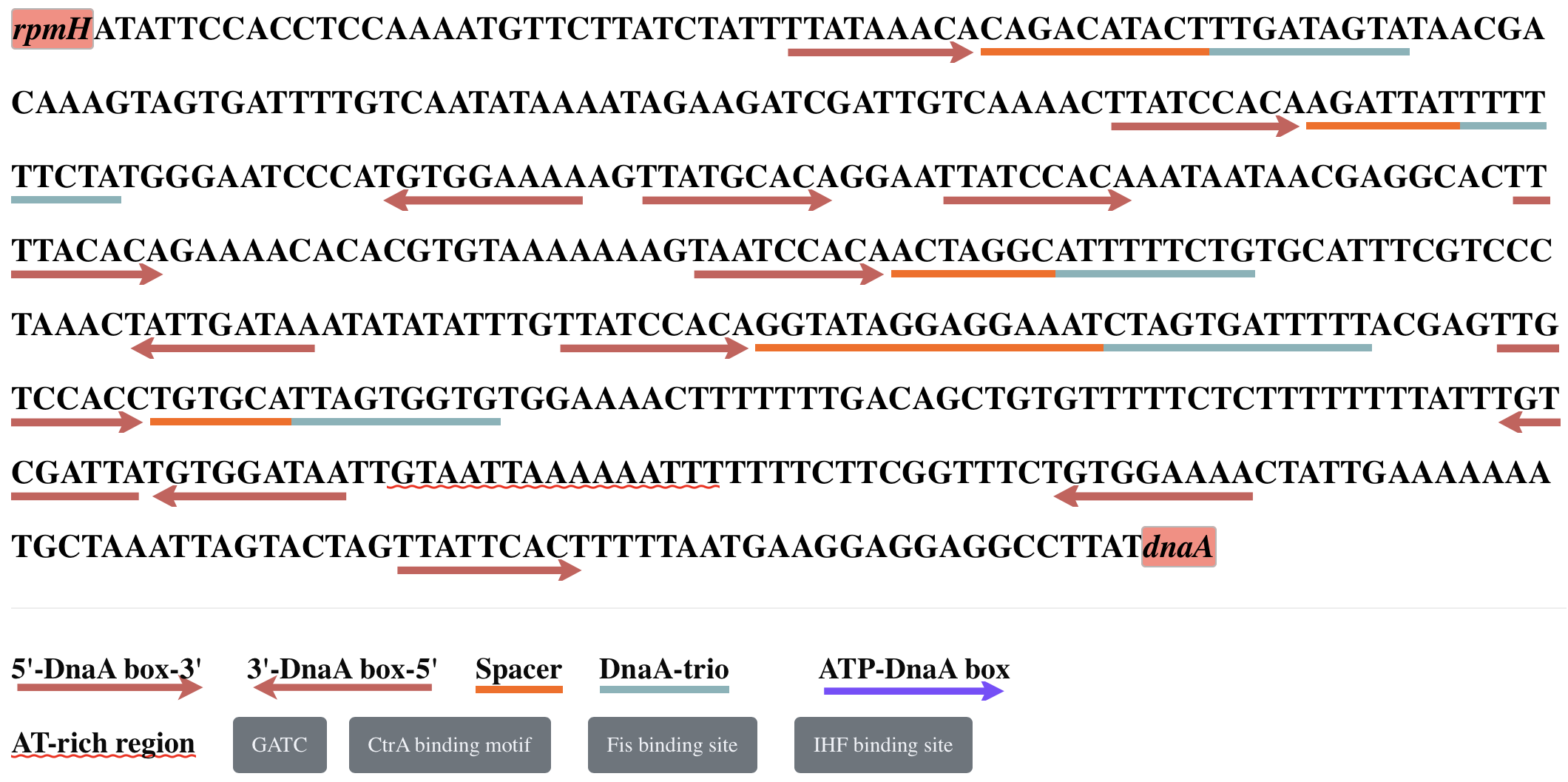
Изображение 2 Последовательность OriC
Авторы
Беккер Диана (bekkerd1203)
Скрипачев Глеб (gskripachev)
Источники
1. Leonard, A. C., & Grimwade, J. E. (2015). The orisome: structure and function. Front Microbiol. 2015 Jun 2;6:545. doi: 10.3389/fmicb.2015.00545
2. Moriya, S., et al. (1992). Structure and function of the region of the replication origin of the Bacillus subtilis chromosome. Nucleic Acids Research, 1985 Apr 11;13(7):2267-79. doi: 10.1093/nar/13.7.2267
3. Donczew, R., et al. (2015). The atypical response regulator HP1021 controls formation of the Helicobacter pylori replication initiation complex. Mol Microbiol, 2015 Jan;95(2):297-312. doi: 10.1111/mmi.12866