Практикум 9
В качестве парных мнемоник я выбрал мнемоники FUR;DER;GLK, по ним выполнялись первые 3 пункта практикума. По мнемоники FUR также было выполнено множественное выравнивание из пункта 5.
Подсчет инделей выполнялся с помощью скрипта написанного на Python - он загружен на kodomo.
2. Глобальное парное выравнивание гомологичных белков
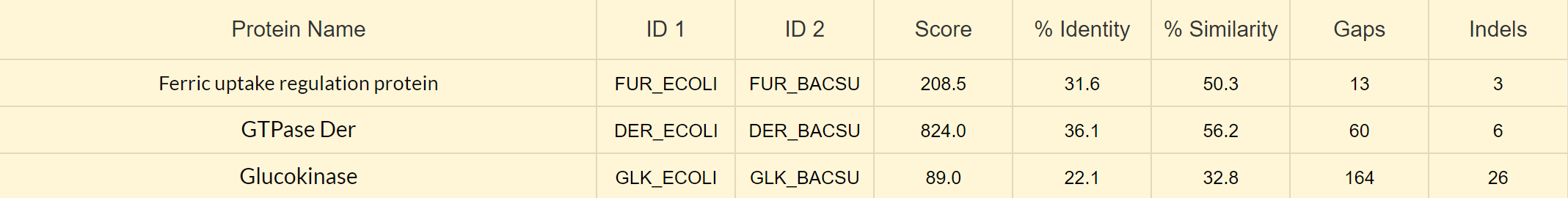
3. Локальное выравнивание
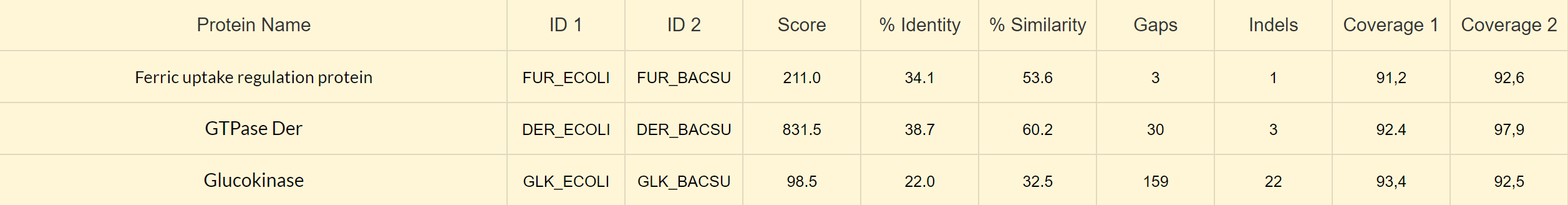
4. Результат применения программ выравнивания к неродственным белкам
В качестве неродственных белков я взял ALF_ECOLI и AMPA_BACSU


Очевидно, что белки негомологичны, у них низкий процент идентичности, довольно много инделей как и в глобальном, так и в локальном выравнивании, также невысокий процент сходства.
5. Множественное выравнивание белков и импорт в Jalview
Я выбрал мнемонику FUR и организмы SHIFL, HAEIN, PSEAE, STAAU, BORPE, по которой нашел дополнительно белков различных организмов к уже имеющимся 2-м. Всего таких белков было найдено 49, поиск я производил на kodomo с помощью команды infoseq 'sw:fur_*' -only -name -nohead -out fur.txt, а подсчет белков при помощи команды wc -l fur.txt. Рекомендуемое полное имя белка - Ferric uptake regulation protein. Данный белок работает как негативный контролирующий элемент, который использует Fe2+ как кофактор при связывании оператора репрессированных генов. Выравнивание проводилось на kodomo командойmuscle с предварительным преобразованием файла в формат fasta командой seqret Файл с выравниванием был импортирован в Jalview, с результатом можно ознакомиться по данной ссылке. Белки гомологичны, это следует из выравнивания, есть высококонсервативные участки, например с 54 по 61.