Практикум №9
1.Данные
В качестве сигнальной последовательности я выбрал последовательность Шайна-Дальгарно, которая играет важную роль в связывании рибосомы с мРНК прокариот. Поиск производился по организму Citrobacter tructae. Файл с нуклеотидной последовательностью.
Далее при помощи скрипта написанного на Python было сделано 3 набора последовательностей: группа положительного контроля (25 нуклеотидов до старт-кодона) Файл, отрицательного контроля (25 нуклеотидов после старт-кодона) Файл и группа, в которой и ожидалось найти сигнал Файл.
2.MEME
При помощи MEME и команды meme TRAIN.fasta -dna -nmotifs 1 -minw 5 -maxw 10, я нашел мотив, похожий на искомый сигнал. Выдача программы.
Исходя из низкого E-value: 5.9e-024, можно сделать вывод о важности данной находки, а именно консенсус: CAGGAG. Данный мотив довольно похож на последовательность SD. Паттерн: VAGGAG
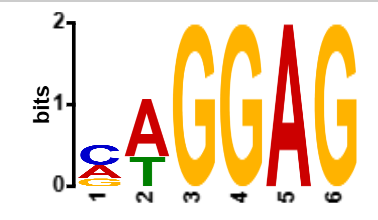

3.FIMO
С помощью FIMO я провел поиск мотива по положительному и отрицательному контролям, использовав команду fimo --oc fimo_pos -thresh 0.001 meme_out/meme.txt POSITIVE.fasta (для отрицательного использовался файл NEGATIVE.fasta).
Результаты выдачи программы: Положительный контроль; Отрицательный контроль.
В группе положительного контроля было обнаружено 869 значимых находок с p-value меньше 0.001. А в группе отрицательного контроля было найдено всего 73 мотива с p-value меньше 0.001. Из этого следует, что мотив CAGGAG может являться консенсусной последовательностью Шайна-Дальгарно, так как в отрицательном контроле у нас мало находок, что правильно, потому что в отрицательной группе последовательности вырезались, по-сути в случайных местах..