Cеми-эмпирические вычисления: Mopac
Задание 1.
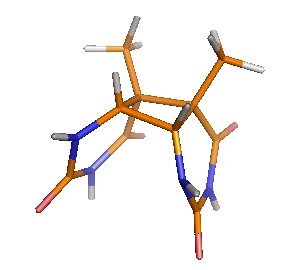
SMILES порфирина: c1cc2cc3ccc(cc4ccc(cc5ccc(cc1n2)[nH]5)n4)[nH]3
Построена 3D структура порфирина, PDB-файл.

С помощью программ babel -ipdb 1.pdb -omop 1_opt.mop -xk "PM6" - параметризация PM6 и
MOPAC2009.exe 1_opt.mop
babel -imopout 1_opt.out -opdb 1_opt.pdb
получили оптимизированную структуру порфирина PDB(MP6).

Структура с другим уровнем параметризации
babel -ipdb 1.pdb -omop 2_opt.mop -xk "AM1" - параметризация AM1 и
MOPAC2009.exe 2_opt.mop
babel -imopout 2_opt.out -opdb 2_opt.pdb
визуально не отличается от полученной ранее PDB(AM1).
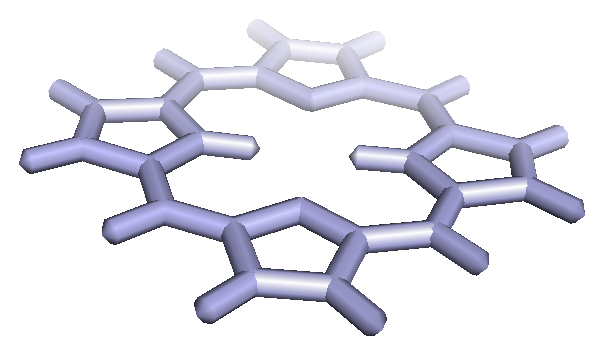
Наложение двух оптимизированных структур:

Задание 2. Рассчет возбужденного состояния порфирина.
С помощью команды
MOPAC2009.exe 1_opt_spectr.mop проводится рассчет возбужденных состояний порфирина. В конце выходного файла -
значения энергии переходов:

Рассчет длины волны:

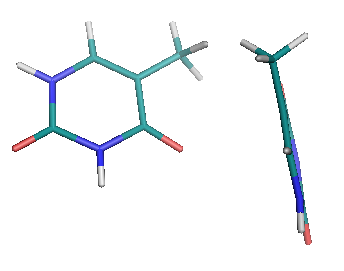
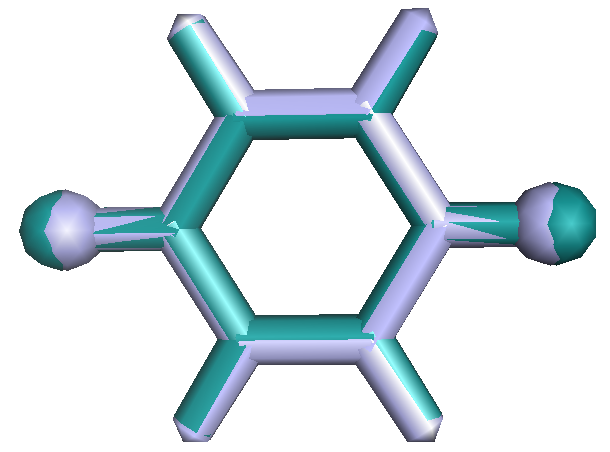
Задание 3. Оптимизация парабензохинона.
SMILE: O=C1C=CC(=O)C=C1
с помощью команд:
obgen 3.smi > 3.mol
babel -ipdb 3.pdb -omop 3_opt.mop -xk "PM6"
MOPAC2009.exe 3_opt.mop
babel -imopout 3_opt.out -opdb 3_opt.pdb
получены две оптимизации структуры данной молекулы. Из кратинки видно, что структуры практически не отличаются друг от друга.
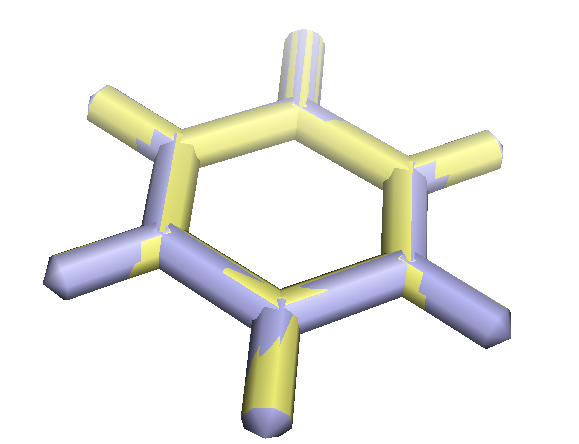
При сравнении парабензохинона с его дианионом (PDB) видно, что связи С-О в дианионе длинее, поскольку двойная связь всегда короче одинарной.
Задание 4. Оптимизация тиминового димера.