Нуклеотидный BLAST
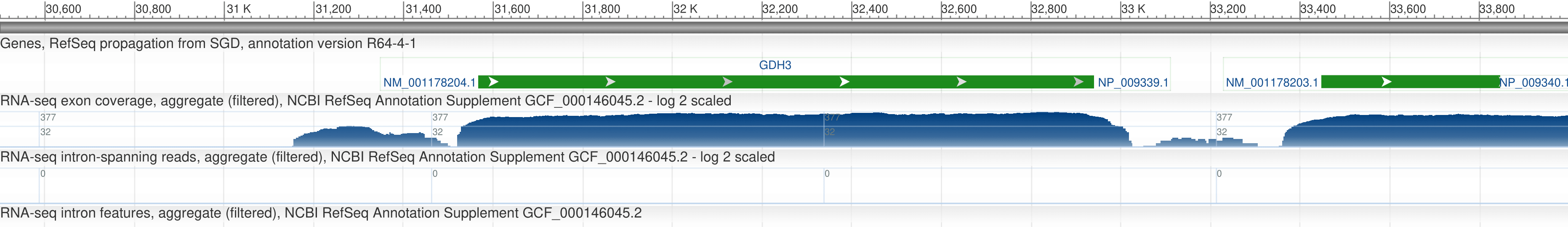
Для выполнения задания явыбрал участок 30500..34000 первой хромосомы Пекарских Дрожжей (идентификатор GCA_000146045.2). На рисунке 1 показана схема данного участка: в середине расположен ген НАДФ+ зависимой дегидрогеназы, а в конце предположительно кусок дегидрогеназы DBH2 (ген предсказан по последовательности). Последовательность можно скачать по следующей ссылке.
Поиск я проводил по таксону Basidiomycota, то есть сравнимые организмы из одного царства Fungi. Запрос при помощи megablast дал только находку Ustilago bromivora, а nblast с длиной слова 11 - 37 находок, blastx с длиной слова 6 и порогом 0.0005 - 4069, tblastn для DGH3 c длиной слова 6 и порогом 0.0005 - 139. В данном случае некорретно сравнивать результаты работы алгоритмов, так как они, как минимум, имеют разные допустимые размеры слова. Но можно заметить, что много находок имеют покрытие больше 30 процентов, что является значимым. С уменьшением длины слова растет количество находок. Выдача tblastn меньше, чем blastx, так как tblastn запускается с белковой последовательностью (не весь участок), и почти все находки обладают покрытием больше 70%.
Алгоритм megablast нужен для того, чтобы картировать риды на референсный геном организма, например, для Hi-C или Red-C (протоколы определения ДНК-ДНК и ДНК-РНК взаимодействий в ядре). Blastn может понадобится для определения вида червя на Белом море. Blastx нужен в том случае, когда у нас есть белок, но мы хотим при выравнивании увидеть все мутации (втч синонимичные), а tblastx пригодится для определения гомологов при сборке генома.
Во втором задании я искал гомологи 16S и 23S рРНК E. coli у выбранного организма. 16S сидит в малой субъединице рибосомы, она учасвтвует в узнавании последовательности Шайн-Дальгарно и стабилизации кодон-антикодонового взаимодействия в А-сайте [1]. 23S находится в большой субЪединице рибосомы и стабилизирует взаимодествия в Р-сайте [2]. Для этого я использовал blastn со стандартными настройками, так как он нужен для поиска гомологов. Для каждой рРНК получилось по одной значимой находке (остальные имеют слишком большое E-value): 16S, 23S. Гомолог 16S, вероятно, связывает последовательность Козак, оба гомолога также участвуют в стабилизации кодон-антикодоного взаимодействия. В геноме дрожжей по этим координатам аннотированы 18S и 25S рРНК соответственно, что было ожидаемо.