| Задание | A- форма |
|---|---|
| Выделить сахарофосфатный остов ДНК | 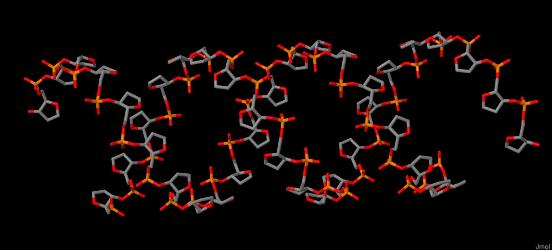 |
| Выделить все нуклеотиды | 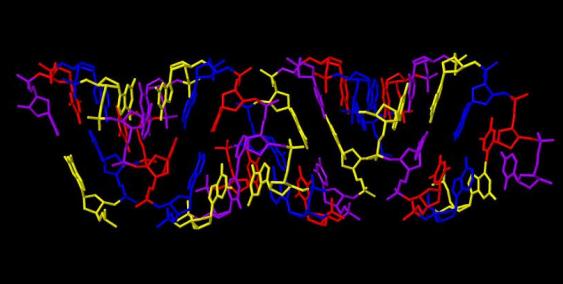 |
| Выделить все аденины |  |
| Выделить атом N7 во всех гуанинах |  |
| Выделить атом N7 только в первом по последовательности гуанине |  |
В директории Term3/Practice2 с помощью программы Putty указываем путь к 3DNA, вводя команды:
| Задание | A- форма |
|---|---|
| Выделить сахарофосфатный остов ДНК |  |
| Выделить все нуклеотиды |  |
| Выделить все аденины |  |
| Выделить атом N7 во всех гуанинах |  |
| Выделить атом N7 только в первом по последовательности гуанине |  |
На сайте PDB выполним поиск по идентификаторам 1FFY и 1R4O. Выберем подходящие варианты структур.
На рисунках 1 и 2 представлены структуры тРНК 1FFY и ДНК-белкового комплекса 1R4O соответственно.
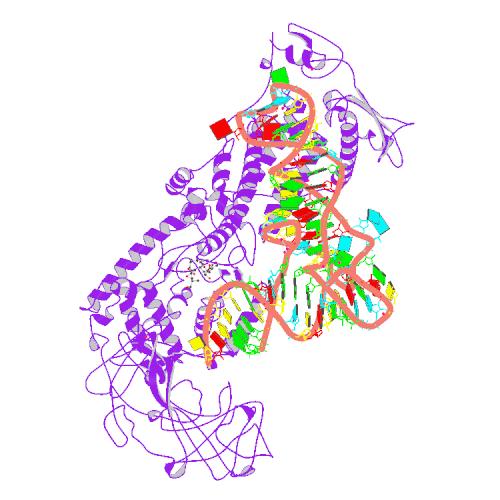
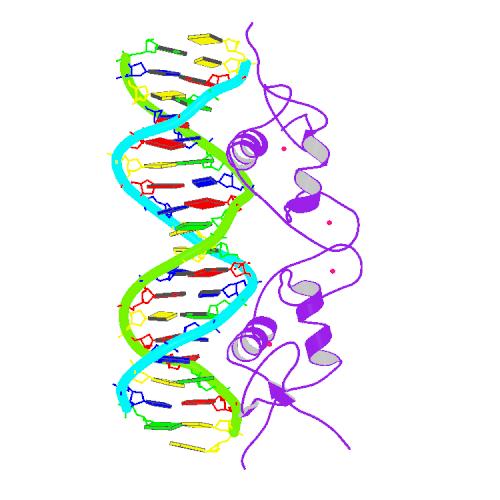
Выбранные варианты структур, представленные выше, сохранены в файлах 1FFY.pdb и 1R4O.pdb.
Данные структуры можно рассмотреть в программе RasMol, изучив их на наличие разрывов.
В рассматриваемых структурах 1FFY и 1R4O разрывы отсутствуют. Ниже представлены изображения только нуклеиновой кислоты в проволочной модели для рассматриваемых структур.
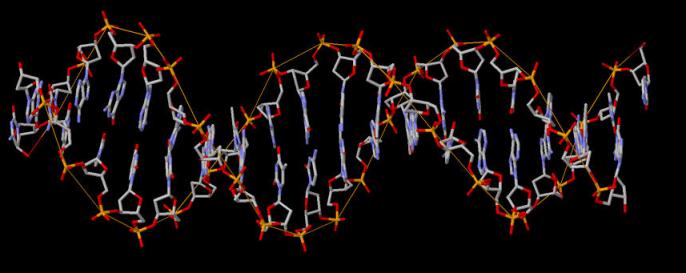
C помощью программы RasMol откроем файл gatc-b.pdb. Рассмотрим структуру B-формы ДНК и визуально определим большую и малую бороздку. Выберем в структуре цитозин и определим, какие атомы основания явно обращены в сторону большой бороздки, а какие в сторону малой:
С помощью программы ChemSketch получим изображение рассматриваемого основания. Выделим красным цветом атомы, смотрящие в сторону большой бороздки, синим - в сторону малой:
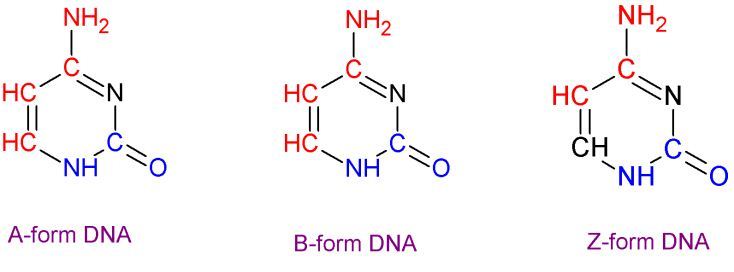
В таблице 1 представлена информация об атомах основания С9.
| Форма ДНК | В сторону большой бороздки обращены атомы: | В сторону малой бороздки обращены атомы: | Остальные атомы основания: |
| A-форма | c9.c6, c9.c5, c9.c4, c9.n4 | c9.n1, c9.c2, c9.o2 | c9.n3 |
| B-форма | c9.c6, c9.c5, c9.c4, c9.n4 | c9.n1, c9.c2, c9.o2 | c9.n3 |
| Z-форма | c9.c5, c9.c4, c9.n4 | c9.n1, c9.c2, c9.o2 | c9.c6, c9.n3 |
Результаты изучения структур из файлов gatc-a.pdb, gatc-b.pdb и gatc-z.pdb с помощью программы RasMol запишем в виде таблицы 2:
| A-форма | B-форма | Z-форма | |
| Тип спирали | Правая | Правая | Левая |
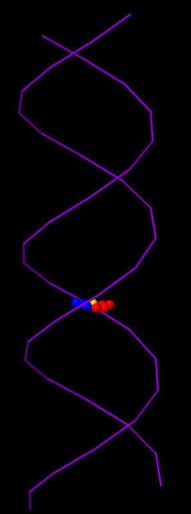
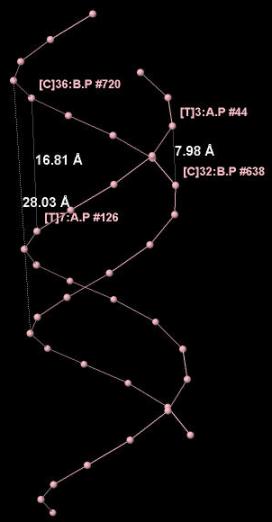
| Шаг спирали (Â) | 28.03 | 33.76 | 43.50 |
| Число оснований на виток | 11 | 10 | 12 |
| Ширина большой бороздки (Â) | 16.81 (от цитозина) |
17.91 (от аденина) |
18.30 (от гуанина) |
| Ширина малой бороздки (Â) | 7.98 (от цитозина) |
11.69 (от аденина) |
8.68 (от цитозина) |
Ниже приведена иллюстрация измерения ширины большой, малой бороздок и шага спирали в A-форме ДНК:
С помощью команды Settings -> Torsion в RasMol измерим торсионные углы цитозина. В таблице 3 приведены, кроме найденных величин, значения торсионных углов, данные для сравнения (см. в презентации).
| α (P - O5') | β (O5' - C5') | γ (C5' - C4') | δ (C4' - C3') | ε (C3' - O3') | ξ (O3' - P) | χ (C1' - N) | |
| A-ДНК | -51.74 | 174.69 | 41.73 | 79.31 | -147.75 | -75.25 | -157.18 |
| B-ДНК | -29.89 | 136.62 | 32.12 | 143.34 | -140.77 | -160.20 | -97.97 |
| Значения торсионных углов из презентации | |||||||
| A-ДНК | -62 | 173 | 52 | 88 или 3 | 178 | -50 | -160 |
| B-ДНК | -63 | 171 | 54 | 123 или 131 | 155 | -90 | -117 |
Для каждого из файлов gatc-a.pdb, gatc-b.pdb и gatc-z.pdb была выполнена следующая команда:
find_pair -t gatc-x.pdb stdout | analyze
В результате для каждой структуры создан ряд файлов с описанием разных её параметров. Например, в файлах gatc-a.out, gatc-b.out и gatc-z.out можно найти значения торсионных углов. Для удобства значения торсионных углов для рассматриваемых структур приведены в файле torsion.txt.
Если сравнить значения соответствующих торсионных углов в структурах A-, B- и Z-форм ДНК, то в наибольшей степени различаются значения следующих углов: у A- и B-форм - δ и χ; у A- и Z-форм - α; у B- и Z-форм - α, ξ и χ. (Cравнения с торсионными углами Z-формы ДНК достаточно условны.)
Так как пакет 3DNA работает только со старым форматом .pdb, используем программу remediator для перевода файлов 1FFY.pdb и 1R4O.pdb в старый формат:
remediator --old "XXXX.pdb" > "XXXX_old.pdb"
Затем применим команды find_pair и analyze (использованные выше).
Значения торсионных углов в структуре тРНК (1FFY_old.pdb) для удобства приведены в файле torsion.txt. Данная структура больше всего похожа на A-форму ДНК, если рассматривать значения торсионных углов.
В заданной ДНК 1R4O нуклеотидами с наиболее отклоняющимися значениями углов являются нуклеотиды с азотистыми основаниями тимин-4 и цитозин-10 из цепи D (см. док. Excel ).Strand I Strand II Helix 13 (0.005) T:..54_:[..U]U-**-xA[..A]:..58_:T (0.007) | 14 (0.008) T:..55_:[..U]Ux**+xG[..G]:..18_:T (0.011) x 15 (0.011) T:..36_:[..U]Ux**+-U[..U]:..33_:T (0.005) | 27 (0.003) T:..14_:[..A]A-**-xU[..U]:...8_:T (0.007) | 28 (0.006) T:..15_:[..G]G-**+xC[..C]:..48_:T (0.008) x 29 (0.005) T:..16_:[..G]Gx**-xU[..U]:..17_:T (0.003) + 30 (0.008) T:..19_:[..G]G-----C[..C]:..56_:T (0.004) +
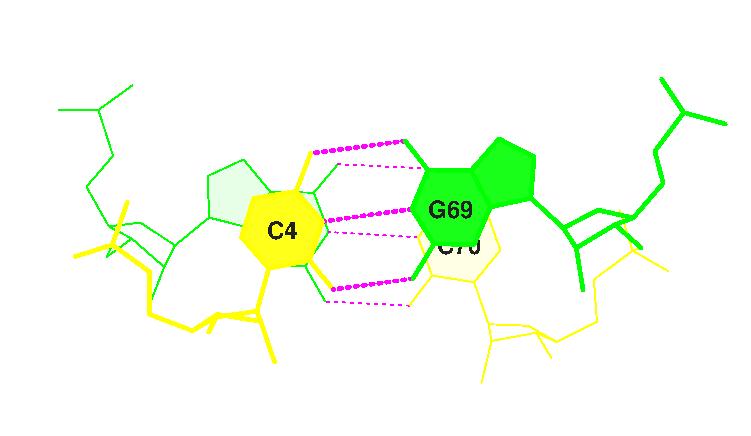
Из файла 1FFY_old.out узнаём, что наибольшая площадь перекрывания пар характерна для третьей секции (Section #0003)
С помощью команд :
ex_str -3 stacking.pdb step3.pdb stack2img -cdolt step3.pdb step3.psи программы Ghost_View получаем изображение самого сильного стекинг-взаимодействия: