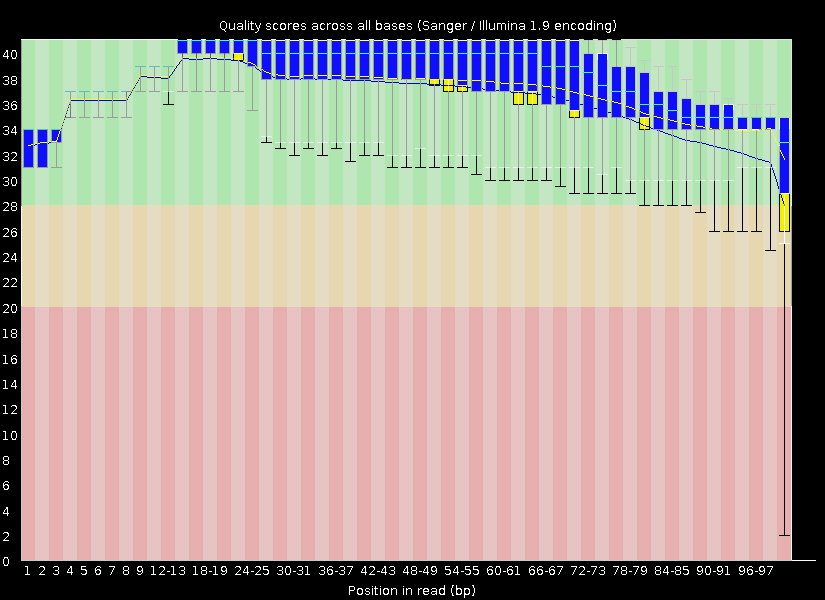
Исходно было получено 8208 чтений. С помощью программы Trimmomatic с конца каждого чтения были отрезаны нуклеотиды с качеством ниже 20 и оставлены только чтения длиной не меньше 50 нуклеотидов. Результат работы Trimmomatic:
Input Reads: 8208 Surviving: 8114 (98,85%) Dropped: 94 (1,15%) Разница в качестве чтений до и после очистки отображена на Рис. 1. Видно, что различия есть не везде и в целом довольно незначительные.
Картирование чтений
8084 (99.63%) чтений были картированы на 5 хромосому. Качество картирования можно считать высоким.
Аннотация полиморфизмов
Всего обнаружено 32 полиморфизма, из них 28 SNP, 4 - делеции и вставки. Интересно, что все полиморфизмы расположены на двух участках: примерно 35857000-35937000 и 74640000-74657000.
| 1 | 2 | 3 | |
|---|---|---|---|
| Координата | 35 857 308 | 35 874 575 | 74 639 544 |
| Тип полиморфизма | Вставка | Замена | Делеция |
| Референс | T | C | CTTGTATTGT |
| Чтения | TC | T | CTTGT |
| Глубина покрытия | 40 | 164 | 23 |
| Качество чтений | 177 | 225 | 73.5 |
База данных refseq в annovar делит snp по их расположению в последовательности.
4 оказались экзонными, 24 - интронными, один - в 3'-нетранслируемой области.
Из экзонных snp 3 попали в ген IL7R - рецептор интерлейкина-7 (замены T197C, G412A, C731T; в аминокислотах соответственно: I66T, V138I, T244I), а один - в ген CAPSL - кальцифозин-подобный белок (замена - G254A ; в аминокислотах соответственно: R85Q).
24 snp имеют rs номер по данным базы dbsnp. В базе данных 1000genomes нашлось 23 snp, средняя частота составила 0.42
Клиничиская аннотация
Согласно базе данных GWAS,
три SNP ассоциированы с нарушениями обмена холестерина, а другие два - с диабетом I типа (в гене IL7R).
База данных ClinVar содержит информацию о трех SNP, все в гене IL7R. Два полиморфизма предположительно
патогенны и могут быть связаны с тяжёлым комбинированным иммунодефицитом, один - неизвестно.
Использованные команды:
| hisat2-build chr5.fasta chr5 | Индексирование референса |
| fastqc chr5.fastq | Проверка качества чтений |
| java -jar /nfs/srv/databases/ngs/suvorova/trimmomatic/trimmomatic-0.30.jar SE -phred33 chr5.fastq chr5_trimmed.fastq TRAILING:20 MINLEN:50 | Очистка чтений |
| fastqc chr5_trimmed.fastq | Проверка качества после очистки чтений |
| hisat2 -x chr5 -U chr5_trimmed.fastq -S chr5_align.sam --no-softclip --no-spliced-alignment | Выравнивание прочтений и референса |
| samtools view -b chr5_align.sam -o chr5_align.bam | Перевод выравнивания в бинарный формат |
| samtools sort chr5_align.bam chr5_align_sorted | Сортировка выравнивания по координате в референсе |
| samtools index chr5_align_sorted.bam | Индексирование отсортированного файла |
| samtools flagstat chr5_align.sam | Получение основной информации о картировании, в т.ч. числа откартированных чтений |
| samtools mpileup -uf chr5.fasta chr5_align_sorted.bam -o chr5_snp.bcf | Создание файла с полиморфизмами |
| bcftools call -cv chr5_snp.bcf -o chr5_snp.vcf | Перевод полученного файла в текстовый формат |
| vcftools --vcf chr5_snp.vcf --remove-indels --recode --out chr5_snp_noind | Удаление инделей из vcf-файла |
| convert2annovar.pl -format vcf4 chr5_snp_noind.recode.vcf -outfile chr5_snp.avinput | Получение входного файла для annovar |
| annotate_variation.pl -out chr5_refgene -build hg19 chr5_snp.avinput /nfs/srv/databases/annovar/humandb.old/ | Аннотация по базе данных refGene |
| annotate_variation.pl -filter -out chr5_dbsnp -build hg19 -dbtype snp138 chr5_snp.avinput /nfs/srv/databases/annovar/humandb.old/ | Аннотация по базе данных dbsnp |
| annotate_variation.pl -filter -dbtype 1000g2014oct_all -buildver hg19 -out chr5_1000genomes chr5_snp.avinput /nfs/srv/databases/annovar/humandb.old/ | Аннотация по базе данных 1000 genomes |
| annotate_variation.pl -regionanno -build hg19 -out chr5_gwas -dbtype gwasCatalog chr5_snp.avinput /nfs/srv/databases/annovar/humandb.old/ | Аннотация по базе данных Gwas |
| annotate_variation.pl chr5_snp.avinput /nfs/srv/databases/annovar/humandb.old/ -filter -dbtype clinvar_20150629 -buildver hg19 -out chr5_clinvar | Аннотация по базе данных Clinvar |