на главную страницу
1. Рассчет веса строк выравнивания программой pfw
Полученный файл отличается от исходного выравнивания одним - изменились цифры характеризующие
вес для каждого белка. Первоначально все они были равны единице.
Веса
2. Создание профиля программой pfmake
pfmake rs6_al.msf /usr/share/pftools23/blosum62.cmp > made_pro.txt
Профиль созданный программой.
3. Поиск по созданному профилю в банке bacteria
Удачный порог =3, с ним найдено 832 белка.
pfsearch -C3.0 -f made_pro.txt bacteria.fasta > my.pfsearch
4. Анализ результатов
Последовательностей найдено - 831.
True positive hits, TP - 47
False positive hits, FP - 784
False negatives FN - 0
Чувствительность TP/(TP+FN) - 1
Селективность TP/(TP+FP) - 5,65%
Аналогично предыдущему практикуму анализ проводился по порогу, при котором селективности
стремились к селективности моего паттерна.
Порог - 89,3617%. Однако селективности выданные программой находятся на уровне до 9 процентов.
True positive hits, TP -
False positive hits, FP -
False negatives FN - 0
Чувствительность TP/(TP+FN) - 1
Селективность TP/(TP+FP) - %
ROC-кривая для результатов поиска белков программой
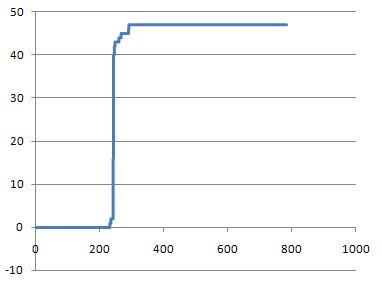
Причина
Выдача белков, произведенная программой получилась весьма интересной: правильные белки, составляющие True positives находятся в соответствии со своими весами в середине массива. Поэтому график roc-кривой
имеет такой вид. И это затрудняет получить селективность т.к. в ряду белков селективности сначала возрастают, а потом убывают достигнув максимума на уровне 9%
Книга Excel показывает это. Третий лист в ней, "cheat" содержит массив белков, с ограничением сверху, так что интересующие нас белки имеют наивысшие веса, но и это не помогло посчитать селективность.
©Джумашев