Восстановление предкового состояния доменной архитектуры
Часть 1. Выбрать объекты изучения и построить выравнивание
NAT (N-ацетилтрасферазный домен) (AC в Pfam PF04768) участвует в катализе начальных этапов синтеза аргинина. Он найден в 931 белковой последовательности, содержится в 18 доменных архитектурах. Будем смотреть на две архитектуры (см. рисунок 1):
- Содержит домен-киназу аминокислот (AA_kinase), NAT-домен и семиальдегид дегидрогеназу, НАД-связывающий домен (Semialdhyde_dh). Встречается в 180 белках
- Содержит AA_kinase и NAT-домен. 128 белков
В качестве таксона верхнего порядка я выбрала Cellulata, подтаксонами стали Fungi и Metazoa.
Excel файлы: файл со сводкой по всем белкам, имеющим домен NAT, файл с выбранными мной последовательностями для дальнейшней работы. Получены при помощи скриптов swisspfam-to-xls.py, uniprot-to-taxonomy.py, Uniprot ID retriever.
Затем получили выравнивание всех последовательностей из данного семейства (ссылка на проект) и выбранных последовательностей (ссылка на проект). Раскраска BLOSUM62, Above identity threshold 70%, убраны пустые колонки. В выравнивании есть сильно консервативные позиции, они выравнены и окрашены, поэтому можно судить о правильности выравнивания. Три последовательности были совсем неправильно выравнены или имели совсем непохожие последовательности. Они были убраны.
Часть 2. Построение филогенетического дерева домена
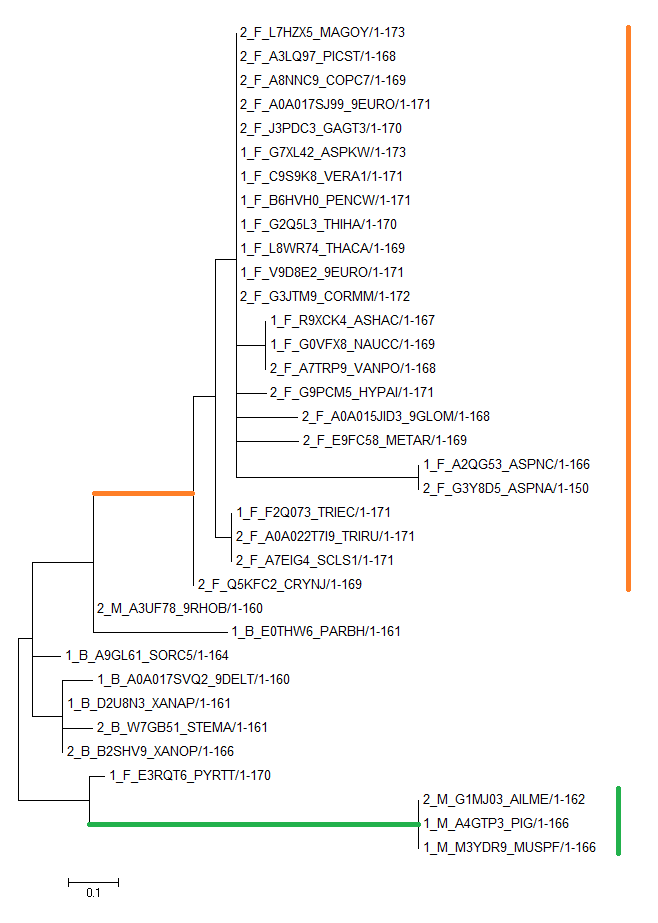
В листьях дерева на рисунке 2 приняты такие обозначения: цифры -- номера архитектур в соответствии с рисунком 1, F - Fungi, M - Metazoa. B - Bacteria (этот таксон был взят на всякий случай, для проверки).
Скобочная структура дерева доступна по ссылке. Дерево построено в MEGA алгоритмом Maximum-Likelihood.
Часть 3. Построить профиль подсемейства и охарактеризовать качество его работы
Для работы было выбрано подсемейство, обозначенное рыжим цветом на рисунке 2. Для построения профиля выравнивания использовали пакет HMMER. Откалиброванный профиль м можно посмотреть по ссылке.
Затем профиль применили к fasta-файлу, содержащему все белки с доменом NAT. Информация, которую программа дала на выход, содержится в файле E-value находок принимают значение от 1.4e-09 до 2.3.
Для того, чтобы определить порог E-value, с которого следует отбирать последовательности, была построена ROC-кривая (рис. 3).
Excel-файл с рассчетом данных для ROC-кривой
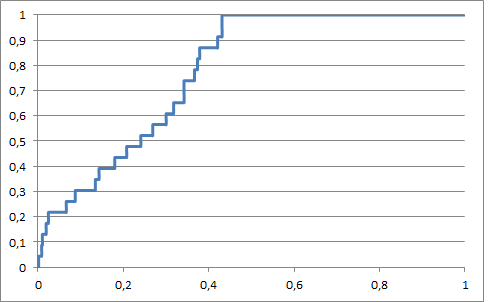
Выбран порог E-value 5.3e-09
Таблица 1. Результаты при пороге E-value 5.3e-09
| На самом деле | принадлежит подсемейству | не принадлежит | сумма |
|---|---|---|---|
| Проходят порог | 19 | 217 | 236 |
| Не проходят порог | 8 | 352 | 360 |
| Cумма | 27 | 569 |
Вывод
По дереву последовательности отлично разделились по по таксонам Fungi, Metazoa и Bacteria (с некоторыми незначительными погрешностями). Однако по архитектурам разделения нет. Это может говорить о том, что домен NAT эволюционировал именно по таксонам. ROC-кривая показала, что построенный профиль подошел для нахождения белков, принадлежащих подсемейству. Но все же есть множество белков, проходящих порог, но не принадлежащих подсемейству.