Учебный сайт Валяевой Анны
Анализ качества и очистка чтений
Работа проводилась с файлом с чтениями генома резуховидки: Ath_tae_CTTGTA_L003_R1_003.fastq.
Анализ качества чтений
С помощью программы FastQC был проведен контроль качества чтений. HTML-версию отчета можно посмотреть здесь. Как видно из графика на рисунке 1 качество ридов изначально достаточно высокое.
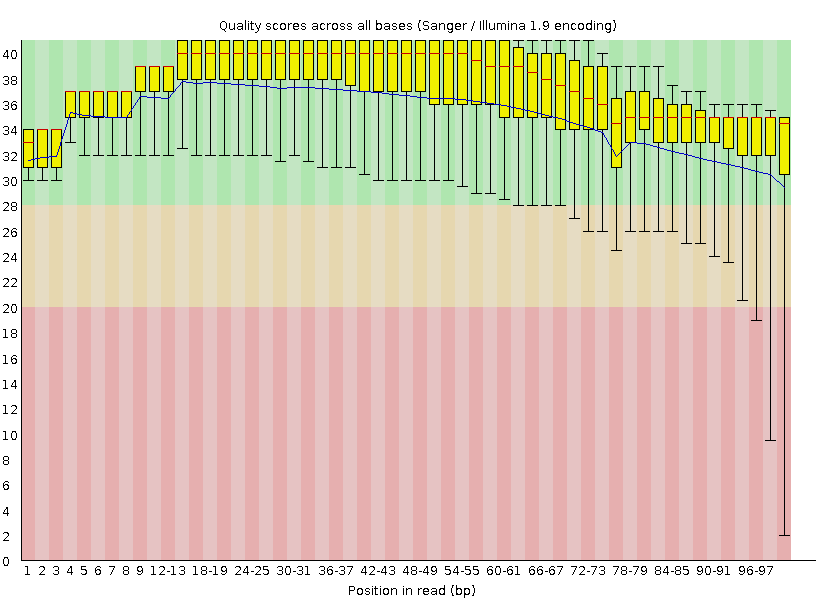
Рис. 1. Графическое представление качества ридов до очистки.
Очистка чтений
Для тримминга (очистки чтений) использовалась программа Trimmomatic. Было нужно удалить последовательности адаптеров, отрезать с конца каждого прочтения нуклеотиды с качеством ниже 20 и оставить только прочтения длиной не меньше 50 нуклеотидов. Это было выполнено с помощью команды:
java -jar /usr/share/java/trimmomatic.jar SE Ath_tae_CTTGTA_L003_R1_003.fastq trimmomatic.fastq
ILLUMINACLIP:adapters.fasta:2:7:7 TRAILING:20 MINLEN:50
Программа автоматически определила форат fastq: phred33. Из 4000000 ридов остались 3849626 (96,24%) и были убраны 150374 (3,76%).
Затем был проведен анализ качества очищенных чтений программой FastQC. HTML-версию отчета можно посмотреть здесь. Как видно из графика на рисунке 2, качество ридов улучшилось.
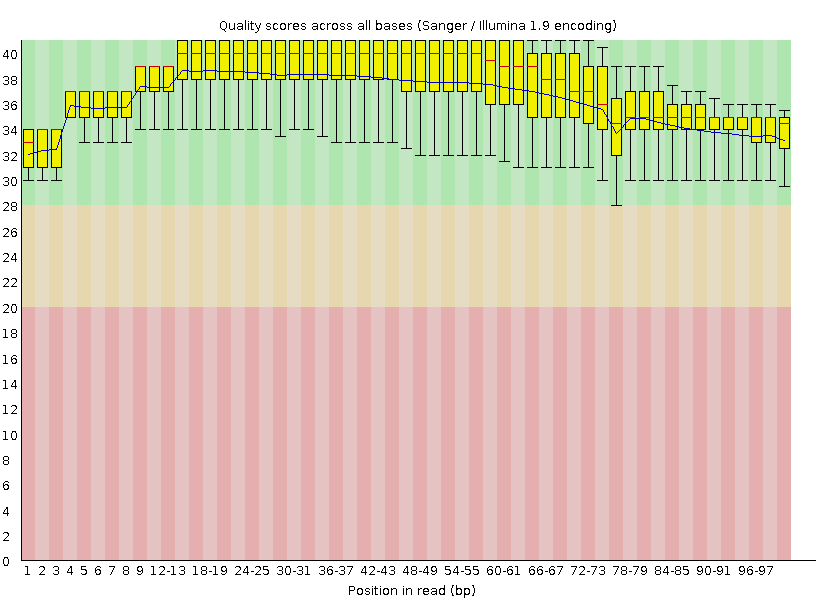
Рис. 2. Графическое представление качества ридов после очистки.
Также из отчета видно, что разброс в длине послледовательностей ридов увеличился, поскольку отрезались адаптеры, но пик сохранился на 100-101 основании. График Per base sequence content практически не изменился.
Дата последнего обновления: 22.12.14
©Валяева Анна