Сигналы и мотивы - 2
Задание 1.
В качестве задачи мной был выбран поиск последовательности Шайно-Дальгарно в геноме бактерии Caldicellulosiruptor owensensis OL, протеом которой я исследовала в рамках работы в первом семестре. Объект исследования представляет собой свободноживущую анаэробную гипертермофильную неспорообразующую бактерию, обитающую в донных отложениях пресноводных водоемов и горячих источников США. Последовательность Шайно-Дальгарно находится перед ATG кодонами генов большинства белков на расстоянии 6-8 нуклеотидов. Она представляет собой сайт связывания рибосом на молекуле мРНК, её консенсус - AGGAGG. На 3'-конце 16S рибосомной РНК находится комплементарная ей последовательность анти-Шайно-Дальгарно, взаимодействием которой с последовательностью Шайно-Дальгарно обеспечивается помещение старт-кодона мРНК в Р-сайт рибосомы для инициации биосинтеза белка. Для белков, upstream-последовательность которых не содержит данной последовательности, вероятно, характерны другие механизмы инициации трансляции.
Для поиска последовательности Шайно-Дальгарно были выбраны гены с прямой цепи ДНК Caldicellulosiruptor owensensis OL, имеющие длину более 300 нуклеотидов, для которых точно определен кодируемый белок (в поле product значение отлично от hypothetical protein). Послная таблица генов из GenBank доступна по ссылке, отобранные из неё CDS - по другой ссылке. С помощью скрипта, использующего файл coords.txt с координатами генов и названиями продуктов трансляции был создан первоначальный входной файл, в котором содержатся 100 последовательностей генов с upstream-участками длиной 40 нуклеотидов и 400 upstream-последовательностей других генов. Также в файл добавлена последовательность гена 16S рибосомальной РНК (рибосомальная РНК должна иметь участок, комплементарный последовательности Шайно-Дальгарно, поэтому взята комплементарная последовательность). Файл был подан на вход программе MEME, я использовала веб-версию. Параметры программы настроены следующим образом: поиск 3 мотивов, встречающихся 0 или 1 раз в каждой последовательности (учитываем, что последовательность Шайно-Дальгарно может отсутствовать перед геном, но мы не ожидаем увидеть её в одной последовательности дважды), длина мотива от 6 до 10 нуклеотидов, поиск только по данной цепи. В результате получаем следующее:
Найден мотив из 10 нуклеотидов, содержащий паттерн A[A,G]GAGG, который, как можно предположить, и является последовательностью Шайно-Дальгарно
Примечательно, что найденная последовательность присутствует в 16S рибосомальной РНК, что видно из картинок выше. E-value этого паттерна составляет 8.4е-044, чтоговорит о том, что он не является очень специфичным. Но, к примеру, р-value для этого мотива в 16S РНК составляет 5.13е-4, что позволяет говорить о значимости находки с точки зрения статистики. Похожий мотив найден в 497 последовательностях, но, как видно из рисунка ниже, не везде он находится в начале последовательности, значит, в каких-то случаях он является частью гена, а не регуляторным участком.
Так как найденный мотив не всегда соответствовал upstream-участку, было решено убрать из выборки "лишние" участки последовательностей и повторить поиск только по участкам, предшествующим ATG. Был соответствующим образом изменен подаваемый в скрипт файл с координатами и получена следующая выборка из upstream-участков генов и 16S РНК. Параметры запуска MEME те же, результат получается следующий:
Это уже идеально соответствует последовательности Шайно-Дальгарно. Для этого участка ниже, хотя и не намного, E-value (3.6е-065), но он находится менее чем в половине последовательностей (242). Большой информационный вес части позиций говорит о том, что они рассматриваются как очень специфичные, поэтому последовательности с мутациями в области последовательности Шайно-Дальгарно.
На скриншотах выше можно видеть порядок p-value для находок в данных последовательностях. Можно отметить, что практически во всех последовательностях данный мотив располагается на одинаковом расстоянии от стартового кодона, что тоже говорит в пользу его специфичности. Если для первого мотива логарифмическое отношение правдоподобия (логарифм отношения вероятности появления мотива в выборке к вероятности его появления в случайной последовательности) составляло 2378, для нового оно равно 1426, при этом информационное содержание для первого мотива составляет 8.3, а для второго - 10.1. Большая информативность соответствует большей разнице между частотами нуклотидов в мотиве и в среднем в последовательности, исходя из этого считаем предпочтительной вторую модель.
Позиционная весовая матрица PWM была построена по аналогии с предыдущим практикумом и доступна по ссылке. Для её построения было взято 200 upstream-участков.
Задание 2.
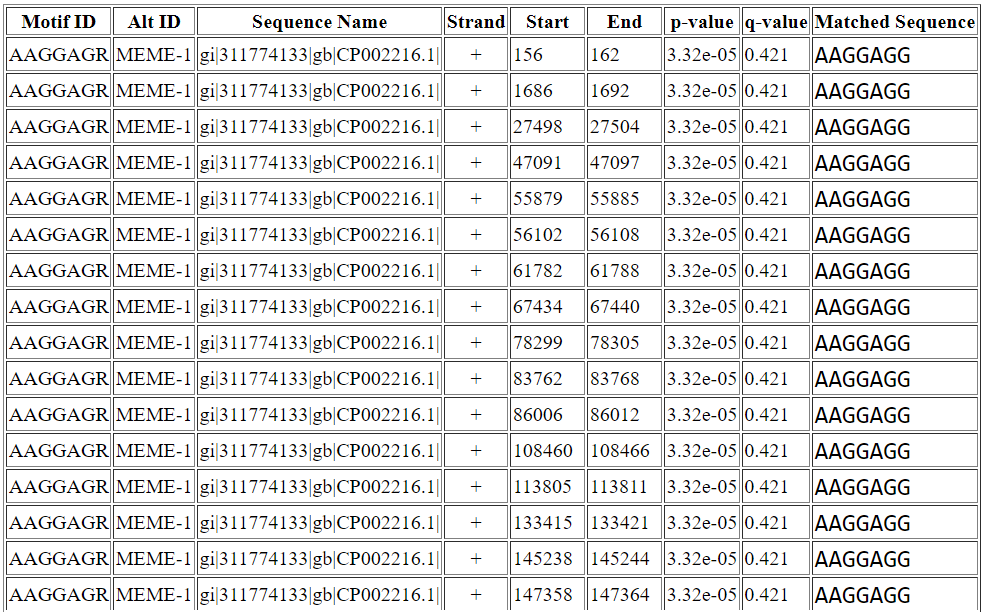
Для проверки матрицы PWM c помощью программы FIMO был создан файл формата MEME motif format с матрицей и входными параметрами: алфавит 4-х-буквенный ACGT, длина мотива до 10 нуклеотидов. Низкого порога на p-value я не устанавливала, чтобы посмотреть любые находки независимо от качества (p-value < 1). Поиск по базе данных для Caldicellulosiruptor owensensis OL не привел к ожидаемому результату (найденный паттерн отличается от AGGAGG), поэтому был создан файл с upstream-последовательностями 974 генов прямой цепи, но мотив опять же не найден. Тогда в качестве мотива я использовала напрямую выдачу программы MEME (Motif: AAGGAGR), осуществляя поиск по базе данных для всего генома Caldicellulosiruptor owensensis OL (Database: db/genbank_Caldicellulosiruptor_owensensis_OL_uid40833_210.fna). На этот раз мотив Best Possible Match, выдаваемый FIMO, совпал с искомым: AAGGAGG. Было найдено 511 мотивов с p-value меньше 0.0001, находки есть как на прямой, так и на обратной цепи, которая не использовалась при формировании выборки.
В результате работы программы получаем таблицу, фрагмент которой представлен на скриншоте. Таблица содержит найденные мотивы с указанием их координат и цепи, на которой они расположены. На следующем шаге исследования нужно проверить, какая часть из них действительно лежит в upstream-участках генов. C этой целью из файла fimo.tsv были взяты координаты, а из ранее составленной таблицы для всех генов Caldicellulosiruptor owensensis OL взяты координаты upstream-участков длиной 40: 40 нуклеотидов до первой координаты для прямой цепи и 40 нуклеотидов после второй координаты для обратной цепи.
Далее был написан очередной скрипт, который принимает на вход списки координат и считает, сколько раз среди мотивов, найденных FIMO, встречаются такие, координаты которых лежат в пределах 40 нуклеотидов перед стартовым кодоном. Для моих данных получилось значение 134. Значит, найденному MEME мотиву соответствуют последовательности в upstreаm-участках 134 генов. На самом деле ожидалось увидеть лучший результат, потому что программа MEME находит данный паттерн в большем количестве последовательностей, но тем не менее некоторый результат, который может свидетельствовать о специфичности найденного мотива, присутствует.