Главная страница > Третий семестр > A- и B-формы ДНК
С помощью программы fiber пакета 3DNA были построены трехмерные структуры A-формы и B-формы ДНК, последовательность одной из цепей которой представляет собой фрагмент gatc, повторенный четыре раза. Команда Linux, с помощью которой была запущена программа fiber для построения структуры A-формы ДНК, приведена ниже (после запуска программа запросила последовательность ДНК и количество повторений этой последовательности):
fiber -a gatcA.pdb
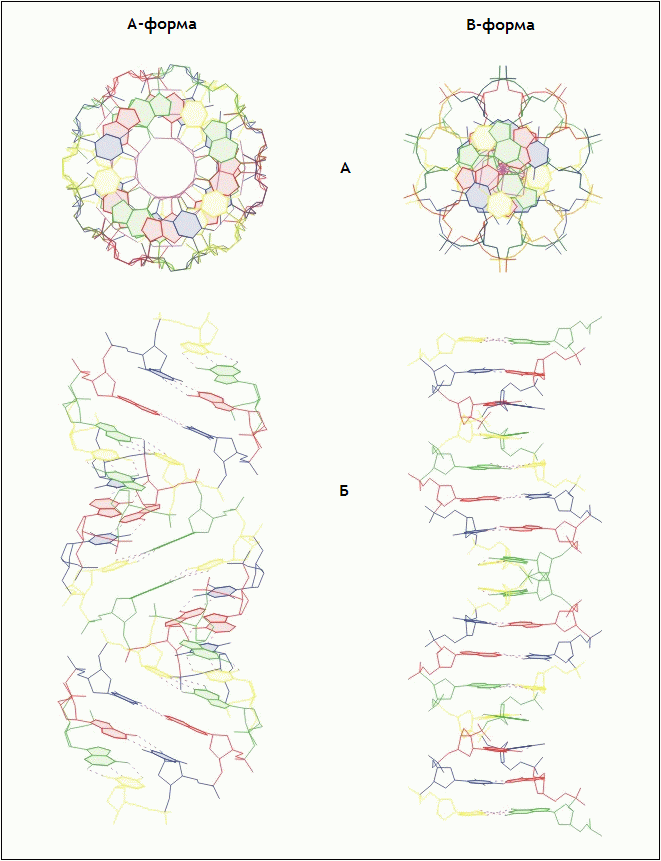
Изображения построенных структур, полученные с помощью stack2img (программа была запущена с параметрами -cdo), приведены на рис. 1. На рис. 2 приведены стопочные модели двух форм ДНК. Для построения стопочных моделей использовалась программа pdb2img. Команда Linux, с помощью которой была запущена pdb2img для построения модели A-формы ДНК (вид с торца), приведена ниже:
pdb2img -cu gatcA.pdb gatcA.ps
При запуске с параметрами -cu программа строит цветное изображение, на котором отдельные азотистые основания представлены закрашенными прямоугольниками*. Для построения повернутых стопочных моделей структур (вид сбоку) предварительно был осуществлен поворот структур в pdb-файлах с помощью программы rotate_mol:
rotate_mol -b gatcA.pdb gatcA_rot.pdb
При запуске с параметром -b программа поворачивает структуру таким образом, что главная ось инерции атомов азотистых оснований становится ориентированной вертикально и параллельно плоскости экрана.
A-форма ДНК отличается от B-формы по следующим признакам [1].
Глубина большого желоба** A-формы ДНК превышает радиус молекулы. Это приводит к тому, что в центре проекции структуры расположено отверстие (рис. 1А и 2А). Глубина желоба B-формы ДНК не превышает радиус молекулы.
В A-форме ДНК атом углерода 3' расположен вне плоскости кольца остатка дезоксирибозы, в B-форме ДНК - атом углерода 2' (остатки дезоксирибозы находятся в конформациях 3'-эндо и 2'-эндо соответственно).
Атомы кислорода фосфатов в структуре A-формы ДНК направлены внутрь большого желоба, в структуре B-формы ДНК - наружу***.
Плоскости азотистых оснований A-формы ДНК расположены под углом 71° к оси молекулы. Плоскости азотистых оснований B-формы ДНК перпендикулярны оси молекулы (рис. 1Б и 2Б).
* Для получения информации о значении параметров потоки stdout и stderr программ pdb2img и rotate_mol при запуске с параметром -h (содержат информацию о значении параметров запуска программ) были перенаправлены в текстовые файлы.
** Большой желоб был идентифицирован как желоб, в который направлены аминогруппы остатков аденина.
*** Показано, что при уменьшении относительной влажности до 75% B-форма ДНК переходит в A-форму [1]. Возможно, это связано с тем, что направленные внутрь большого желоба атомы кислорода фосфатов в A-форме по стерическим причинам взаимодействуют с меньшим количеством молекул воды, чем атомы кислорода фосфатов в B-форме, которые направлены наружу.

Рис. 1. Структуры A- и B-форм ДНК, построенные программой fiber (А - вид с торца, Б - вид сбоку). Различные нуклеотиды показаны цветами. Изображения получены с помощью программы stack2img.
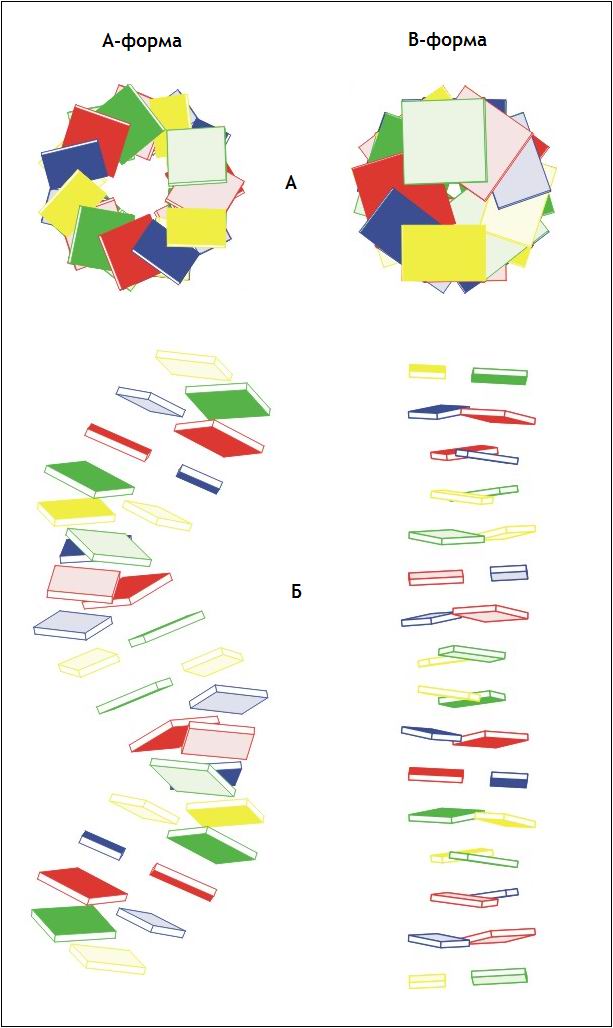
Рис. 2. Стопочные модели A- и B-форм ДНК, построенные программой fiber (А - вид с торца, Б - вид сбоку). Различные нуклеотиды показаны цветами. Изображения получены с помощью программы pdb2img.
Геометрия построенных структур была исследована с помощью RasMol (табл. 1). Обе формы ДНК представляют собой правые спирали (в отличие от Z-формы, которая является левой спиралью). На один виток A-формы ДНК приходится 11 нуклеотидов, что превышает количество нуклеотидов, приходящихся на один виток B-формы (10).
Значения шага спирали и ширины бороздок в обоих структурах практически не зависят от номера нуклеотида, от которого производились измерения. Это обусловлено следующими причинами:
структуры включают в себя только канонические пары азотистых оснований (AT и GC), близкие по размерам (например, пары YY и RR отличаются от канонических пар по размерам и, вероятно, могут вызывать нарушения правильной структуры дуплекса);
алгоритм fiber не учитывает влияние нуклеотидного состава на структуру ДНК (структуры, построенные только из пар GC и только из пар AT, не отличались от исследуемых структур по своим параметрам).
в отличие от реальных структур, построенных по результатам рентгеноструктурного анализа (РСА) или по методу ЯМР, структуры, построенные программой fiber, не учитывают внешних воздействий со стороны других молекул (например, белков, взаимодействующих с нуклеиновыми кислотами).
Как показали измерения, шаг спирали B-формы ДНК превышает шаг спирали A-формы ДНК. Большая бороздка в A-форме ДНК уже, чем малая бороздка; в B-форме ДНК большая бороздка шире малой. Так как большинство белков, участвующих в специфическом узнавании последовательности ДНК, взаимодействуют с атомами остатков азотистых оснований, обращенных в большую бороздку, переход из B-формы в A-форму может служить для инактивации экспрессии определенных участков ДНК (при переходе в A-форму ширина большой бороздки уменьшается от 17 Å до 8 Å).
Табл. 1. Геометрия структур A-
и B-форм ДНК, построенных с помощью
программы fiber, и структуры реальной ДНК (см.
ниже).
| Форма ДНК |
|
|
|
| Тип спирали |
|
|
|
| Шаг спирали, Å |
(измерен между 1-м и 12-м нуклеотидами) |
(измерен между 1-м и 12-м нуклеотидами) |
|
| Число оснований на виток |
|
|
|
| Ширина большой бороздки, Å |
(измерена от 7-го нуклеотида) |
(измерена от 7-го нуклеотида) |
|
| Ширина малой бороздки, Å |
(измерена от 7-го нуклеотида) |
(измерена от 7-го нуклеотида) |
|
Для дальнейшего исследования геометрии структуры двух форм ДНК использовались программы find_pair и analize пакета 3DNA. Программа find_pair осуществляет поиск пар нуклеотидов в трехмерных структурах нуклеиновых кислот. Запуск программы для проведения поиска нуклеотидных пар в составе структур двух форм ДНК осуществлен следующими командами Linux:
find_pair -t gatcA.pdb pairsA.txt
find_pair -t gatcB.pdb pairsB.txt
Параметр -t, с которым была запущена программа, указывает на то, что описание координат атомов нуклеиновой кислоты может находиться в полях HETATM исследуемого pdb-файла*. О значении параметра -p, с которым также может быть запущена программа, см. "Структура тРНК". Помимо pairsA.txt и pairsB.txt, для каждой из форм ДНК программой find_pair были созданы следующие файлы:
bestpairs.pdb (3D-структуры всех выявленных пар нуклеотидов);
hel_regions.pdb (3D-структуры всех дуплексов нуклеиновых кислот, выявленных в pdb-файле);
col_chains.scr (скрипт RasMol, выделяющий различные цепи ДНК pdb-файла различными цветами);
col_helices.scr (скрипт RasMol, выделяющий различные дуплексы ДНК различными цветами);
bp_order.dat (содержит информацию о выявленных парах азотистых оснований);
ref_frames.dat (содержит информацию о выявленных парах азотистых оснований).
Информация, содержащаяся в выходных файлах pairsA.txt и pairsB.txt, приведена ниже (цифры соответствуют номерам строк, см. 3DNA Home Page):
1) название входного pdb-файла;
2) название выходного
файла;
3) содержит ли
структура дуплексные участки (принимает значение 2 если содержит);
4) количество
выявленных пар азотистых оснований;
5) первая единица показывает, что информация о
выявленных парах азотистых оснований будет приведена ниже (в последних версиях
программы данный параметр не может принимать других значений); вторая
единица показывает, что программа была запущена с параметром
-t;
6) - 21)
перечисление выявленных пар азотистых оснований и их основных характеристик:
номера нуклеотидов, образующих пару,
характеристика участка дуплекса, в состав которого входит пара (параметр принимает значение 0 если пара входит в состав протяженного дуплексного участка, 1 - если пара является изолированной, 9 - если дуплекс прерывается),
названия цепей,
одно- и трехбуквенные (в квадратных скобках) обозначения азотистых оснований, образующих пару,
основные геометрические характеристики пары.
22) критерии отбора
пар азотистых оснований;
23) количество
неканонических пар, выявленных программой; количество выявленных дуплексов;
24) выявленные
дуплексы с указанными номерами входящих в них нуклеотидов.
Анализ структур ДНК был проведен с помощью программы analyze. Команды Linux, с помощью которых была запущена программа, приведены ниже:
analyze
pairsA.txt
analyze pairsB.txt
Выходные файлы с расширениями out (gactA.out и gatcB.out) содержат следующую информацию.
Перечисление нуклеотидных пар анализируемых структур (указаны неканонические пары, изолированные пары и пары, на которых прерывается дуплекс; в данных структурах такие пары отсутствуют). В скобках приведены значения среднеквадратических отклонений (RMS deviation), характеризующих отклонение "экспериментальных" структур остатков азотистых оснований, взятых из pdb-файла, от идеальных структур, взятых из базы данных NDB (Nucleic Acid Database). Среднеквадратические отклонения вычисляются на основе матриц координат атомов остатков азотистых оснований (см. 3DNA Home Page).
Перечисление возможных водородных связей, образующихся между остатками азотистых оснований каждой нуклеотидной пары.
Площади областей перекрывания пар азотистых оснований.
Координаты пар азотистых оснований.
Значения, характеризующие различные типы отклонений азотистых оснований, образующих пару, от идеального расположения (shear, stretch, stagger, buckle, propeller, opening).
Значения, характеризующие различные типы отклонений соседних пар азотистых оснований от идеального расположения (shift, slide, rise, tilt, roll, twist).
Значения, характеризующие различные типы отклонений пар азотистых оснований от идеального расположения относительно оси дуплекса (x-disp, y-disp, h-rise, incl., tip, h-twist).
Является ли структура правой или левой спиралью.
Углы между гликозидными связями и осью, соединяющей атомы углерода 1' каждой нуклеотидной пары; расстояния между атомами 1' каждой нуклеотидной пары; расстояния между атомами пуринов N9 и C8 и пиримидинов C8 и C6 соседних нуклеотидов. Данные параметры могут быть использованы для выявления неканонических пар азотистых оснований.
Форма ДНК, к которой относятся каждые две соседние пары нуклеотидов. Форма ДНК определяется на основе торсионных углов структуры и различных типов отклонений остатков азотистых оснований от идеального расположения.
Ширина большой и малой бороздок структуры.
Параметры векторов, образующих ось дуплекса (вектора определяются положениями 1' атомов углерода) и код, который может быть добавлен в pdb-файл для выявления главной оси структуры. Главная ось B-формы ДНК, выявленная таким способом, показана на рисунке.
Значения торсионных углов α, β, γ, δ, ε, ζ и χ.
Значения торсионных углов, определяющих конформацию остатка дезоксирибозы; конформация остатка дезоксирибозы.
Расстояния между атомами углерода 1' и фосфора соседних нуклеотидов. Данные значения также могут быть использованы для определения формы ДНК (например, в A-форме ДНК расстояния между соседними 1' атомами составляют 5.4 Å, в B-форме - 4.9 Å).
Смещение атомов фосфора, кислорода 4' и углерода 1' каждых двух соседних нуклеотидных пар относительно центральной оси.
Координаты центра каждых двух соседних нуклеотидных пар; координаты вектора центральной оси каждых двух соседних нуклеотидных пар.
Значения торсионных углов в структурах A- и B-форм ДНК практически не зависят от номера нуклеотида (различия не превышают 0.1°) и приведены в табл. 2. Наибольшие различия между A- и B-формами ДНК наблюдается по углам δ, ζ и χ.
* Для получения информации о значении параметров потоки stdout и stderr программ find_pair и analyze при запуске с параметром -h были перенаправлены в текстовые файлы.
Табл. 2.
Торсионные углы
α, β, γ, δ, ε,
ζ и χ
в структурах A- и B-форм ДНК (углы, значения
которых для A- и B-форм
различаются более чем на 40°,
выделены красным),
а также в структуре реальной ДНК (см. ниже).
|
Форма ДНК |
|
|
Различия между A- и B-формами |
|
|
Торсионные углы, градусы |
||||
| α |
|
|
21.8 | -52.9 |
| β |
|
|
38.5 | 164.7 |
| γ |
|
|
10.6 | 37.3 |
| δ |
|
|
64.2 | 140.2 |
| ε |
|
|
7.0 | -132.2 |
| ζ |
|
|
85.4 | -143.7 |
| χ |
|
|
59.2 | -101.5 |
Рассмотрена структура реальной ДНК, представленная в pdb-файле dna22. Геометрические параметры структуры, измеренные с помощью RasMol, приведены в табл. 1. Так как значения шага спирали и ширины бороздок различаются при проведении измерения от разных нуклеотидов, в таблице указаны средние значения этих параметров. Значения параметров, измеренные от каждого из нуклеотидов, приведены здесь. Большая бороздка структуры была идентифицирована как бороздка, в которую направлены аминогруппы остатков аденина.
Структура представляет собой правую спираль, как и рассмотренные выше формы ДНК. На один виток спирали приходится 10 нуклеотидов, шаг спирали приблизительно составляет 34 Å (данные значения соответствуют параметрам B-формы ДНК). Средняя ширина большой бороздки структуры приблизительно равна ширине большой бороздки B-формы ДНК и значительно превышает среднюю ширину малой бороздки, которая также близка к ширине малой бороздки B-формы. Таким образом, структура, представленная в файле dna22, представляет собой B-форму ДНК.
Дальнейшее исследование структуры было проведено с помощью программ find_pair и analyze. Команда Linux, с помощью которой были запущены программы, приведена ниже:
find_pair -t dna22.pdb stdout | analyze
Рассмотрен выходной файл программы analyze dna22.out. В составе структуры выявлены две неканонические пары азотистых оснований, состоящие из двух остатков гуанина. В каждой из пар образуются две водородные связи между атомами N2 и N7 и между атомами O6 и O6. Так как атомы O6 являются акцепторами водорода, один из гуанинов каждой пары, возможно, переходит из кето-формы в енольную форму, при этом его атом O6 становится донором водорода*. Предположительная структура водородных связей между остатками азотистых оснований таких пар приведена на рис. 3. Следует отметить, что диаметр пары GG, измеренный между 1' атомами дезоксирибозы (10.73 Å), соответствует диаметру канонических пар (10.68 для B-формы ДНК). Таким образом, данная пара не приводит к значительным изменениям диаметра дуплекса.
В связи с тем, что структура ДНК содержит неканонические пары нуклеотидов, возможно, она была взята из pdb-файла, содержащего комплекс какого-либо белка мисматч-репарации и ДНК, содержащей мисматч, или из структуры вирусной частицы.
Средние значения торсионных углов α, β, γ, δ, ε, ζ и χ в рассмотренной структуре ДНК приведены в табл. 2 (вычисления проводились с помощью электронной таблицы Excel). Значения углов δ, ζ и χ, по которым наблюдаются наибольшие различия A- и B-форм ДНК, близки к значением соответствующих углов B-формы.
Принадлежность к A- или B-форме ДНК может быть также определена по конформации остатков дезоксирибозы. По результатам analyze, 11 из 24 остатков дезоксирибозы структуры находится в конформации C2'-эндо (как и в идеальной B-форме ДНК). Остальные остатки находятся в C1'- и C3'-экзо конформациях, но не в C3'-эндо конформации, характерной для A-формы ДНК. Это подтверждает принадлежность структуры к B-форме ДНК, однако, данный признак не является достаточно надежным (менее половины остатков дезоксирибозы находятся в "правильной" конформации).
Программа analyze способна определять форму исследуемой нуклеиновой кислоты по значениям торсионных углов и различных типов отклонений остатков азотистых оснований от идеального расположения (см. выше). По результатам программы, большая часть нуклеотидных пар также принадлежит к B-форме, а два коротких участка не могут быть отнесены ни к одной форме ДНК. Оба этих участка содержат неканонические пары GG. Возможно, такие пары оказывают влияние на дуплекс, вызывая отклонения от идеальной структуры.
С помощью программы pdb2img были построены стопочные модели рассматриваемой структуры (рис. 4). Полученные изображения также подтверждают принадлежность структуры к B-форме ДНК:
плоскости остатков азотистых оснований практически перпендикулярны оси молекулы,
в центре одной из проекций (вид с торца) отсутствует отверстие.
* Вероятно, структура была получена методом РСА и, следовательно, не содержит координат атомов водорода.
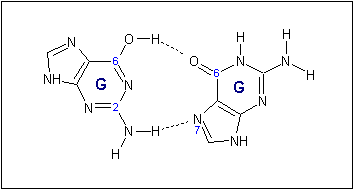
Рис. 3. Предположительная структура неканонической пары азотистых оснований, состоящей из двух гуанинов.
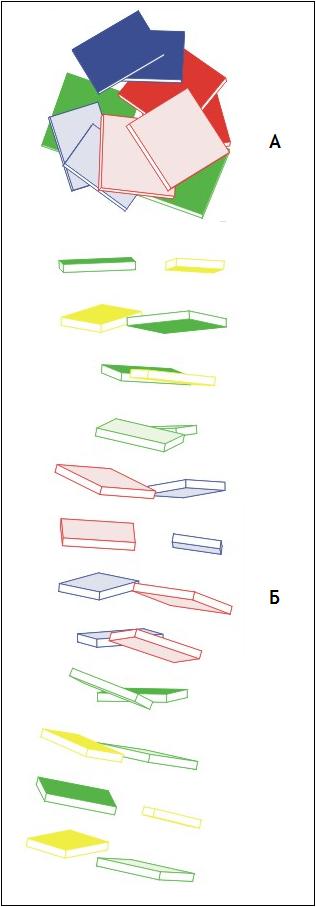
Рис. 4. Стопочные модели реальной структуры ДНК (А - вид с торца, Б - вид сбоку). Различные нуклеотиды показаны цветами. Изображения получены с помощью программы pdb2img.
1. Berg, Tymoczko, Stryer, Clarke Biochemistry, 5th edition. New York: W. H. Freeman and Co.; 2002.
© Куравский Михаил Львович, 2006