Реконструкция эволюционной доменной архитектуры.
1. Выбор домена и его описание.
Для работы был взят домен В3 (ID: B3, AC: PF02362), из семейства высоконсервативных ДНК-связывающих доменов В3, присутствующих исключительно в ТФ высших растений и не только. Состоит обычно из 100-120 а.о., представленных в виде семи β-листов и двух α‑ спиралей; сам домен имеет форму бочонка. Он найден у 65 видов в составе 7718 последовательностей. Для домена В3 известно 103 архитектуры. Считают, что гены транскрипционных факторов, содержащих В3 домен специфичны для фотосинтезирующих эукариот, так как они не были найдены у дрожжей Saccharomyces cerevisiae, нематоды Caenorhabditis elegans, насекомыхDrosophila melanogaster, рыб Danio rerio и у млекопитающих Mus musculus и Homo sapiens.
Ссылка на список доменных архитектур белков в Pfam.
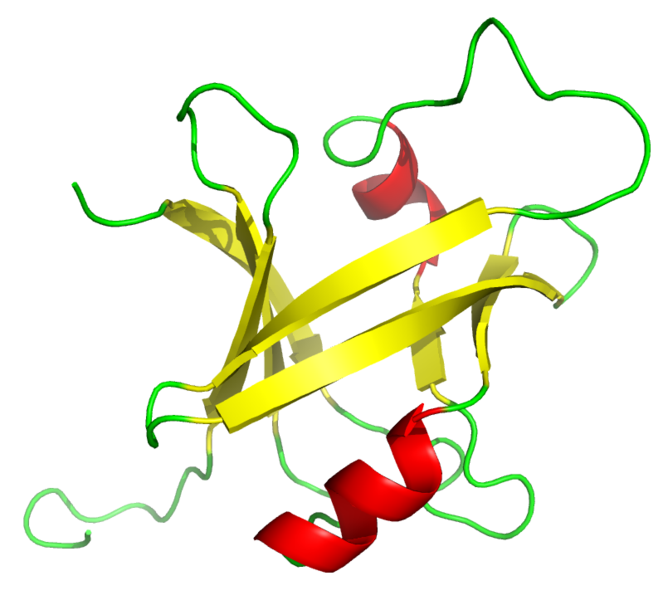
Рис.1. Проволочно-ленточная модель домена В3.
Построение выравнивания.
Для домена В3 было скачано выравнивание (Jalview: File → Fetch Sequences → Pfam (Full) → PF02362), которое было окрашено ClustalX (порог консервативности 30).
Полученное выравнивание в формате проекта.2. Выбор двух архитектур, включающих домен В3.
Среди 103 доменных архитектур были выбраны следующие:
Название архитектуры
B3, Auxin_resp, AUX_IAA x 2
AP2, B3
Схема
Число последовательностей
148
224
Характеристика соседствующих доменов
Auxin_resp (PF06507) – входит в состав белков-факторов ответа на ауксина (растительный гормон)
AUX_IAA (PF02309) – гены, кодирующие белки содержащие домен из этого семейства, активно транскрибируются в ответ на воздействие ауксинаAP2 (PF00847) – присутствует в составе ТФ из семейства AP2/EREBP. До недавнего времени считалось, что белки, содержащие данный домен характерны лишь для растений, однако, как показали последние исследования, данный домен был обнаружен у Апикомплексов.
Файл swisspfam с архитектурами последовательностей Uniprot доступен на сервере kodomo (/srv/databases/pfam/swisspfam.gz). Он был обработан скриптом swisspfam_to_xls.py:
python swisspfam_to_xls.py -z /srv/databases/pfam/swisspfam.gz -p PF02362 -o PF02362.xlsИз полученной таблицы сделали сводную, где строки – это AC последовательностей, а столбцы – домены Pfam. После, на сайте Uniprot → Retrieve, ввели список искомых АС и получили текстовый файл с полными записями интересующих нас последовательностей. С помощью скрипта командой получили таксономию скачанных последовательностей
python uniprot-to-taxonomy.py -i LIST.txt -o tax.xlsТаблица с указанием последовательности, доменной архитектуры, таксономии и длины CHASE-домена (лист TABLE).
На основании анализа полученных данных в качестве таксона был выбран отдел Цветковые растения (Magnoliophyta), а в качестве подтаксонов класс Однодольные (Liliopsida) и класс Двудольные (Еudicotyledons). Индексы: Е1 и Е2 – Еudicotyledons с архитектурой 1 и 2 соответственно; L1 и L2 – Liliopsida с архитектурами 1 и 2 соответственно.
Далее необходимо было выбрать последовательности так, чтобы в каждом подтаксоне от каждой архитектуры было по 20 последовательностей. Отобранные последовательности представлены на листе sum в таблице.
Всего последовательностей с первой архитектурой - 146, со второй – 221.
Файл с добавленными идентификаторами ссылка.
Чтобы в выравнивании оставить только отобранные последовательности я воспользоваласть скриптом filter-alignment.pypython filter-alignment.py –I B3align.mfa –m ac.txt –o align_selected.fa –a “_”
Опция -i нужна для указания входного файла с выравниванием в fasta формате, опция -m нужна, чтобы задать файл с идентификаторами или AC последовательностей (должны совпадать со входным файлом), опция -o задает файл вывода, и опция -a позволяет задать символ разделителя. Было отобрано лишь 353 последовательности из 367.Полученный файл был открыт в программе Jalview, где был дополнительно почищен. В целом можно сказать, что в обеих группах можно выделить более-менее консервативные участки (блоки). Стоит отметить, что в последовательностях L2 консервативных участков мало и они сильно «выбиваются из колеи» при выравнивании, поэтому многие из них были удалены. Ссылка на проект.
2. Построение филогенетического дерева домена В3.
Для построения филогенетического дерева домена В3, были отобраны по 20 последовательностей, входящих в первую и вторую архитектуру у классов Однодольные и Двудольные. Обозначения: L1 – Однодольные с первой архитектурой; L2 – Однодольные со второй архитектурой; E1 – Двудольные с первой архитектурой; Е2 – Двудольные со второй архитектурой.
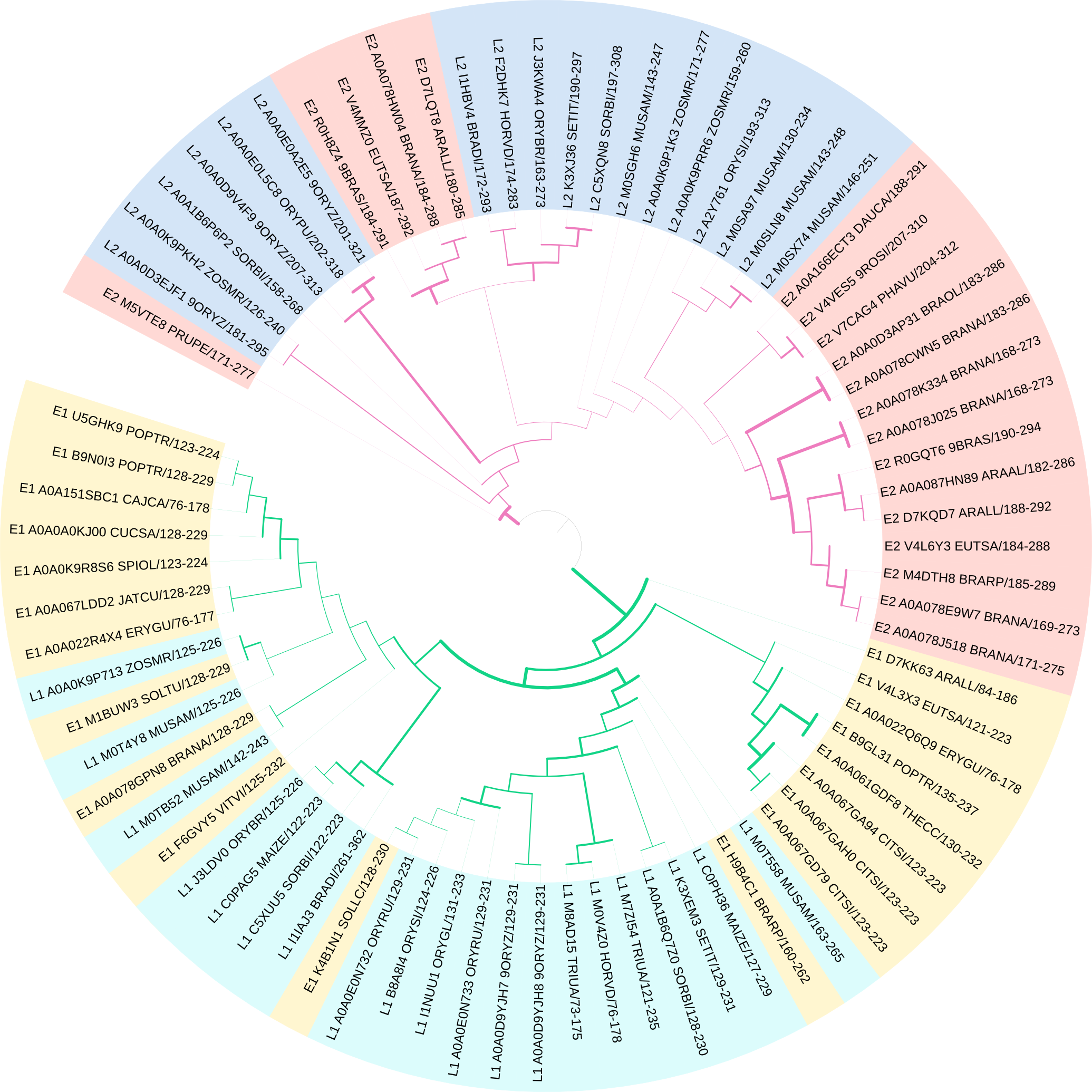
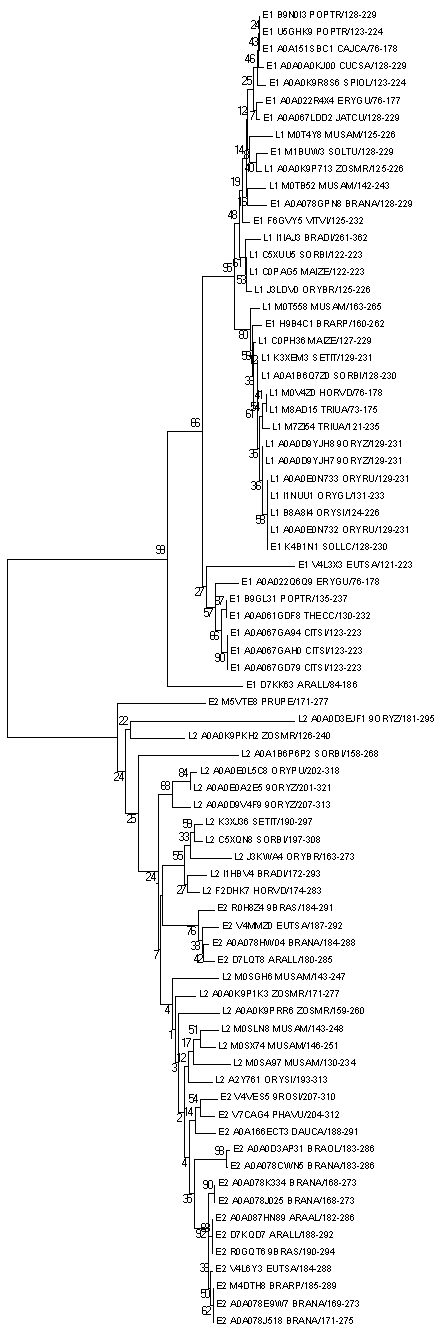
Дерево было построено в программе MEGA методом минимальной эволюции с бутстреп-поддержкой 100. Изображение дерева было отредактировано в программе iTOL , где в качестве настроек было выбрано следующее: толщина ветвей отражает величину бутстрепа; длина ветветвей проигнорирована; розовым выделены последовательности класса Двудольные, голубым – Однодольные.
Рис.4. Филогенетическое дерево домена В3, отредактированное в программе iTOL.
Рис.5. Филогенетическое дерево домена В3.
Как видно из построенного дерева, наблюдается четкое эволюционное разделение первой и второй архитектур, чего не скажешь о разделении по классам. Можно отметить, что у клады, куда вошли все последовательности архитектуры 1 класса Однодольные (и несколько последовательностей из класса Двудольные), наблюдается очень хорошая бутстреп поддержка 95.Ссылка на скобочную формулу дерева.