С помощью сайта IUPAC найдено значение кода фермента: 1.3.3.1.
- EC 1.Фермент oксидоредуктаза. Оксидоредуктазы - класс ферментов, катализирующих окислительно-восстановительные реакции. Оксидоредуктазы играют важную роль в обеспечении клеток энергией.
- EC 1.3 Действует на СН-СН связи, которые являются донорами электрона.
- EC 1.3.3 Кислород выступает в роли акцептора электрона.
- EC 1.3.3.1 Дегидрогеназа(оксидаза) дигидропиримидинкарбоксиловой(родицоновой) кислоты
(дигидрооротата).
Реакция: (S)-dihydroorotate + O2 = orotate + H2O2.
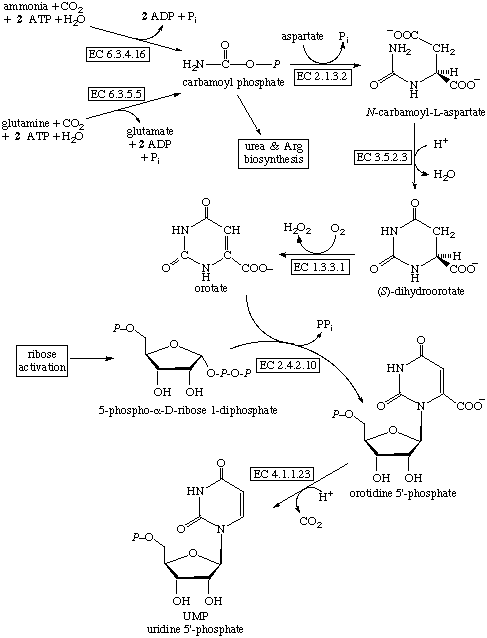
- Название по систематике: (S)-дигидрооротат:кислород оксидоредуктаза.
C помощью SRS найдены в UniProt ферменты из 3-х хорошо изученных модельных организмов - кишечной палочки Escherichia coli K-12, археи Methanococcus jannaschii и человека.
Запрос выглядел так:
(((([uniprot-ID:*_Ecoli*] | [uniprot-ID:*_Human*]) | [uniprot-ID:*_Metja*]) & [uniprot-ECNumber:1.3.3.1*]) & [uniprot-GeneName:*])
По полученным данным в протоколе создана
таблица такого вида:
Сравнение доменной структуры ферментов из далеких организмов
UniProt ID |
UniProt AC |
Имя гена |
Домен |
||
Идентификатор Pfam |
Положение в последовательности |
||||
| 1 | PYRD_ECOLI | P0A7E1 | PYRD | PF01180 | 47-336 |
| 2 | PYRD_HUMAN | Q02127 | DHODH | PF01180 | 77-377 |
| 3 | PYRD_METJA | Q58070 | PYRD | PF01180 | 5-290 |
С использованием встроенных в SRS программ(а именно было использовано NeedleP), были получены попарные выравнивания:
Выравнивание PYRD_ECOLI и PYRD_HUMAN