Восстановление предкового состояния доменной архитектуры
Цель проекта: реконструировать эволюцию доменной архитектуры выбранного домена.
1. Выбор объектов и построение выравнивания
Для выполнения задания был выбран домен PF07738 - SUN domain (рисунок 1). Для этого домена в Pfam находится 2000 последовательности для 591 вида.SUN-домен обычно расположен после трансмембранного домена. Большинство белков, содержащих такой домен, вероятно, участвуют в позиционировании ядра внутри клетки. SUN-белки, предположительно, расположены во внутренней мембране ядерной оболочки.
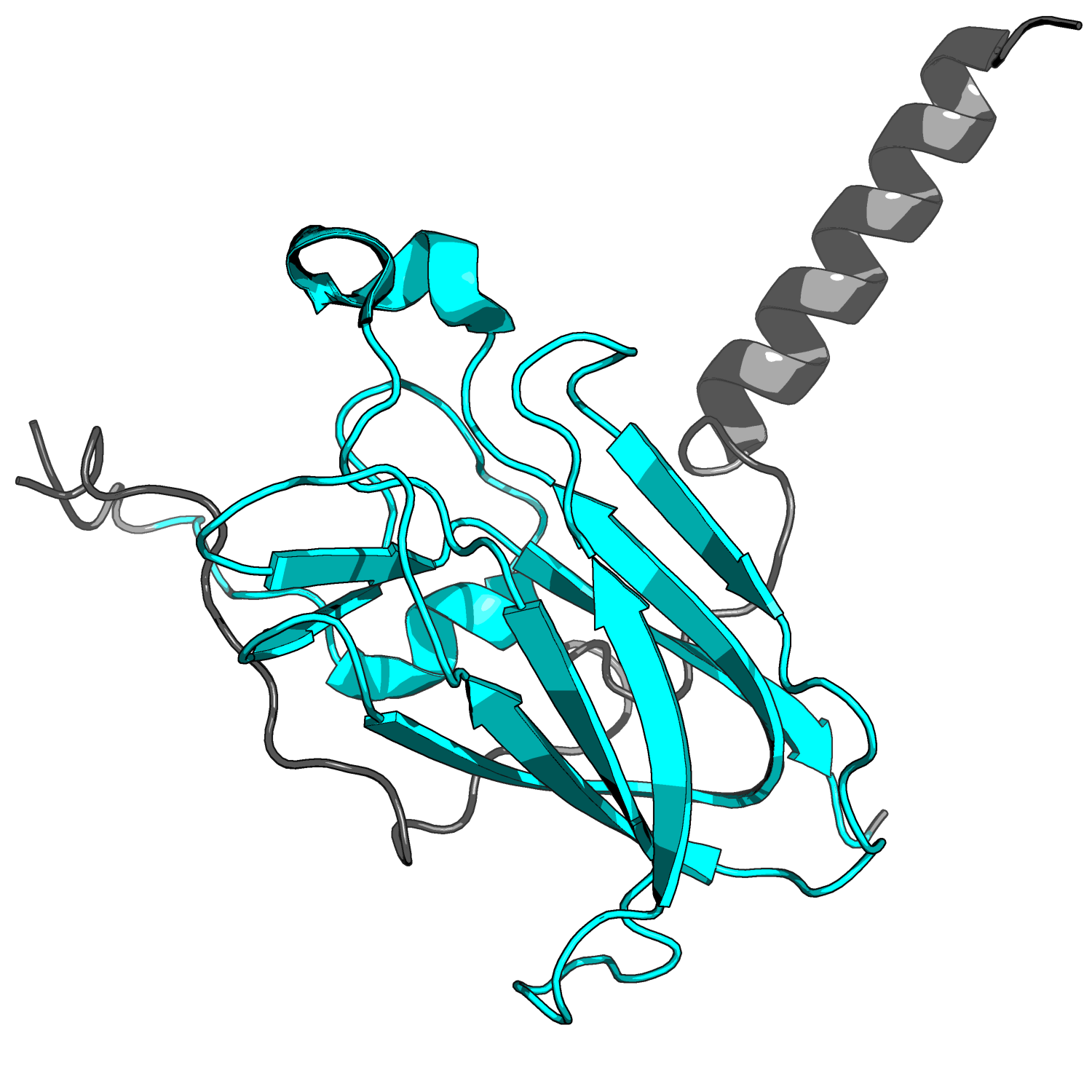
Рисунок 1. SUN-домен в составе структуры 3UNP. Домен покрашен голубым цветом.
Далее была получена таблица, содержащая информацию о доменной структуре всех последовательностей, содержащих исследуемый домен. Это было сделано с помощью скрипта swisspfam-to-xls.py (в файле domain_AC.txt находится идентификатор выбранного домена):
python swisspfam-to-xls.py -z /srv/databases/pfam/swisspfam.gz -m domain_AC.txt -o PF07738.txtДалее для каждой последовательности была получена информация о таксономической принадлежности. Для из файла PF07738.txt был получен список идентификаторов последовательностей, которые были скачаны из Uniprot. Файл с таксономией был получен с помощью скрипта uniprot_to_taxonomy.py:
python uniprot-to-taxonomy.py -i PF07738_uniprot.txt -o PF12229_taxonomy.txtВ результате была получена сводная таблица.
Выбранный домен входит в 58 доменных архитектур. Из списка доменных архитектур для дальнейшего рассмотрения были выбраны 1 и 3 (рисунок 2). Первая архитектура - однодоменная (обозначение - 1), а вторая содержит второй домен MRP - Mitochondrial RNA binding protein, который связывается с РНК и стабилизирует ее в развернутом состоянии, обеспечивая РНК-РНК гибридизацию (обозначение архитектуры - 2).
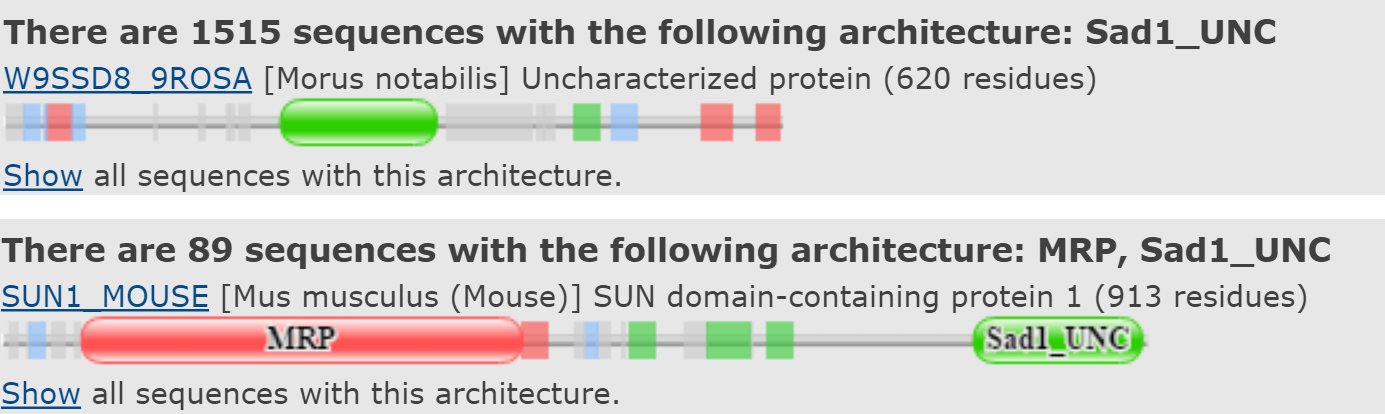
Рисунок 2. Выбранные доменные архитектуры.
Среди всех последовательностей, содержащих исследуемый домен, было отобрано по 10, принадлежащих каждому подтаксону и каждой архитектуре, а также последовательность с известной 3D-структурой (всего 41 последовательность). Выбранные последовательности были вырезаны из общего выравнивания и переименованы в соответствии с принадлежностью к подтаксону и архитектуре. Полученное выравнивание было открыто в программе Jalview, были удалены пустые столбцы и небольшие фрагменты на C- и N-концах. Все последовательности были разделены на две группы в соответствии с принадлежностью к одной из двух архитектур. Каждая группа была раскрашена ClustalX By conservation с порогом в 17%. Последовательности из группы двухдоменной архитектуры выровнялись очень хорошо (рисунок 3). Последовательности из группы однодоменной архитектуры оказались менее консервативные, кроме того среди них присутствовало две последовательности с недостающими C-концами, они были удалены. На рисунке 3 представлено изображение .jvp-проекта для итогового выравнивания.

Рисунок 3. Проект .jvp для итогового выравнивания. Последовательности раскрашены по группам в соответствии с принадлежностью к различным архитектурам. Для обеих групп использована раскраска ClustalX By conservation с порогом в 17%.
2. Построение филогенетического дерева домена
Для полученного итогового выравнивания с помощью программы MEGA было построено филогенетическое дерево домена методом Neighbor-joining (рисунок 4).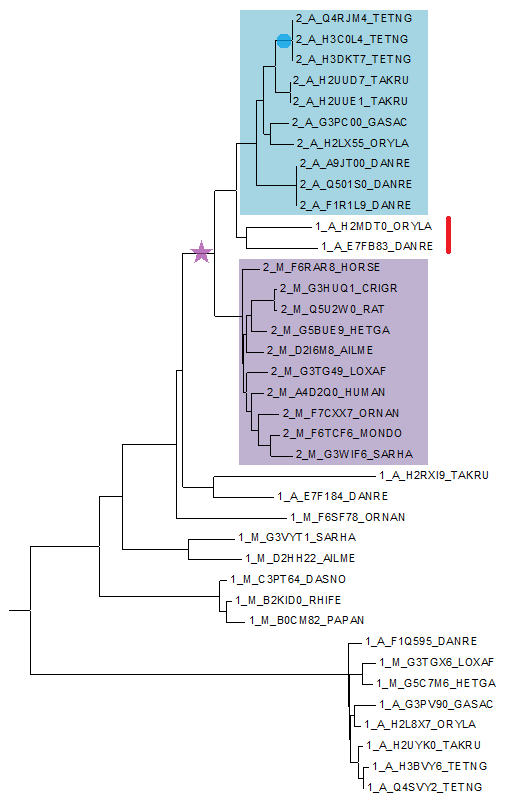
Рисунок 4. Изображение филогенетического дерева домена, полученное с помощью программы MEGA методом Neighbor-joining.
((((((((((((2_A_Q4RJM4_TETNG,2_A_H3C0L4_TETNG),2_A_H3DKT7_TETNG),(2_A_H2UUD7_TAKRU,2_A_H2UUE1_TAKRU)),(2_A_G3PC00_GASAC,2_A_H2LX55_ORYLA)),(2_A_A9JT00_DANRE,(2_A_Q501S0_DANRE,2_A_F1R1L9_DANRE))),(1_A_H2MDT0_ORYLA,1_A_E7FB83_DANRE)),(2_M_F6RAR8_HORSE,((((2_M_G3HUQ1_CRIGR,2_M_Q5U2W0_RAT),2_M_G5BUE9_HETGA),2_M_D2I6M8_AILME),(2_M_G3TG49_LOXAF,(2_M_A4D2Q0_HUMAN,(2_M_F7CXX7_ORNAN,(2_M_F6TCF6_MONDO,2_M_G3WIF6_SARHA))))))),(1_A_H2RXI9_TAKRU,1_A_E7F184_DANRE)),1_M_F6SF78_ORNAN),(1_M_G3VYT1_SARHA,1_M_D2HH22_AILME)),(1_M_C3PT64_DASNO,(1_M_B2KID0_RHIFE,1_M_B0CM82_PAPAN))),(1_A_F1Q595_DANRE,((1_M_G3TGX6_LOXAF,1_M_G5C7M6_HETGA),((1_A_G3PV90_GASAC,1_A_H2L8X7_ORYLA),(1_A_H2UYK0_TAKRU,(1_A_H3BVY6_TETNG,1_A_Q4SVY2_TETNG))))));
На рисунке дерева видно, что двухдоменные архитектуры четко разбились на две клады в соответствии с принадлежностью к подтаксонам (клада лучеперых рыб выделена голубым цветом, клада млекопитающих - фиолетовым). В тоже время внутри группы однодоменных архитектур такой четкой закономерности не наблюдается. Легко заметить, что среди двухдоменных архитектур "затерялись" две последовательности из группы однодоменных архитектур (отмечены красной скобкой). Скорее всего это связано с ошибкой аннотации и упущением домена MRP в данных белках.
На построенном дереве показаны примеры возникновения ортологов (разделение путей эволюции белков в результате видообразования) - место видообразования отмечено звездочкой, и парологов (дупликация гена с последующей эволюцией) - место дупликации отмечено кружком.
3. Профиль
В качестве подсемейства последовательностей для построения профиля были выбраны последовательности 2_M_* (на рисунке 4 они показаны фиолетовым цветом), так как они выделяются в отдельную кладу.Для выравнивания выбранных последовательностей программой hmm2build был построен профиль последовательностей, который затем был откалиброван программой hmm2calibrate. Далее с использованием полученного профиля программой hmm2search был проведен поиск по всем белкам, содержащим домен PF07738:
hmm2build result PF07738_2_M.fasta hmm2calibrate result hmm2search result uniprot-PF07738.fasta > finДалее на основе полученных результатов с помощью средств Excel была построена ROC-кривая (рисунок 5). Расчет для построение кривой приведен в файле на листе "roc".
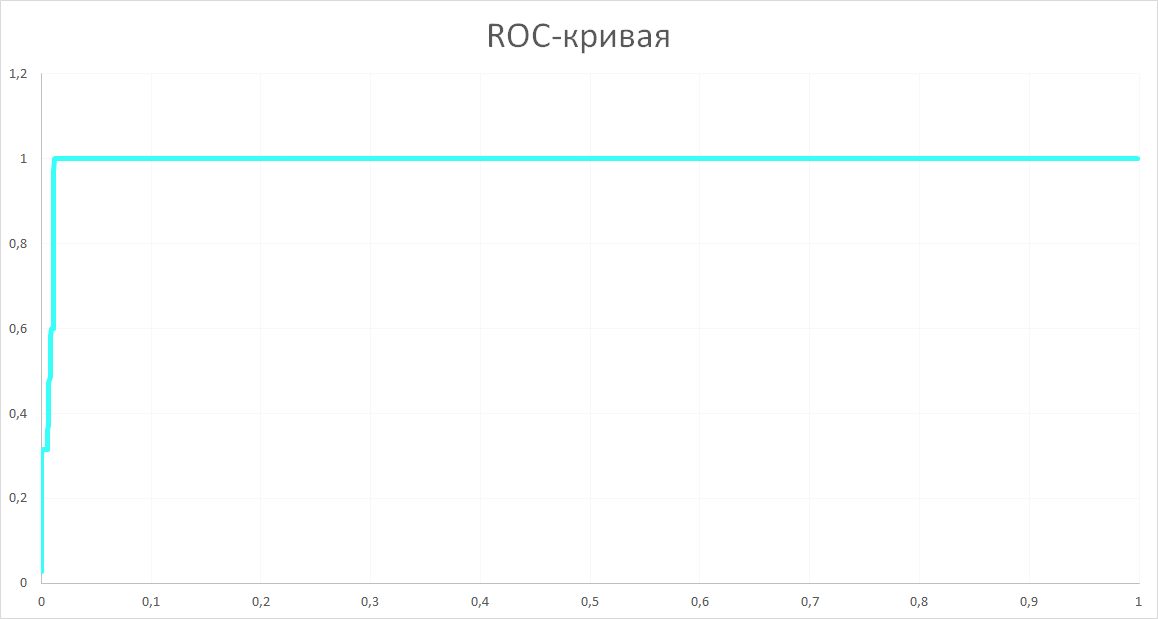
Рисунок 5. ROC-кривая, по оси X отложена специфичность, по оси Y - чувствительность.
| На самом деле | принадлежит подсемейству | не принадлежит | сумма |
| Выше порога по профилю | 34 | 8 | 42 |
| Ниже порога по профилю | 2 | 736 | 738 |
| сумма | 36 | 744 | 780 |
© Наталья Ланина
e-mail: n.lanina@fbb.msu.ru
последний раз обновлялось: 04.12.15