Для того, чтобы собрать из всех чтений моего набора (прошедших очистку) контиги без использования референса, я использовала пакет velvet. Этот пакет программ производит сборку геномов на основе алгоритма, использующего граф Де Брейна. Пакет состоит из друх программ. Первая - velveth - создаёт на основе чтений chr6_out.fastq банк из заданного количества k-меров (последовательностей из k нуклеотидов). Вторая - velvetg - производит сборку контигов на основе графа этих k-меров. Я выбрала длину k-мера равной 31. Команды: velveth velvetDir 31 -fastq chr6_out.fastq; velvetg velvetDir -cov_cutoff auto. После окончания работы программы velvetg на экран выдаётся следующия информация о полученной сборке, представленная на рис.1. N50: 661; размер самого длинного контига получившейся сборки: 27402. Файл с последовательностями контигов: contigs.fa.
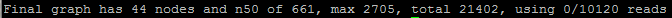
Рис.1 Выдача программы velvetg
С помощью программы blastn (алгоритма megablast) я сравнила получившиеся контиги с последовательностью хромосомы. Я создала базу blast из последовательности хромосомы командой: makeblastdb -in chr6.fasta -dbtype nucl. Затем я запустила blastn командой: blastn -query contigs.fa -db chr6.fasta -out blastn.out -outfmt 6. Полученный файл я обработала с помощью Excel. Общее количеоство контигов: 41. Количество контингов, которые единственным образом картируются на хромосому: 36. Список таких контигов в порядке возрастания меньшей координаты по хромосоме представлен в таблице. Велечина разрывов между ними представлена в столбце gap. Перекрытий найдено не было. В таблице представлены контиги, которые отображаются не единственным образом.
.jpg)